-

Sekwencjonowanie metagenomiczne -NGS
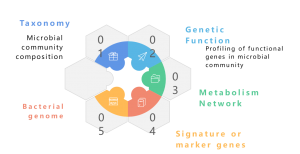
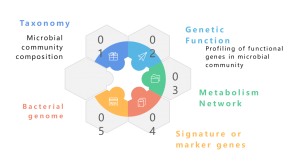
Metagenom to zbiór całkowitego materiału genetycznego mieszanej społeczności organizmów, na przykład metagenomów środowiskowych i ludzkich. Zawiera genomy zarówno mikroorganizmów, które można hodować, jak i tych, których nie można hodować. Sekwencjonowanie metagenomiczne metodą shotgun za pomocą NGS umożliwia badanie tych skomplikowanych krajobrazów genomowych osadzonych w próbkach środowiskowych, zapewniając coś więcej niż tylko profilowanie taksonomiczne, dając również szczegółowy wgląd w różnorodność gatunków, dynamikę liczebności i złożone struktury populacji. Oprócz badań taksonomicznych metagenomika typu shotgun oferuje również perspektywę genomiki funkcjonalnej, umożliwiając badanie zakodowanych genów i ich domniemanej roli w procesach ekologicznych. Wreszcie ustanowienie sieci korelacji między elementami genetycznymi a czynnikami środowiskowymi przyczynia się do całościowego zrozumienia skomplikowanych wzajemnych zależności między zbiorowiskami drobnoustrojów a ich pochodzeniem ekologicznym. Podsumowując, sekwencjonowanie metagenomiczne stanowi kluczowy instrument umożliwiający rozwikłanie zawiłości genomicznych różnorodnych społeczności drobnoustrojów, rzucając światło na wieloaspektowe relacje między genetyką i ekologią w tych złożonych ekosystemach.
Platformy: Illumina NovaSeq i DNBSEQ-T7
-

Sekwencjonowanie metagenomiczne – TGS
Metagenom to zbiór materiału genetycznego mieszanej społeczności organizmów, takich jak metagenomy środowiskowe i ludzkie. Zawiera genomy zarówno mikroorganizmów, które można hodować, jak i tych, których nie można hodować. Sekwencjonowanie metagenomiczne umożliwia badanie tych skomplikowanych krajobrazów genomicznych osadzonych w próbkach ekologicznych, zapewniając coś więcej niż tylko profilowanie taksonomiczne. Oferuje także perspektywę genomiki funkcjonalnej poprzez badanie zakodowanych genów i ich domniemanej roli w procesach środowiskowych. Podczas gdy tradycyjne podejście typu shotgun z sekwencjonowaniem Illumina było szeroko stosowane w badaniach metagenomicznych, pojawienie się sekwencjonowania o długim odczycie Nanopore i PacBio zmieniło tę dziedzinę. Technologie Nanopore i PacBio usprawniają dalsze analizy bioinformatyczne, zwłaszcza składanie metagenomu, zapewniając bardziej ciągłe składanie. Raporty wskazują, że metagenomika oparta na Nanopore i PacBio z powodzeniem wygenerowała kompletne i zamknięte genomy bakteryjne ze złożonych mikrobiomów (Moss, EL i in., Nature Biotech, 2020). Integracja odczytów Nanopore z odczytami Illumina zapewnia strategiczne podejście do korekcji błędów, łagodząc nieodłączną niską dokładność Nanopore. To synergiczne połączenie wykorzystuje mocne strony każdej platformy sekwencjonowania, oferując solidne rozwiązanie umożliwiające pokonanie potencjalnych ograniczeń oraz zwiększenie precyzji i wiarygodności analiz metagenomicznych.
Platforma: Nanopore PromethION 48, Illumia i PacBio Revio
-

Sekwencjonowanie wodorosiarczynem całego genomu (WGBS)
Sekwencjonowanie wodorosiarczynem całego genomu (WGBS) to złoty standard metodologii dogłębnego badania metylacji DNA, w szczególności piątej pozycji w cytozynie (5-mC), kluczowym regulatorze ekspresji genów i aktywności komórkowej. Zasada leżąca u podstaw WGBS obejmuje obróbkę wodorosiarczynem, indukującą konwersję niemetylowanych cytozyn do uracylu (C do U), pozostawiając niezmienione metylowane cytozyny. Technika ta zapewnia rozdzielczość jednozasadową, umożliwiając naukowcom kompleksowe badanie metylomu i odkrywanie nieprawidłowych wzorców metylacji związanych z różnymi schorzeniami, zwłaszcza rakiem. Stosując WGBS, naukowcy mogą uzyskać niezrównany wgląd w krajobrazy metylacji całego genomu, zapewniając szczegółowe zrozumienie mechanizmów epigenetycznych leżących u podstaw różnorodnych procesów biologicznych i chorób.
-

Test na chromatynę dostępną dla transpozazy z sekwencjonowaniem o dużej przepustowości (ATAC-seq)
ATAC-seq to wysokoprzepustowa technika sekwencjonowania stosowana do analizy dostępności chromatyny w całym genomie. Jej zastosowanie zapewnia głębsze zrozumienie złożonych mechanizmów globalnej kontroli epigenetycznej nad ekspresją genów. Metoda wykorzystuje hiperaktywną transpozazę Tn5 do jednoczesnej fragmentacji i znakowania otwartych regionów chromatyny poprzez wstawienie adapterów sekwencjonowania. W wyniku późniejszej amplifikacji PCR powstaje biblioteka sekwencjonowania, która pozwala na kompleksową identyfikację otwartych regionów chromatyny w określonych warunkach czasoprzestrzennych. ATAC-seq zapewnia całościowy obraz dostępnych krajobrazów chromatyny, w przeciwieństwie do metod, które skupiają się wyłącznie na miejscach wiązania czynników transkrypcyjnych lub określonych regionach zmodyfikowanych histonami. Sekwencjonując te otwarte regiony chromatyny, ATAC-seq ujawnia regiony, w których istnieje większe prawdopodobieństwo aktywnych sekwencji regulatorowych i potencjalnych miejsc wiązania czynników transkrypcyjnych, dostarczając cennych informacji na temat dynamicznej modulacji ekspresji genów w genomie.
-

Sekwencjonowanie amplikonu 16S/18S/ITS – PacBio
Geny 16S i 18S rRNA wraz z regionem Internal Transcribed Spacer (ITS) służą jako kluczowe markery molekularnego odcisku palca ze względu na kombinację wysoce konserwatywnych i hiperzmiennych regionów, co czyni je nieocenionymi narzędziami do charakteryzowania organizmów prokariotycznych i eukariotycznych. Amplifikacja i sekwencjonowanie tych regionów zapewnia podejście niewymagające izolacji do badania składu i różnorodności drobnoustrojów w różnych ekosystemach. Chociaż sekwencjonowanie Illumina zazwyczaj celuje w krótkie regiony hiperzmienne, takie jak V3-V4 z 16S i ITS1, wykazano, że lepszą adnotację taksonomiczną można osiągnąć poprzez sekwencjonowanie pełnej długości 16S, 18S i ITS. To kompleksowe podejście skutkuje wyższym odsetkiem dokładnie sklasyfikowanych sekwencji, osiągając poziom rozdzielczości, który rozciąga się na identyfikację gatunku. Platforma sekwencjonowania pojedynczych cząsteczek w czasie rzeczywistym (SMRT) firmy PacBio wyróżnia się zapewnianiem bardzo dokładnych długich odczytów (HiFi), które obejmują amplikony pełnej długości, dorównując precyzją sekwencjonowania Illumina. Możliwość ta pozwala naukowcom osiągnąć niezrównaną przewagę — panoramiczny widok na krajobraz genetyczny. Rozszerzony zasięg znacznie zwiększa rozdzielczość adnotacji gatunków, szczególnie w obrębie zbiorowisk bakterii lub grzybów, umożliwiając głębsze zrozumienie zawiłości populacji drobnoustrojów.
-

Sekwencjonowanie amplikonu 16S/18S/ITS-NGS
Sekwencjonowanie amplikonu przy użyciu technologii Illumina, w szczególności ukierunkowanej na markery genetyczne 16S, 18S i ITS, to skuteczna metoda odkrywania filogenezy, taksonomii i liczebności gatunków w społecznościach drobnoustrojów. Podejście to obejmuje sekwencjonowanie hiperzmiennych regionów markerów genetycznych metabolizmu podstawowego. Pierwotnie wprowadzony jako molekularny odcisk palca przezWoeses i inw 1977 r. technika ta zrewolucjonizowała profilowanie mikrobiomów, umożliwiając analizy bez izolacji. Dzięki sekwencjonowaniu 16S (bakterie), 18S (grzyby) i wewnętrznego transkrybowanego odstępnika (ITS, grzyby) badacze mogą zidentyfikować nie tylko gatunki występujące powszechnie, ale także rzadkie i niezidentyfikowane. Powszechnie przyjęte jako kluczowe narzędzie, sekwencjonowanie amplikonów stało się instrumentalne w rozpoznawaniu zróżnicowanego składu drobnoustrojów w różnych środowiskach, w tym w ludzkiej jamie ustnej, jelitach, stolcu i poza nim.
-

Ponowne sekwencjonowanie całego genomu bakterii i grzybów
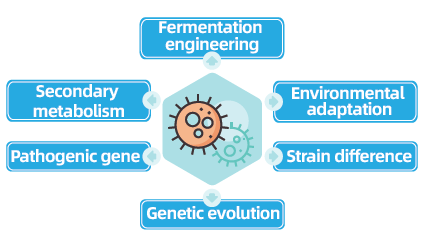
Projekty ponownego sekwencjonowania całego genomu bakterii i grzybów mają kluczowe znaczenie dla postępu w genomice drobnoustrojów, umożliwiając uzupełnienie i porównanie genomów drobnoustrojów. Ułatwia to inżynierię fermentacji, optymalizację procesów przemysłowych i badanie szlaków metabolizmu wtórnego. Co więcej, ponowne sekwencjonowanie grzybów i bakterii ma kluczowe znaczenie dla zrozumienia adaptacji środowiskowej, optymalizacji szczepów i ujawnienia dynamiki ewolucji genetycznej, co ma szerokie implikacje w medycynie, rolnictwie i naukach o środowisku.
-

PacBio – Pełnowymiarowe sekwencjonowanie amplikonów 16S/18S/ITS
Platforma Amplicon (16S/18S/ITS) została opracowana na podstawie wieloletniego doświadczenia w analizie projektów różnorodności mikrobiologicznej, która zawiera standaryzowaną analizę podstawową i analizę spersonalizowaną: analiza podstawowa obejmuje główny nurt analizy bieżących badań mikrobiologicznych, treść analizy jest bogata i wszechstronna, a wyniki analiz prezentowane są w formie raportów z projektów; Treść spersonalizowanej analizy jest zróżnicowana. Można elastycznie wybierać próbki i ustawiać parametry zgodnie z podstawowym raportem z analizy i celem badawczym, aby spełnić spersonalizowane wymagania. System operacyjny Windows, prosty i szybki.
-

Pełnowymiarowy transkryptom PacBio (niereferencyjny)
Pobierając dane sekwencjonowania izoform Pacific Biosciences (PacBio) jako dane wejściowe, ta aplikacja jest w stanie zidentyfikować sekwencje transkryptów pełnej długości (bez składania). Mapując sekwencje pełnej długości względem genomu referencyjnego, transkrypty można optymalizować pod kątem znanych genów, transkryptów, regionów kodujących itp. W tym przypadku można uzyskać dokładniejszą identyfikację struktur mRNA, np. alternatywny splicing itp. Wspólna analiza z danymi sekwencjonowania transkryptomu NGS umożliwia bardziej wszechstronną adnotację i dokładniejszą ocenę ilościową ekspresji na poziomie transkryptu, co w dużej mierze przynosi korzyści w dalszej części ekspresji różnicowej i analizie funkcjonalnej.
-

Sekwencjonowanie wodorosiarczynem o zmniejszonej reprezentacji (RRBS)
Sekwencjonowanie wodorosiarczynem o zmniejszonej reprezentacji (RRBS) okazało się opłacalną i wydajną alternatywą dla sekwencjonowania wodorosiarczynem całego genomu (WGBS) w badaniach nad metylacją DNA. Chociaż WGBS zapewnia kompleksowe spostrzeżenia poprzez badanie całego genomu w rozdzielczości pojedynczej zasady, jego wysoki koszt może być czynnikiem ograniczającym. RRBS strategicznie łagodzi to wyzwanie poprzez selektywną analizę reprezentatywnej części genomu. Metodologia ta opiera się na wzbogacaniu regionów bogatych w wyspy CpG przez rozszczepienie MspI, a następnie selekcję wielkości fragmentów o wielkości 200-500/600 bps. W rezultacie sekwencjonowaniu podlegają tylko regiony położone najbliżej wysp CpG, podczas gdy te z odległymi wyspami CpG są wykluczane z analizy. Proces ten, w połączeniu z sekwencjonowaniem wodorosiarczynem, pozwala na wykrywanie metylacji DNA z wysoką rozdzielczością, a metoda sekwencjonowania, PE150, koncentruje się w szczególności na końcach wstawek, a nie na środku, zwiększając skuteczność profilowania metylacji. RRBS jest nieocenionym narzędziem umożliwiającym opłacalne badania nad metylacją DNA i pogłębiającym wiedzę na temat mechanizmów epigenetycznych.
-

Sekwencjonowanie prokariotycznego RNA
Sekwencjonowanie RNA umożliwia kompleksowe profilowanie wszystkich transkryptów RNA w komórkach w określonych warunkach. Ta najnowocześniejsza technologia stanowi potężne narzędzie, ujawniające skomplikowane profile ekspresji genów, struktury genów i mechanizmy molekularne powiązane z różnorodnymi procesami biologicznymi. Powszechnie stosowane w badaniach podstawowych, diagnostyce klinicznej i opracowywaniu leków, sekwencjonowanie RNA zapewnia wgląd w zawiłości dynamiki komórkowej i regulacji genetycznej. Nasze przetwarzanie próbek prokariotycznego RNA jest dostosowane do transkryptomów prokariotycznych, obejmujące wyczerpywanie rRNA i kierunkowe przygotowanie biblioteki.
Platforma: Illumina NovaSeq
-

Sekwencjonowanie metatranskryptomu
Wykorzystując technologię sekwencjonowania Illumina, usługa sekwencjonowania metatranskryptomu BMKGENE ujawnia dynamiczną ekspresję genów różnorodnego zestawu drobnoustrojów, od eukariontów po prokarioty i wirusy, w środowiskach naturalnych, takich jak gleba, woda, morze, odchody i jelita. Nasza kompleksowa usługa umożliwia badaczom zagłębianie się w kompletne profile ekspresji genów złożonych społeczności drobnoustrojów. Oprócz analizy taksonomicznej nasza usługa sekwencjonowania metatranskryptomów ułatwia badanie wzbogacenia funkcjonalnego, rzucając światło na geny o zróżnicowanej ekspresji i ich role. Odkryj bogactwo spostrzeżeń biologicznych, poruszając się po złożonych krajobrazach ekspresji genów, różnorodności taksonomicznej i dynamiki funkcjonalnej w tych różnorodnych niszach środowiskowych.
-

Zgromadzenie genomu grzyba de novo
BMKGENE oferuje wszechstronne rozwiązania w zakresie genomów grzybów, zaspokajając różnorodne potrzeby badawcze i pożądaną kompletność genomu. Wykorzystanie samego sekwencjonowania Illumina z krótkim odczytem umożliwia wygenerowanie szkicu genomu. Sekwencjonowanie z krótkim i długim odczytem przy użyciu Nanopore lub Pacbio łączy się w celu uzyskania bardziej dopracowanego genomu grzyba z dłuższymi kontigami. Co więcej, integracja sekwencjonowania Hi-C jeszcze bardziej zwiększa możliwości, umożliwiając uzyskanie kompletnego genomu na poziomie chromosomu.