Sekwencjonowanie mRNA oparte na odniesieniach
Cechy
● Schwytanie mRNA Poly przed przygotowaniem biblioteki
● Niezależnie od jakiegokolwiek genomu referencyjnego: na podstawie montażu transkryptów de novo, generując listę unigenów, które są adnotacjami z wieloma bazami danych (NR, Swiss-Prot, COG, KOG, EgGnog, PFAM, Go, Kegg)
● Kompleksowa analiza bioinformatyczna ekspresji genów i struktury transkryptu
Zalety serwisowe
●Rozległa wiedza specjalistyczna: Dzięki osiągnięciu przetwarzania ponad 600 000 próbek w BMKGENE, obejmującym różnorodne typy próbek, takie jak kultury komórkowe, tkanki i płyny ustrojowe, nasz zespół wprowadza bogate doświadczenie w każdym projekcie. Z powodzeniem zamknęliśmy ponad 100 000 projektów mRNA w różnych dziedzinach badań.
●Rygorystyczna kontrola jakości: Wdrażamy podstawowe punkty sterowania na wszystkich etapach, od przygotowania próbek i biblioteki po sekwencjonowanie i bioinformatykę. To skrupulatne monitorowanie zapewnia dostarczanie konsekwentnie wysokiej jakości wyników.
● Kompleksowa adnotacja: Używamy wielu baz danych do funkcjonalnego adnotacji genów o różnej ekspresji (DEG) i przeprowadzania odpowiedniej analizy wzbogacania, zapewniając wgląd w procesy komórkowe i molekularne leżące u podstaw odpowiedzi transkryptomu.
●Wsparcie po sprzedaży: Nasze zobowiązanie wykracza poza zakończenie projektu z 3-miesięcznym okresem usług po sprzedaży. W tym czasie oferujemy obserwację projektu, pomoc w rozwiązywaniu problemów oraz sesje pytań i odpowiedzi, aby rozwiązać wszelkie zapytania związane z wynikami.
Przykładowe wymagania i dostawa
| Biblioteka | Strategia sekwencjonowania | Zalecane dane | Kontrola jakości |
| Poli A wzbogacony | Illumina PE150 DNBSEQ-T7 | 6-10 GB | Q30 ≥85% |
Przykładowe wymagania:
Nukleotydy:
| Conc. (Ng/μl) | Ilość (μg) | Czystość | Uczciwość |
| ≥ 10 | ≥ 0,2 | OD260/280 = 1,7-2,5 OD260/230 = 0,5-2,5 Ograniczone lub brak zanieczyszczenia białka lub DNA pokazanego na żelu. | Dla roślin: rin ≥4,0; Dla zwierząt: rin ≥4,5; 5.0 ≥28S/18S ≥1,0; Ograniczona lub brak wyjściowej wzniesienia |
● Rośliny:
Korzeń, łodyga lub płatek: 450 mg
Liść lub nasiona: 300 mg
Owoce: 1,2 g
● Zwierzę:
Serce lub jelita: 300 mg
Wnętrzności lub mózg: 240 mg
Mięsień: 450 mg
Kości, włosy lub skóra: 1G
● Stawonogi:
Owady: 6G
Crustacea: 300 mg
● Cała krew: 1 rurka
● Komórki: 106 komórki
Zalecana dostawa próbki
Pojemnik: 2 ml rurki wirówki (folia cyny nie jest zalecana)
Etykietowanie przykładowe: grupa+replikuj EG A1, A2, A3; B1, B2, B3.
Wysyłka:
1. Suche ice: Próbki należy pakować w torby i zakopane w suchym lodzie.
2. Rnastable rur: Próbki RNA można wysuszyć w rurce stabilizacji RNA (np. Rnastable®) i wysyłania w temperaturze pokojowej.
Przepływ pracy usług

Projekt eksperymentu

Dostawa próbki

Ekstrakcja RNA

Konstrukcja biblioteki

Sekwencjonowanie

Analiza danych

Usługi po sprzedaży
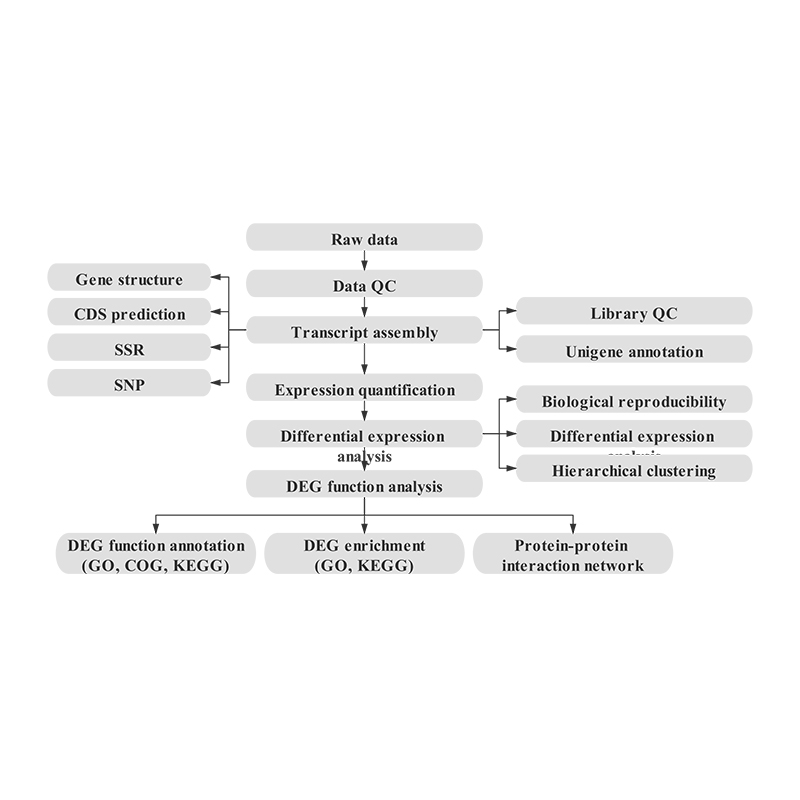
Bioinformatyka
Zespół transkryptomu i wybór unigenu
Adnotacja Unigene
Korelacja próbki i ocena powtórzeń biologicznych
Geny o różnej ekspresji (DEG)
Funkcjonalna adnotacja DEG
Funkcjonalne wzbogacenie DEG
Przeglądaj postępy ułatwione przez eukariotyczne usługi sekwencjonowania MRNA BMKGENE w celu wyselekcjonowanego zbioru publikacji.
Shen, F. i in. (2020) „Zgromadzenie transkryptomu de novo i ekspresja genów o uprzedzeniu płci w gonadach suma amur (Silurus asotus)”, Genomics, 112 (3), s. 2603–2614. doi: 10.1016/j.ygeno.2020.01.026.
Zhang, C. i in. (2016) „Analiza transkryptomu metabolizmu sacharozy podczas obrzęku i rozwoju cebuli (Allium Cepa L.)”, Frontiers in Plant Science, 7 (wrzesień), s. 1. 212763. Doi: 10.3389/fpls.2016.01425/bibtex.
Zhu, C. i in. (2017) „Zgromadzenie de novo, charakterystyka i adnotacja dla transkryptomu Sarcocheilichthys sinensis”, PLOS One, 12 (2). doi: 10.1371/journal.pone.0171966.
Zou, L. i in. (2021) „Analiza transkryptomu de novo zapewnia wgląd w tolerancję soli podocarpus makrofilus pod stresem zasolenia”, BMC Plant Biology, 21 (1), s. 1–17. DOI: 10.1186/S12870-021-03274-1/Figures/9.