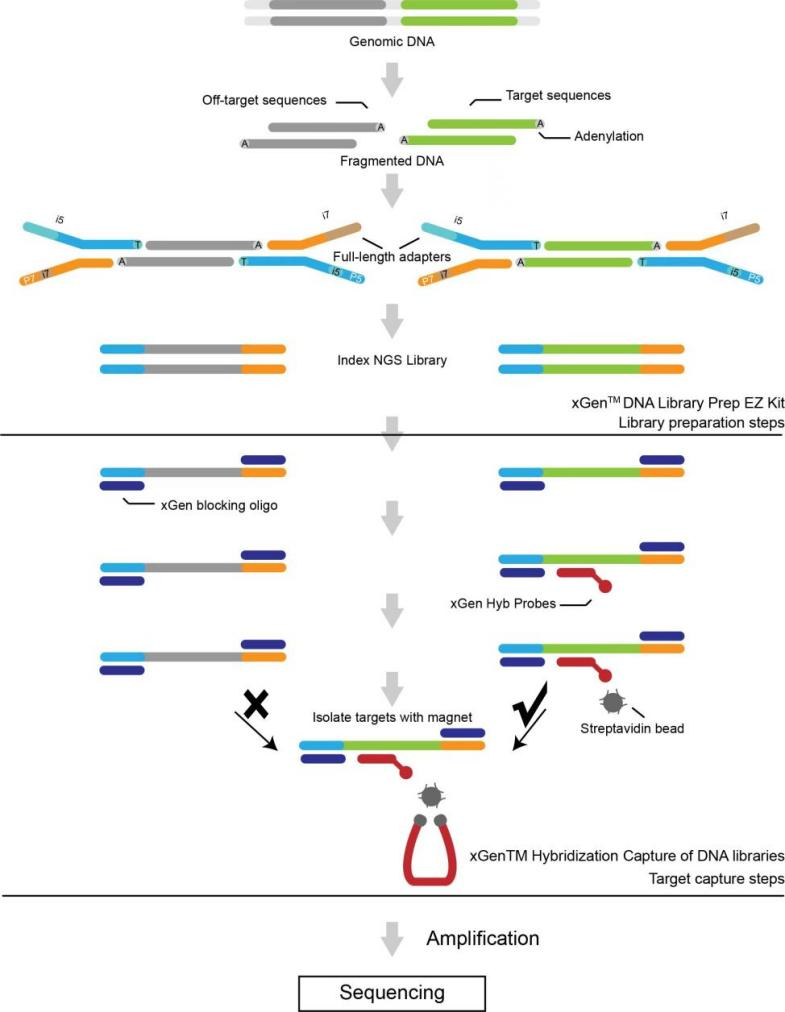
Sekwencjonowanie całego egzomu człowieka
Funkcje usługi
● Dostępne są dwa panele egzomów oparte na wzbogacaniu obiektów docelowych sondami: Sure Select Human All Exon v6 (Agilent) i xGen Exome Hybridization Panel v2 (IDT).
● Sekwencjonowanie na Illumina NovaSeq.
● Potok bioinformatyczny ukierunkowany na analizę chorób lub analizę nowotworów.
Zalety serwisu
●Celuje w region kodujący białko: Wychwytując i sekwencjonując regiony kodujące białka, hWES wykorzystuje się do ujawniania wariantów związanych ze strukturą białka.
●Opłacalne:hWES daje około 85% mutacji związanych z chorobami ludzkimi z 1% ludzkiego genomu.
●Wysoka dokładność: Dzięki dużej głębokości sekwencjonowania hWES ułatwia wykrywanie zarówno powszechnych wariantów, jak i rzadkich wariantów z częstotliwościami niższymi niż 1%.
●Rygorystyczna kontrola jakości: Wdrażamy pięć podstawowych punktów kontroli na wszystkich etapach, od przygotowania próbek i bibliotek po sekwencjonowanie i bioinformatykę. To skrupulatne monitorowanie zapewnia niezmiennie wysoką jakość wyników.
●Kompleksowa analiza bioinformatyczna: nasz plan wykracza poza identyfikację zmian w genomie referencyjnym, ponieważ obejmuje zaawansowaną analizę zaprojektowaną specjalnie w celu odpowiedzi na pytania badawcze związane z genetycznymi aspektami chorób lub analizą nowotworów.
●Wsparcie posprzedażowe:Nasze zaangażowanie wykracza poza realizację projektu i obejmuje 3-miesięczny okres obsługi posprzedażnej. W tym czasie oferujemy monitorowanie projektu, pomoc w rozwiązywaniu problemów oraz sesje pytań i odpowiedzi, aby odpowiedzieć na wszelkie pytania związane z wynikami.
Przykładowe specyfikacje
| Strategia przechwytywania egzonów | Strategia sekwencjonowania | Zalecane wyjście danych |
| Sure Select Human All Exon v6 (Agilent) lub panel hybrydyzacyjny xGen Exome v2 (IDT)
| Illumina NovaSeq PE150 | 5–10 Gb W przypadku zaburzeń mendlowskich/rzadkich chorób: > 50x Dla próbek nowotworu: ≥ 100x |
Przykładowe wymagania
| Typ próbki
| Kwota(Qubit®)
| Stężenie | Tom
| Czystość (NanoDrop™) |
|
Genomowe DNA
| ≥ 50 ng | ≥ 6 ng/µl | ≥ 15 µl | OD260/280=1,8-2,0 bez degradacji i bez zanieczyszczeń
|
Bioinformatyka

Analiza bioinformatyczna próbek z chorobą hWES obejmuje:
● Kontrola jakości danych sekwencjonowania
● Referencyjne dopasowanie genomu
● Identyfikacja SNP i InDels
● Adnotacja funkcjonalna SNP i InDels

Analiza bioinformatyczna próbek nowotworu obejmuje:
● Kontrola jakości danych sekwencjonowania
● Referencyjne dopasowanie genomu
● Identyfikacja SNP, InDels i odmian somatycznych
● Identyfikacja wariantów linii zarodkowej
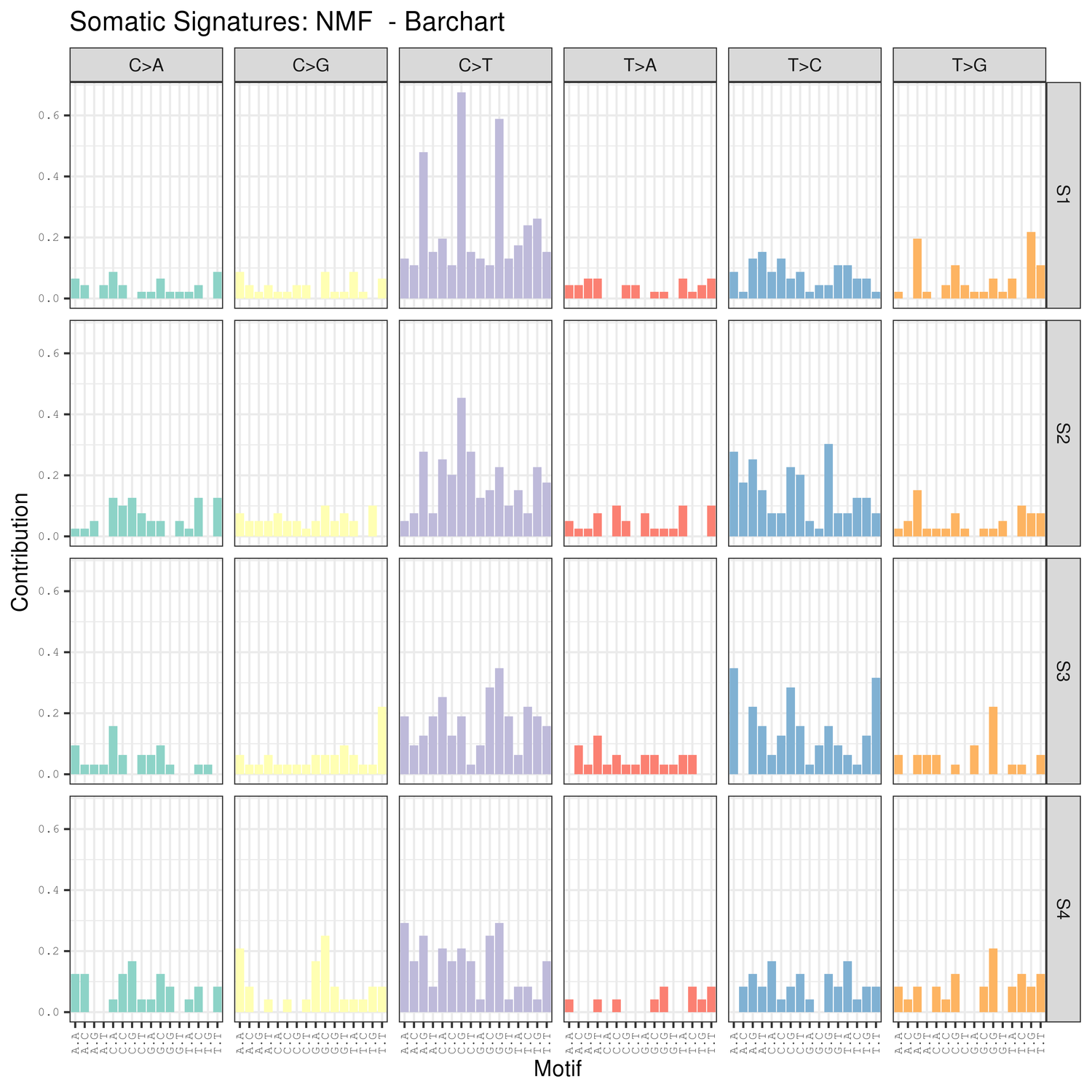
● Analiza sygnatur mutacji
● Identyfikacja genów napędowych w oparciu o mutacje polegające na wzmocnieniu funkcji
● Adnotacja o mutacji na poziomie wrażliwości na lek
● Analiza heterogeniczności – obliczenie czystości i ploidalności
Przebieg prac serwisowych

Dostawa próbek

Ekstrakcja DNA

Budowa biblioteki

Sekwencjonowanie

Analiza danych

Dostarczanie danych

Usługi posprzedażowe
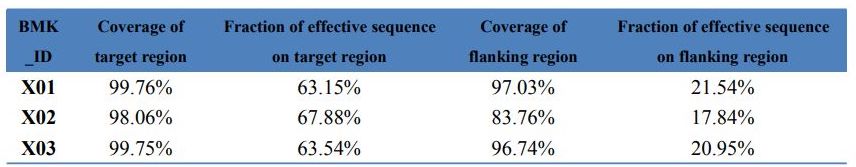
Data QC – statystyki przechwytywania egzomu
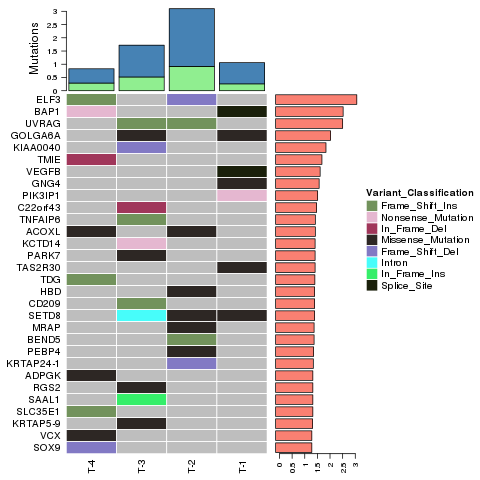
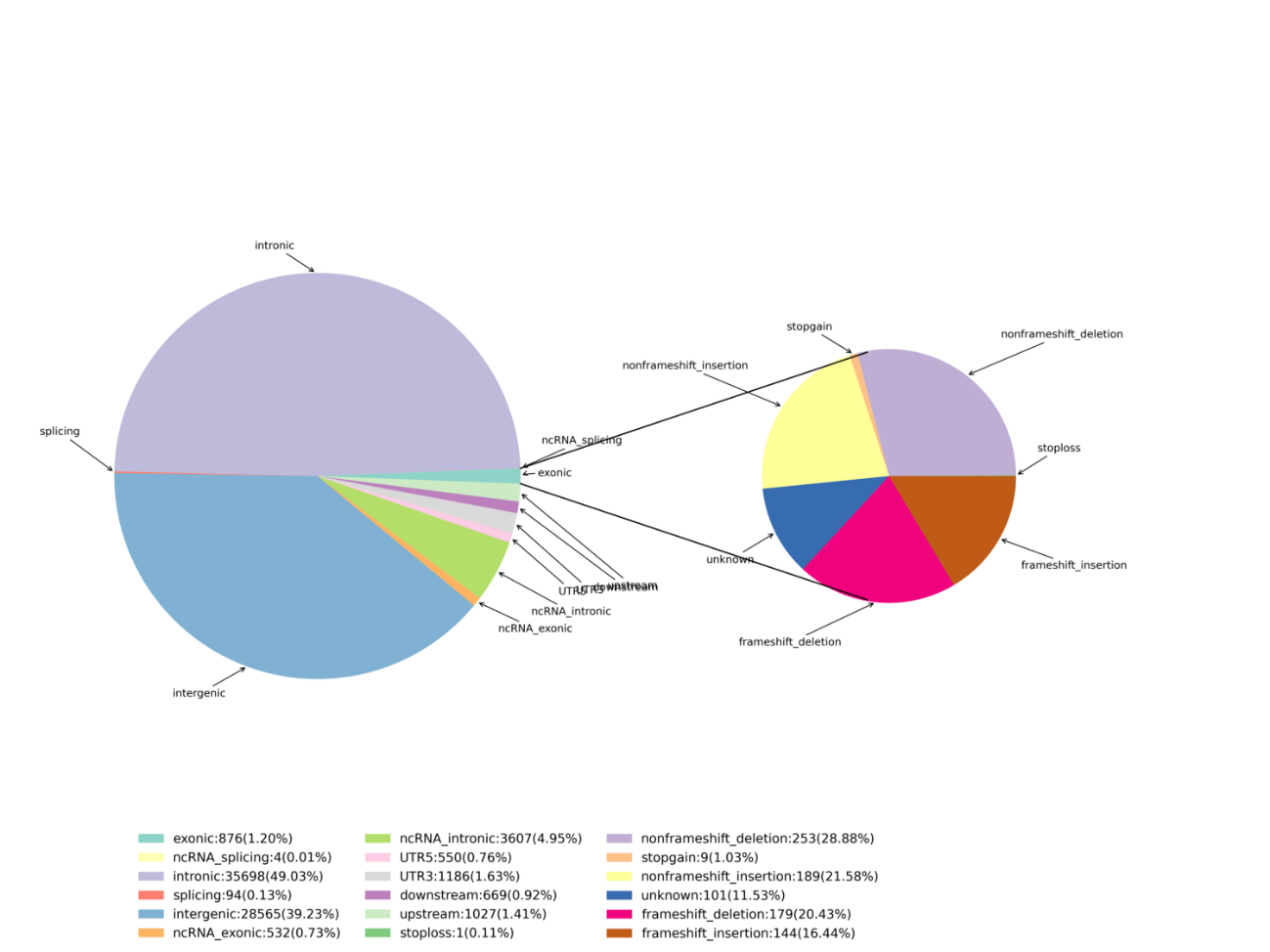
Identyfikacja wariantu – InDels
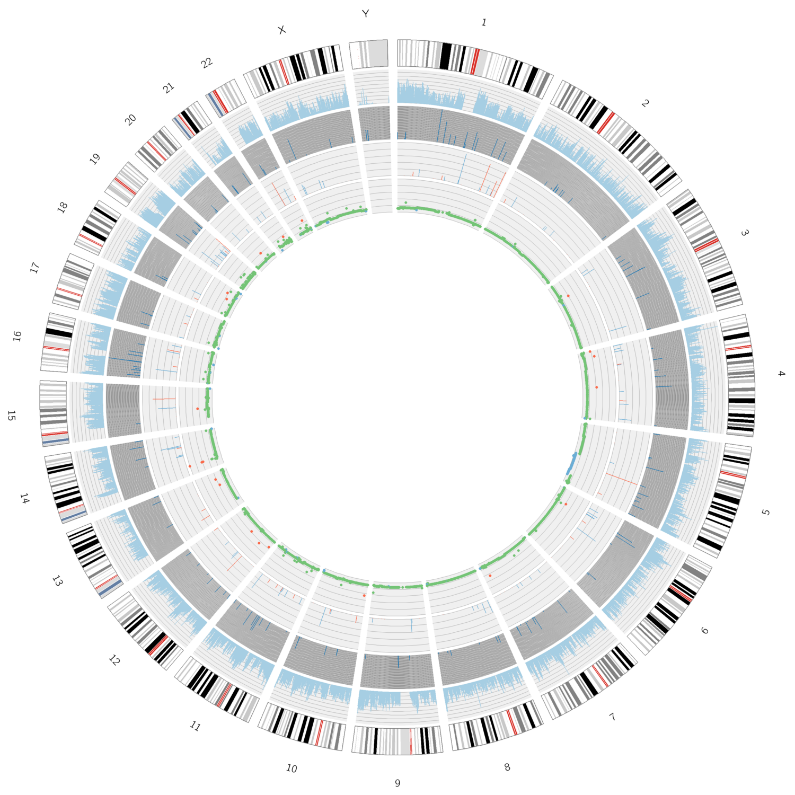
Zaawansowana analiza: identyfikacja i dystrybucja szkodliwych SNP/InDels – wykres Circos
Analiza nowotworu: identyfikacja i rozkład mutacji somatycznych – wykres Circos
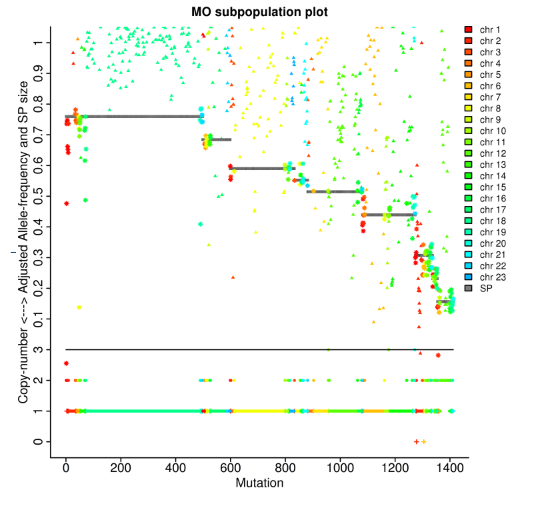
Analiza nowotworu: linie klonalne