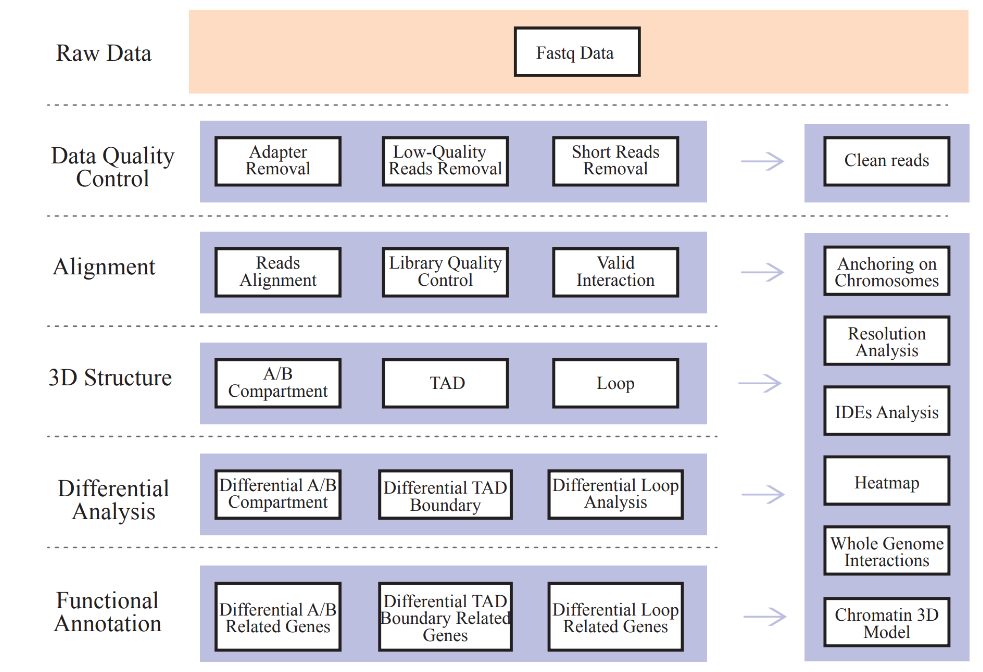
Interakcja chromatyny oparta na Hi-C
Funkcje usługi
● Sekwencjonowanie na Illumina NovaSeq z PE150.
● Usługa wymaga, aby próbki tkanek, a nie wyekstrahowane kwasy nukleinowe, sieciowały się formaldehydem i chroniły interakcje DNA-białko.
● Eksperyment Hi-C obejmuje ograniczenie i naprawę lepkich końcówek za pomocą biotyny, a następnie cyrkulację powstałych tępych końcówek przy zachowaniu interakcji. Następnie DNA ściąga się za pomocą kulek streptawidyny i oczyszcza w celu późniejszego przygotowania biblioteki.
Zalety serwisu
●Optymalny projekt enzymu restrykcyjnego: aby zapewnić wysoką skuteczność Hi-C w przypadku różnych gatunków z maksymalnie 93% prawidłowymi parami interakcji.
●Obszerna wiedza specjalistyczna i dokumentacja publikacyjna:BMKGene ma ogromne doświadczenie z >2000 projektami sekwencjonowania Hi-C z 800 różnych gatunków i różnymi patentami. Ponad 100 opublikowanych przypadków o skumulowanym współczynniku wpływu ponad 900.
●Wysoko wykwalifikowany zespół bioinformatyki:z własnymi patentami i prawami autorskimi do oprogramowania do eksperymentów Hi-C i analizy danych oraz samodzielnie opracowanym oprogramowaniem do wizualizacji danych.
●Wsparcie posprzedażowe:Nasze zaangażowanie wykracza poza realizację projektu i obejmuje 3-miesięczny okres obsługi posprzedażnej. W tym czasie oferujemy monitorowanie projektu, pomoc w rozwiązywaniu problemów oraz sesje pytań i odpowiedzi, aby odpowiedzieć na wszelkie pytania związane z wynikami.
●Obszerna adnotacja: korzystamy z wielu baz danych, aby funkcjonalnie opisywać geny ze zidentyfikowanymi odmianami i przeprowadzać odpowiednią analizę wzbogacania, zapewniając wgląd w wiele projektów badawczych.
Specyfikacje usług
| Biblioteka | Strategia sekwencjonowania | Zalecane wyjście danych | Rozdzielczość sygnału Hi-C |
| Biblioteka Hi-C | Illumina PE150 | Pętla chromatyny: 150x TAD: 50x | Pętla chromatyny: 10 Kb TAD: 40 KB |
Wymagania serwisowe
| Typ próbki | Wymagana ilość |
| Tkanka zwierzęca | ≥2g |
| Pełna krew | ≥2 ml |
| Grzyby | ≥1g |
| Roślina- młoda tkanka | 1 g na porcję, zalecane 2-4 porcje |
| Hodowane komórki | ≥1x107 |
Zawiera następującą analizę:
● Kontrola jakości surowych danych;
● Mapowanie i biblioteka Hi-C QC: prawidłowe pary interakcji i wykładniki zaniku interakcji (IDE);
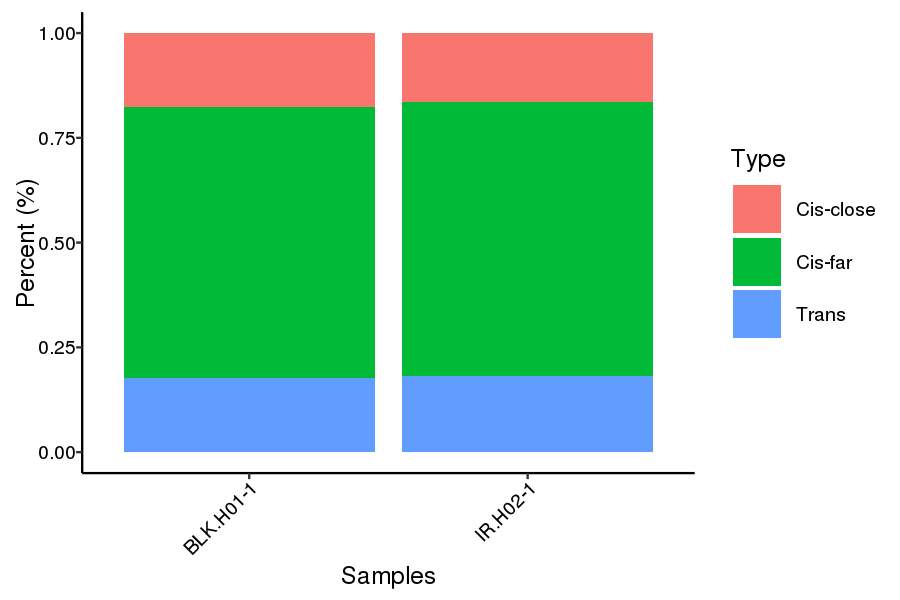
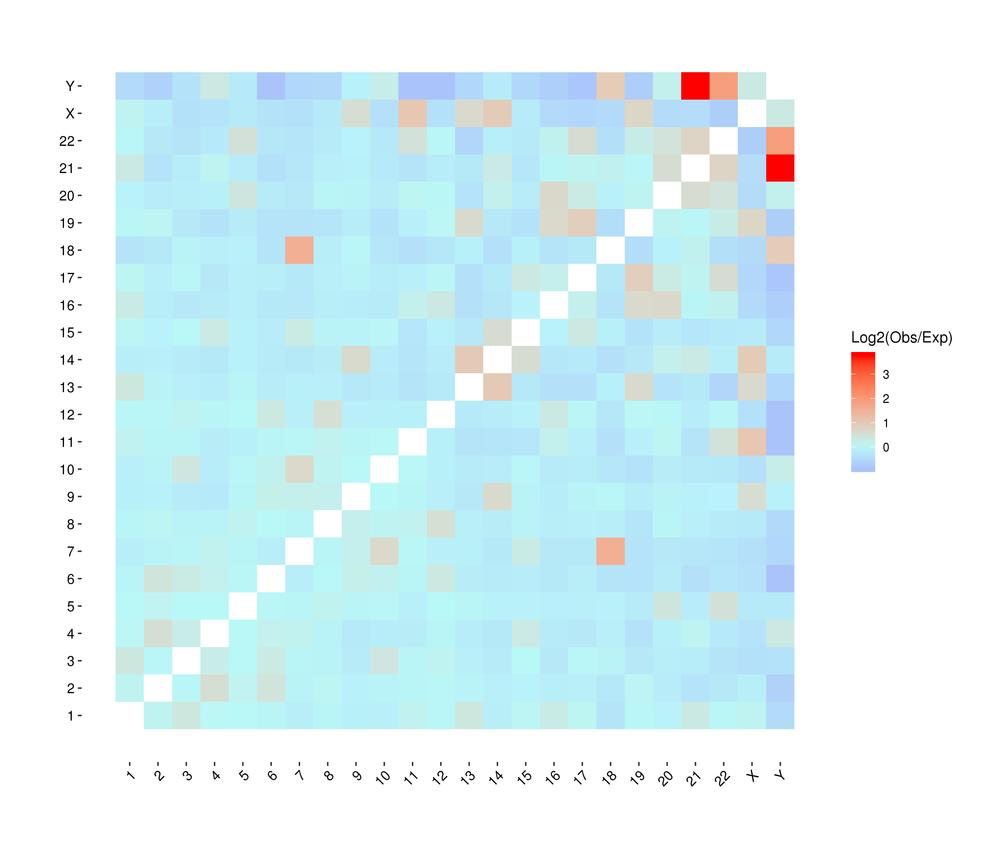
● Profilowanie interakcji obejmujące cały genom: analiza cis/trans i mapa interakcji Hi-C;
● Analiza rozkładu przedziałów A/B;
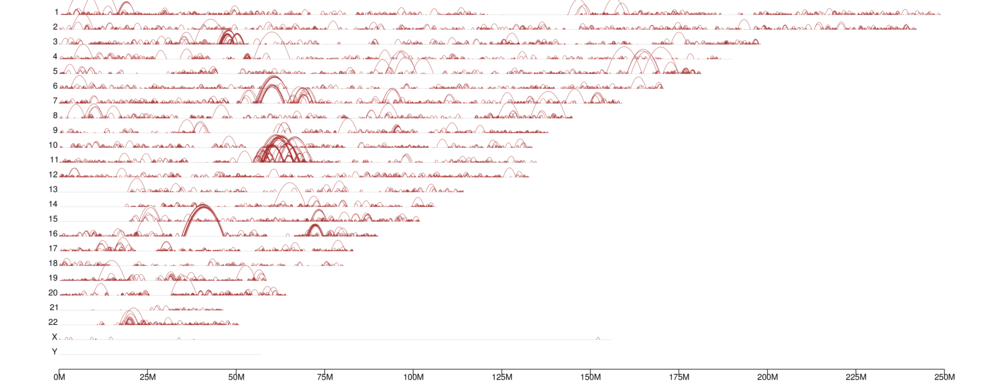
● Identyfikacja TAD i pętli chromatyny;
● Analiza różnicowa elementów struktury chromatyny 3D wśród próbek i odpowiednia adnotacja funkcjonalna powiązanych genów.
Rozkład proporcji cis i trans
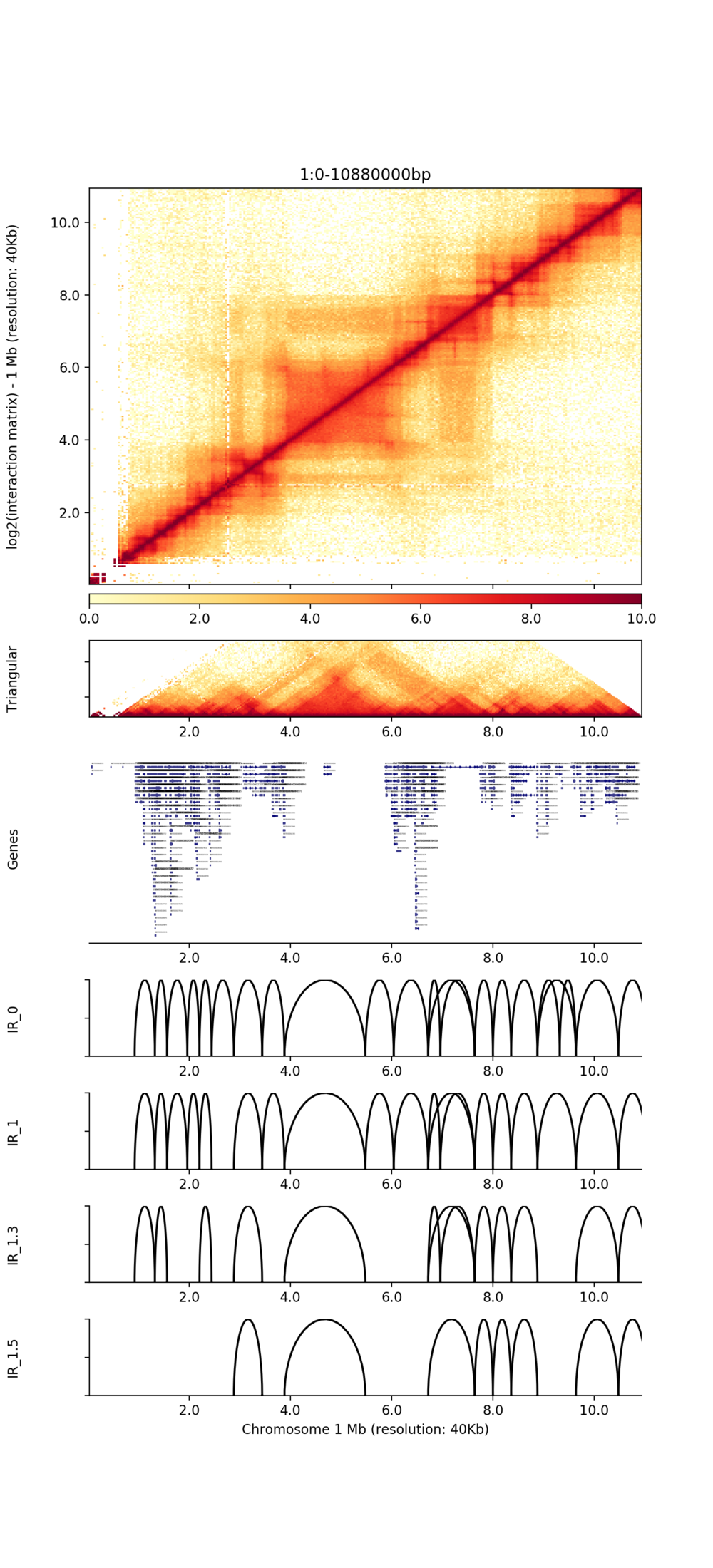
Mapa cieplna interakcji chromosomalnych między próbkami
Rozkład przedziałów A/B w całym genomie
Rozkład pętli chromatyny w całym genomie
Wizualizacja TAD
Zapoznaj się z postępami badawczymi, jakie umożliwiły usługi sekwencjonowania Hi-C firmy BMKGene, poprzez wyselekcjonowany zbiór publikacji.
Meng, T. i in. (2021) „Porównawcza zintegrowana analiza multiomiczna identyfikuje CA2 jako nowy cel dla struniaka”,Neuro-Onkologia, 23(10), s. 1709–1722. doi: 10.1093/NEUONC/NOAB156.
Xu, L. i in. (2021) „Dezorganizacja 3D i rearanżacja genomu zapewniają wgląd w patogenezę NAFLD poprzez zintegrowane sekwencjonowanie Hi-C, Nanopore i RNA”,Acta Pharmaceutica Sinica B, 11(10), s. 3150–3164. doi: 10.1016/J.APSB.2021.03.022.