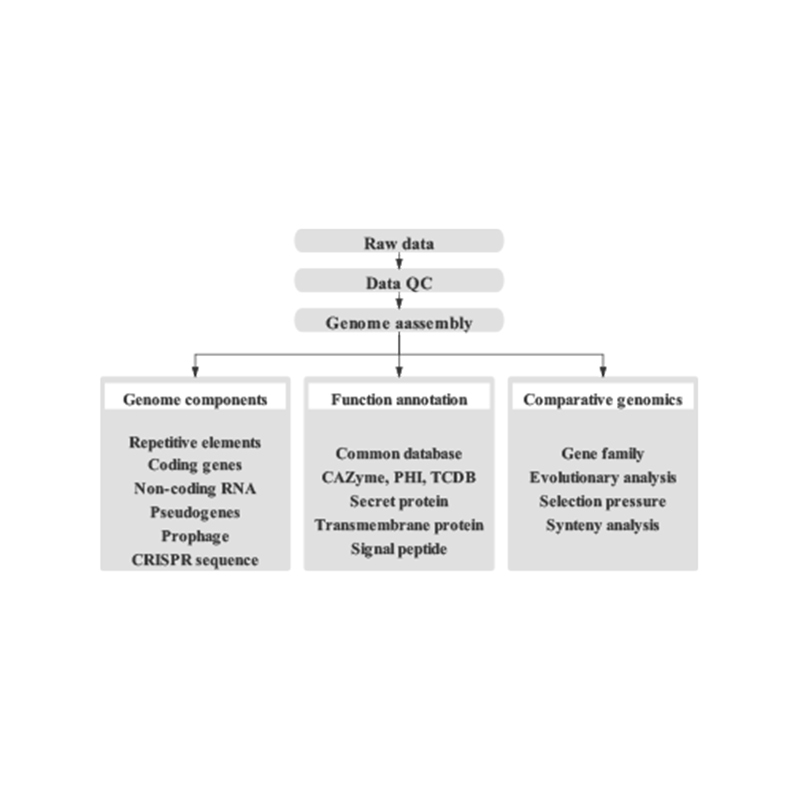
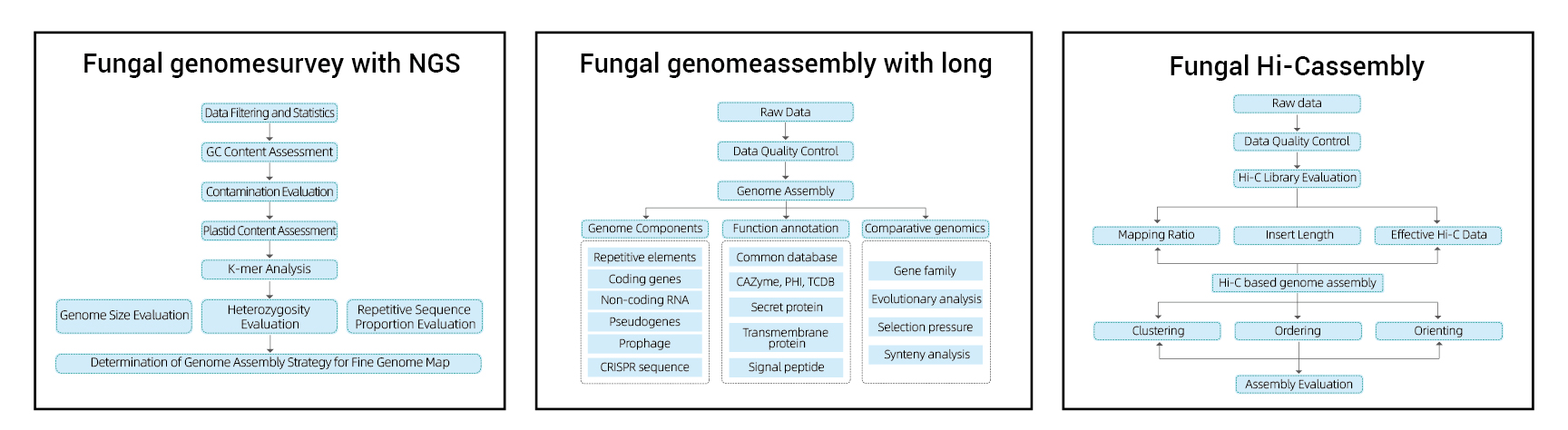
Zgromadzenie genomu grzyba de novo
Funkcje usługi
Z trzema możliwymi opcjami do wyboru w zależności od pożądanego stopnia kompletności genomu:
● Opcja szkicu genomu: sekwencjonowanie z krótkim odczytem za pomocą Illumina NovaSeq PE150.
● Opcja drobnego genomu grzybów:
Badanie genomu: Illumina NovaSeq PE150.
Montaż genomu: PacBio Revio (odczyt HiFi) lub Nanopore PromethION 48.
● Genom grzyba na poziomie chromosomu:
Badanie genomu: Illumina NovaSeq PE150.
Montaż genomu: PacBio Revio (odczyt HiFi) lub Nanopore PromethION 48.
Kotwienie Contig z montażem Hi-C.
Zalety serwisu
●Dostępnych jest wiele strategii sekwencjonowania: Dla różnych celów badawczych i wymagań kompletności genomu
●Kompletny proces bioinformatyki:Obejmuje to składanie genomu i przewidywanie wielu elementów genomu, adnotację funkcjonalnego genu i zakotwiczenie kontigu.
●Rozległa wiedza specjalistyczna: Dzięki zgromadzeniu ponad 12 000 genomów drobnoustrojów zapewniamy ponad dziesięcioletnie doświadczenie, wysoko wykwalifikowany zespół analityczny, obszerną treść i doskonałe wsparcie posprzedażowe.
●Wsparcie posprzedażowe:Nasze zaangażowanie wykracza poza realizację projektu i obejmuje 3-miesięczny okres obsługi posprzedażnej. W tym czasie oferujemy monitorowanie projektu, pomoc w rozwiązywaniu problemów oraz sesje pytań i odpowiedzi, aby odpowiedzieć na wszelkie pytania związane z wynikami.
Specyfikacje usług
| Praca | Strategia sekwencjonowania | Kontrola jakości |
| Projekt genomu | Illumina PE150 100x | Q30≥85% |
| Świetny genom | Badanie genomu: Illumina PE150 50 x Montaż: PacBio HiFi 30x lub Nanopore 100x | contig N50 ≥1Mb (Pacbio Unicell) kontig N50 ≥2Mb (ONT jednokomórkowy) kontig N50 ≥500kb (Inne) |
| Genom na poziomie chromosomu | Badanie genomu: Illumina PE150 50 x Montaż: PacBio HiFi 30x lub Nanopore 100x Zespół Hi-C 100x | Współczynnik zakotwiczenia Contig>90%
|
Wymagania serwisowe
| Stężenie (ng/µL) | Całkowita ilość (µg) | Objętość (µL) | OD260/280 | OD260/230 | |
| PacBio | ≥20 | ≥2 | ≥20 | 1,7-2,2 | ≥1,6 |
| Nanopor | ≥40 | ≥2 | ≥20 | 1,7-2,2 | 1,0-3,0 |
| Ilumina | ≥1 | ≥0,06 | ≥20 | - | - |
Grzyb jednokomórkowy: ≥3,5x1010 komórki
Makro Grzyb: ≥10 g
Przebieg prac serwisowych

Dostawa próbek

Budowa biblioteki

Sekwencjonowanie

Analiza danych

Usługi posprzedażowe
Zawiera następującą analizę:
Badanie genomu:
- Kontrola jakości danych sekwencjonowania
- Ocena genomu: wielkość, heterozygotyczność, elementy powtarzalne
Drobny montaż genomu:
- Kontrola jakości danych sekwencjonowania
- Od nowaMontaż
- Analiza składników genomu: przewidywanie CDS i wielu elementów genomu
- Adnotacja funkcjonalna z wieloma ogólnymi bazami danych (GO, KEGG itp.) i zaawansowanymi bazami danych (CARD, VFDB itp.)
Montaż Hi-C:
- Ocena biblioteki Hi-C.
- Kontigi zakotwiczające grupowanie, porządkowanie i orientowanie
- Ocena złożenia HI-C: Na podstawie genomu referencyjnego i mapy cieplnej
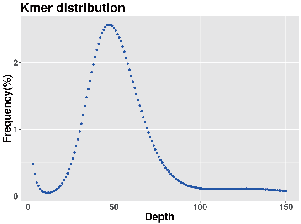
Badanie genomu: rozkład k-merów
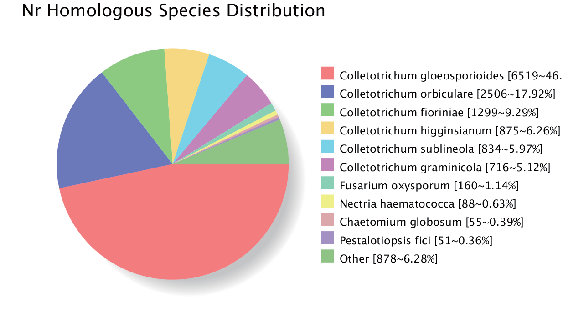
Składanie genomu: adnotacja homologiczna genu (baza danych NR)
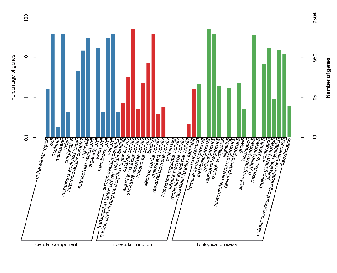
Składanie genomu: funkcjonalna adnotacja genu (GO)
Zapoznaj się z postępami, jakie umożliwiają usługi składania genomu grzybów BMKGene, poprzez wyselekcjonowany zbiór publikacji.
Hao, J. i in. (2023) „Zintegrowane profilowanie omiczne grzyba leczniczego Inonotus obliquus w warunkach zanurzenia”,BMC Genomika, 24(1), s. 1–12. doi: 10.1186/S12864-023-09656-Z/FIGURY/3.
Lu, L. i in. (2023) „Sekwencjonowanie genomu ujawnia ewolucję i mechanizmy chorobotwórcze Rhizoctonia zbożowego, patogenu ostrej plamistości pszenicy”,Dziennik Upraw, 11(2), s. 405–416. doi: 10.1016/J.CJ.2022.07.024.
Zhang, H. i in. (2023) „Zasoby genomu czterech gatunków Clarireedia powodujących plamistość dolara na różnorodnych trawach darniowych”,Choroba roślin, 107(3), s. 929–934. doi: 10.1094/PDIS-08-22-1921-A
Zhang, SS i in. (2023) „Genetyczne i molekularne dowody czterobiegunowego systemu kojarzenia u grzyba jadalnego Grifola frondosa”,Dziennik grzybów, 9(10), s. 1. 959. doi: 10.3390/JOF9100959/S1.