Nanopor sekwencjonowania mRNA o pełnej długości
Cechy
● Wychwycenie mRNA Poly-A, a następnie syntezy cDNA i przygotowanie biblioteki
● Sekwencjonowanie transkryptów pełnej długości
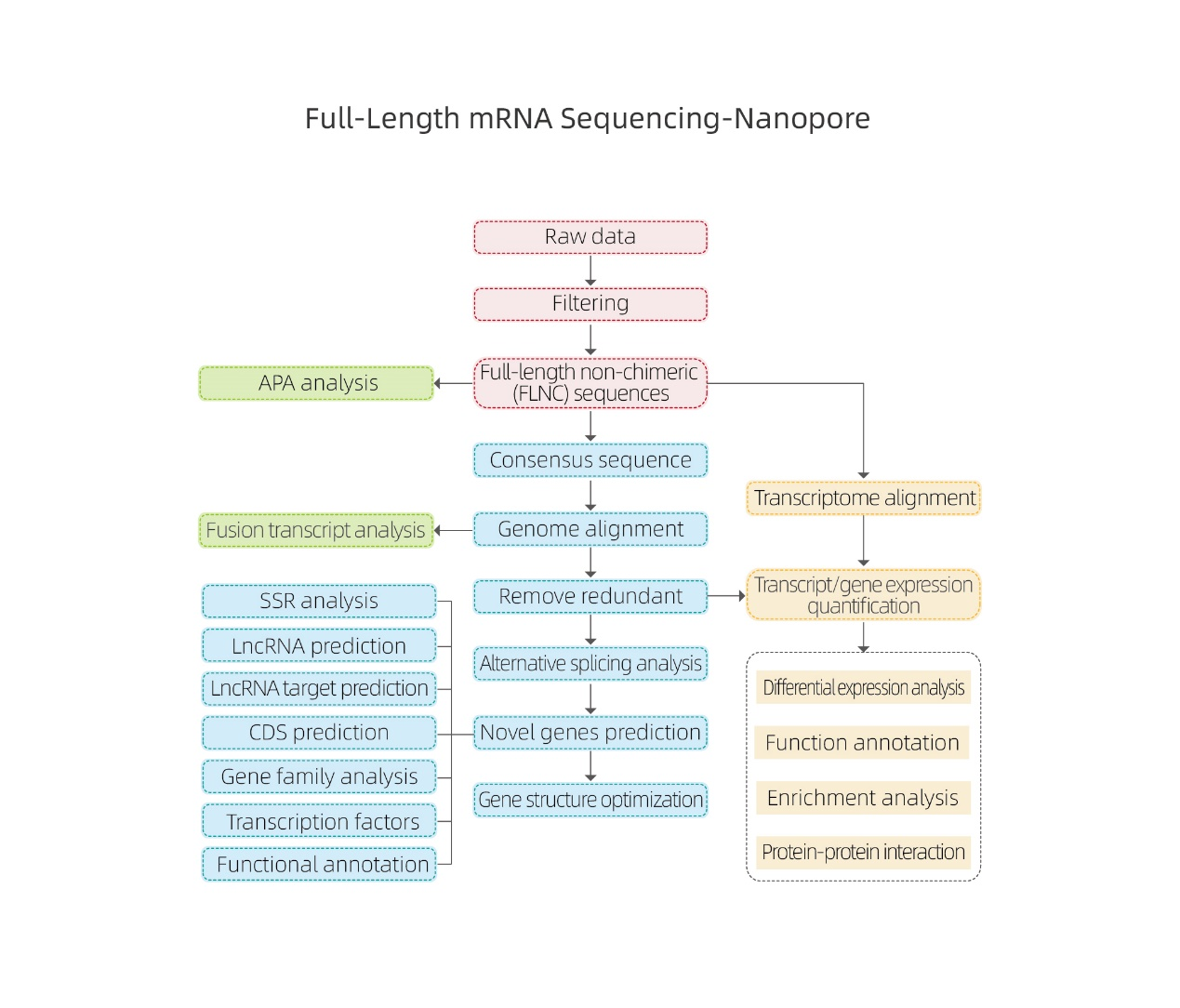
● Analiza bioinformatyczna oparta na wyrównaniu do genomu referencyjnego
● Analiza bioinformatyczna obejmuje nie tylko ekspresję na poziomie genów i izoform, ale także analizę lncRNA, fuzje genów, poli-adenylację i strukturę genów
Zalety serwisowe
●Ocena ilościowa ekspresji na poziomie izoform: Włączając szczegółową i dokładną analizę ekspresji, odsłonięcie zmiany, które można maskować podczas analizy ekspresji całego genu
●Zmniejszone wymagania danych:W porównaniu do sekwencjonowania nowej generacji (NGS) sekwencjonowanie nanoporów wykazuje niższe wymagania danych, umożliwiając równoważne poziomy nasycenia kwantyfikacji ekspresji genów mniejszymi danymi.
●Wyższa dokładność kwantyfikacji ekspresji: zarówno na poziomie genów, jak i izoform
●Identyfikacja dodatkowych informacji transkryptomicznych: Alternatywna poliadenylacja, geny fuzyjne i lcnRNA i ich geny docelowe
●Rozległa wiedza specjalistyczna: Nasz zespół wnosi bogate doświadczenie w każdym projekcie, po ukończeniu ponad 850 nanoporowych projektów transkryptomu i przetworzył ponad 8 000 próbek.
●Wsparcie po sprzedaży: Nasze zobowiązanie wykracza poza zakończenie projektu z 3-miesięcznym okresem usług po sprzedaży. W tym czasie oferujemy obserwację projektu, pomoc w rozwiązywaniu problemów oraz sesje pytań i odpowiedzi, aby rozwiązać wszelkie zapytania związane z wynikami.
Przykładowe wymagania i dostawa
| Biblioteka | Strategia sekwencjonowania | Zalecane dane | Kontrola jakości |
| Poli A wzbogacony | Illumina PE150 | 6/12 GB | Średni wynik jakości: Q10 |
Przykładowe wymagania:
Nukleotydy:
| Conc. (Ng/μl) | Ilość (μg) | Czystość | Uczciwość |
| ≥ 100 | ≥ 1,0 | OD260/280 = 1,7-2,5 OD260/230 = 0,5-2,5 Ograniczone lub brak zanieczyszczenia białka lub DNA pokazanego na żelu. | Dla roślin: rin ≥7,0; Dla zwierząt: rin ≥7,5; 5.0 ≥28S/18S ≥1,0; Ograniczona lub brak wyjściowej wzniesienia |
● Rośliny:
Korzeń, łodyga lub płatek: 450 mg
Liść lub nasiona: 300 mg
Owoce: 1,2 g
● Zwierzę:
Serce lub jelita: 300 mg
Wnętrzności lub mózg: 240 mg
Mięsień: 450 mg
Kości, włosy lub skóra: 1G
● Stawonogi:
Owady: 6G
Crustacea: 300 mg
● Cała krew: 1 rurka
● Komórki: 106 komórki
Zalecana dostawa próbki
Pojemnik: 2 ml rurki wirówki (folia cyny nie jest zalecana)
Etykietowanie przykładowe: grupa+replikuj EG A1, A2, A3; B1, B2, B3.
Wysyłka:
1. Suche ice: Próbki należy pakować w torby i zakopane w suchym lodzie.
2. Rnastable rur: Próbki RNA można wysuszyć w rurce stabilizacji RNA (np. Rnastable®) i wysyłania w temperaturze pokojowej.
Przepływ pracy usług
Nukleotydy:

Dostawa próbki

Konstrukcja biblioteki

Sekwencjonowanie

Analiza danych

Usługi po sprzedaży
Przepływ pracy usług
Tkanka:

Projekt eksperymentu

Dostawa próbki

Ekstrakcja RNA

Konstrukcja biblioteki

Sekwencjonowanie

Analiza danych

Usługi po sprzedaży
● Przetwarzanie danych surowych
● Identyfikacja transkrypcji
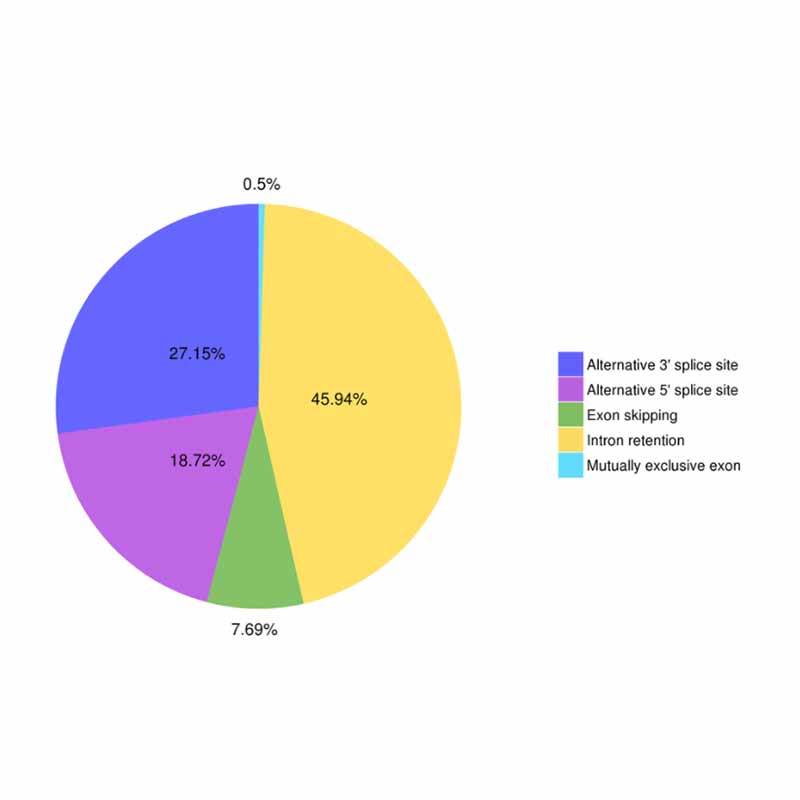
● Alternatywne splicing
● Kwantyfikacja ekspresji na poziomie genów i poziomu izoform
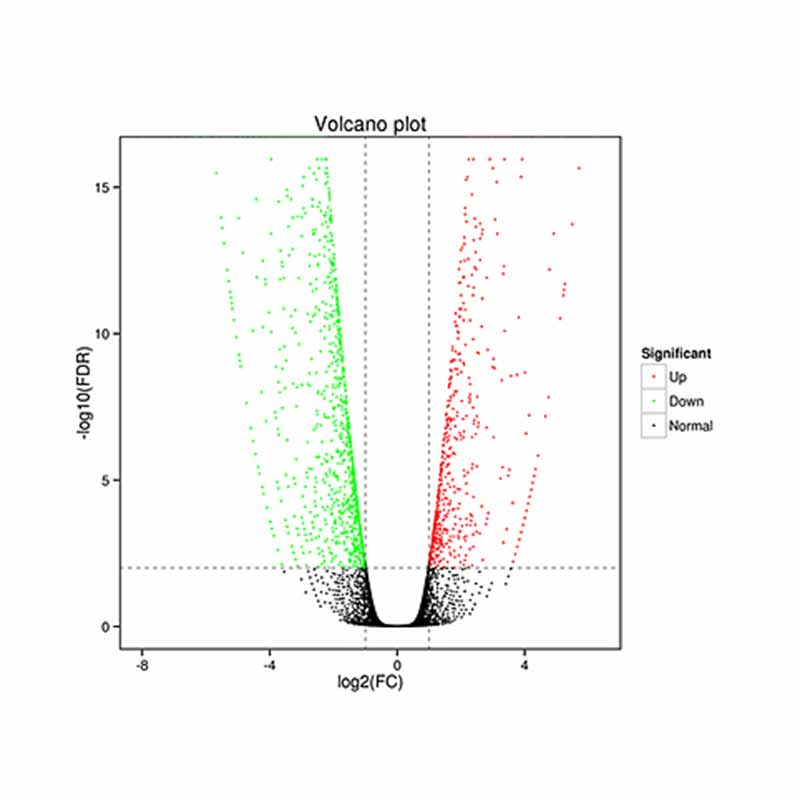
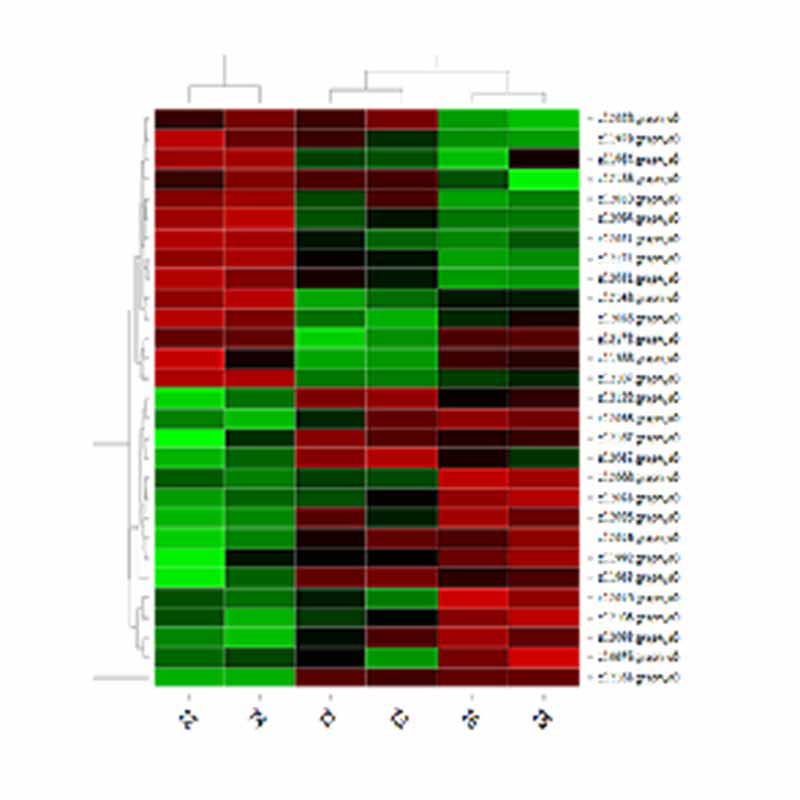
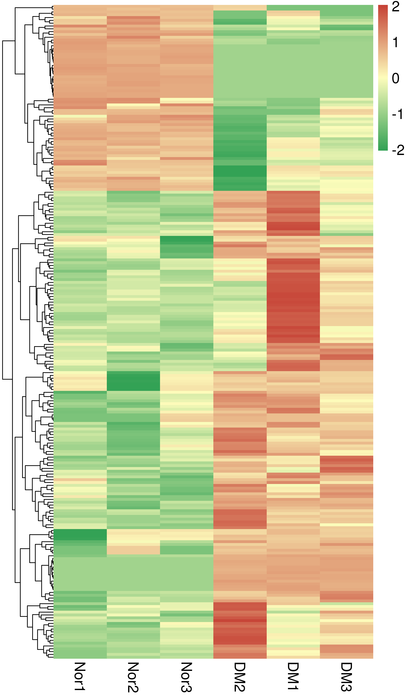
● Analiza różnicowa wyrażenia
● Adnotacja funkcji i wzbogacenie (DEGS i DETS)
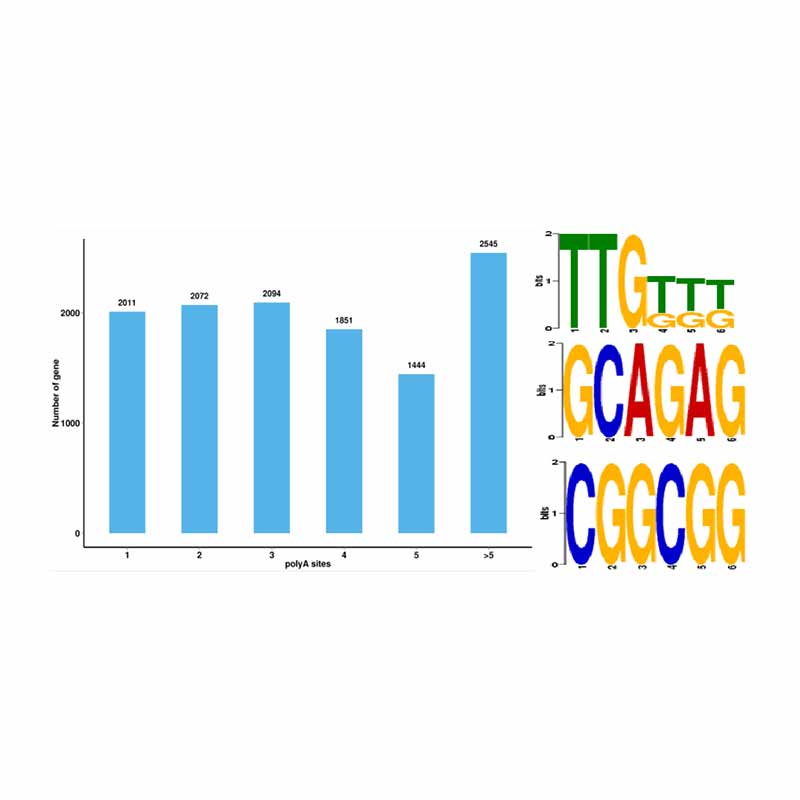
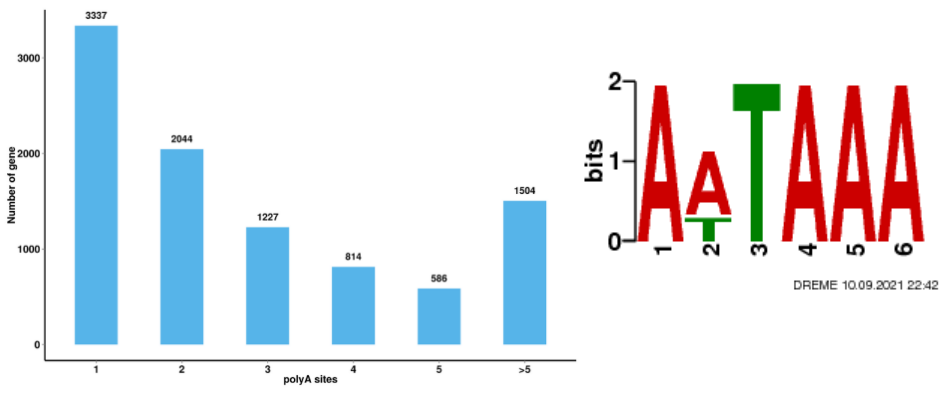
Alternatywna analiza splicingu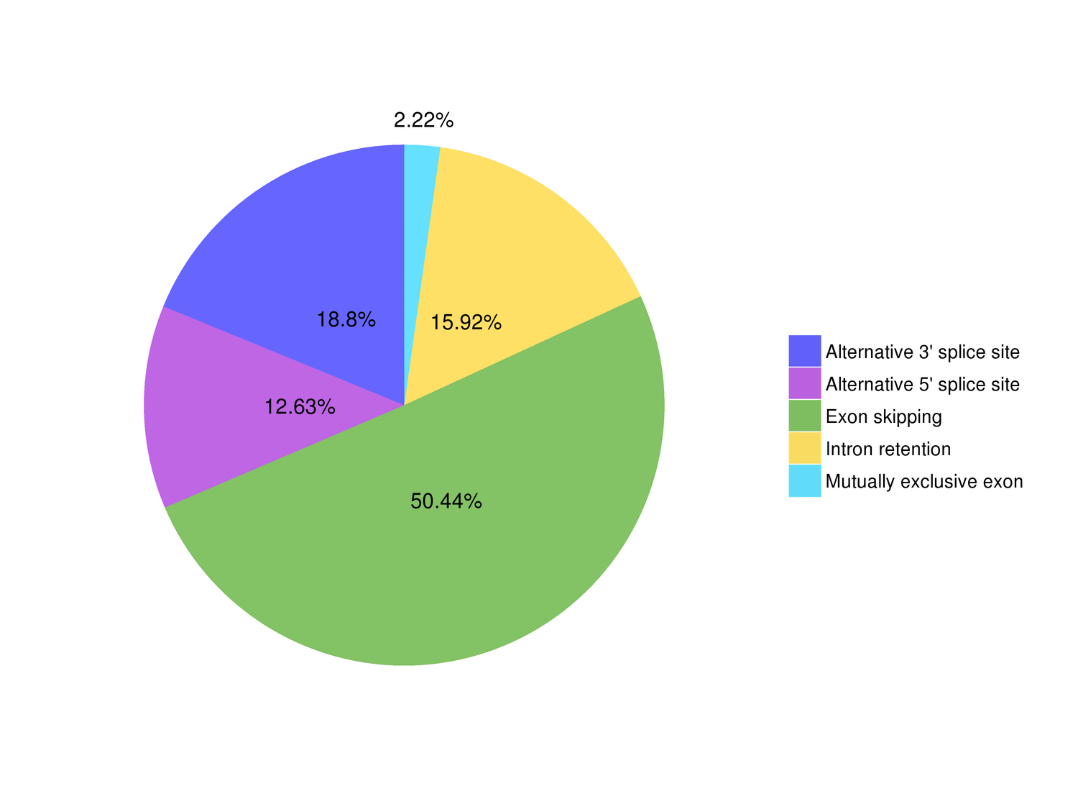 Alternatywna analiza poliadenylacji (APA)
Alternatywna analiza poliadenylacji (APA)
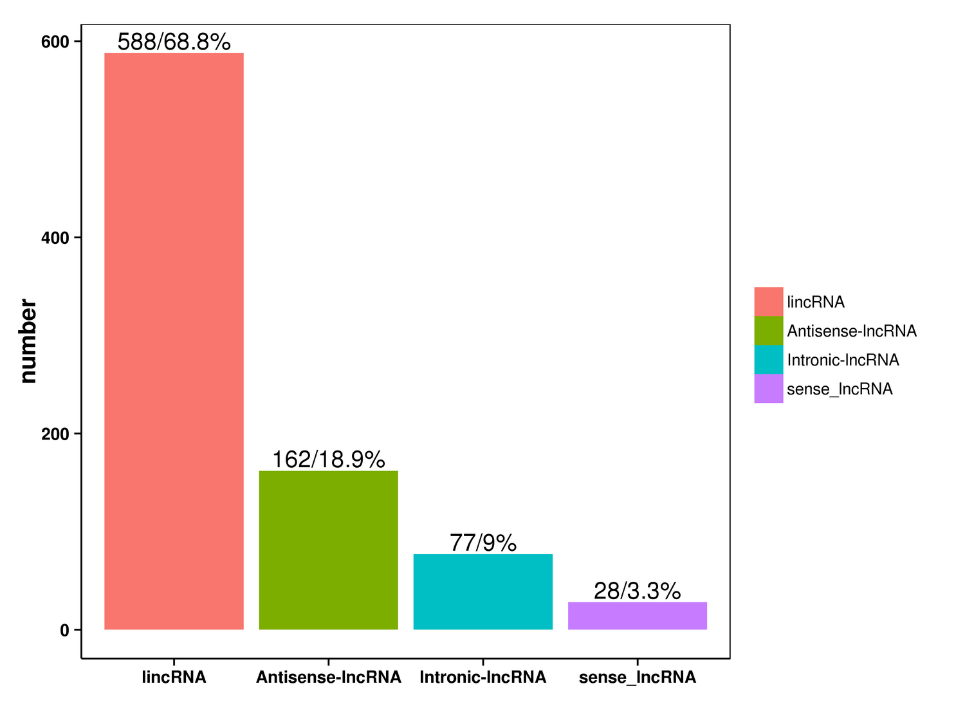
prognoza lncRNA
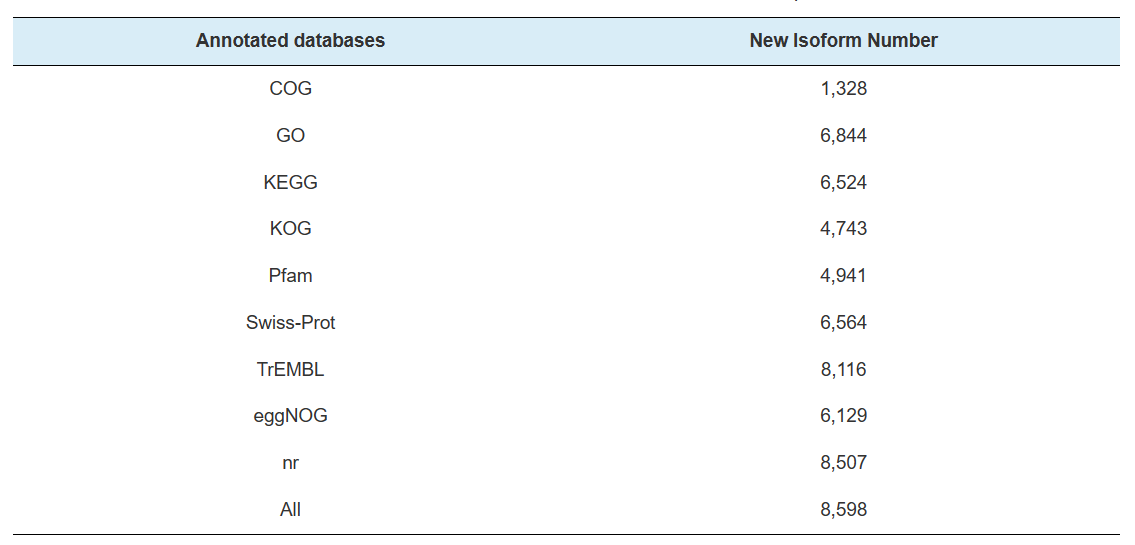
Adnotacja nowych genów
Grupowanie dets
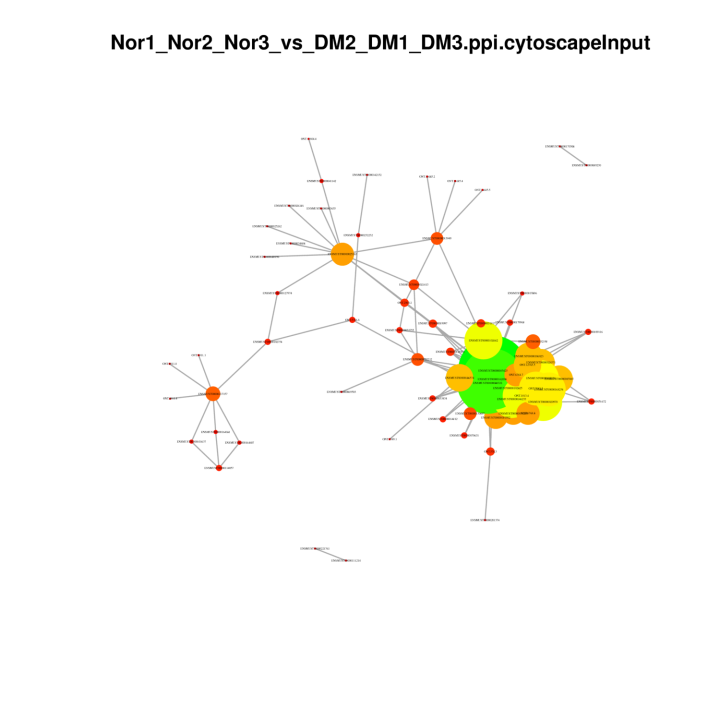
Sieci białko-białko w DEG
Przeglądaj postępy ułatwione przez pełnometrażowe usługi sekwencjonowania mRNA BMKGENE w celu wyselekcjonowanego zbioru publikacji.
Gong, B. i in. (2023) „Aktywacja epigenetyczna i transkrypcyjna kinazy wydzielniczej FAM20C jako onkogenu w glejaku”, Journal of Genetics and Genomics, 50 (6), s. 422–433. doi: 10.1016/j.jgg.2023.01.008.
On, Z. i in. (2023) „Sekwencjonowanie transkryptomu limfocytów w pełnej długości reaguje na IFN-γ, ujawnia odpowiedź immunologiczną z th1 w flądrze (Paralichthys olivaceus)”, Fish & Shellfish Immunology, 134, s. 1. 108636. Doi: 10.1016/j.fsi.2023.108636.
Ma, Y. i in. (2023) „Analiza porównawcza metod sekwencjonowania PACBIO i ONT RNA dla identyfikacji nomurai nomurai nomurai”, Genomics, 115 (6), s. 1. 110709. Doi: 10.1016/j.ygeno.2023.110709.
Yu, D. i in. (2023) „Analiza Nano-Seq ujawnia różną tendencję funkcjonalną między egzosomami i mikroprzepustkami pochodzącymi z HUMSC”, Research and Therapy komórek macierzystych, 14 (1), s. 1–13. doi: 10.1186/s13287-023-03491-5/tabele/6.