Wstępne biblioteki DNBSEQ
Cechy
●Platforma:MGI-DNBSEQ-T7
●Tryby sekwencjonowania:PE150
●Przeniesienie bibliotek Illumina doMGI:Włączanie sekwencjonowania wysokich objętości danych przy niskich kosztach.
●Kontrola jakości bibliotek przed sekwencjonowaniem.
●Sekwencjonowanie danych QC i dostawa:Dostarczanie raportu QC i surowych danych w formacie FASTQ po demultipleksowaniu i filtrowaniu odczytów Q30.
Zalety
●Wszechstronność usług sekwencjonowania:Klient może zdecydować się na sekwencję według linii lub ilości danych.
●Wysokie wyjście danych:1500 GB/pas
●Dostawa sekwencjonowania Raport QC:Dzięki wskaźnikom jakości, dokładności danych i ogólnej wydajności projektu sekwencjonowania.
●Proces dojrzałego sekwencjonowania:z krótkim czasem skrętu.
●Rygorystyczna kontrola jakości: Wdrażamy ścisłe wymagania QC, aby zagwarantować dostarczanie konsekwentnie wysokiej jakości wyników.
Wymagania przykładowe
| Kwota danych (x) | Stężenie (QPCR/nm) | Tom | |
| Częściowy pas
| X ≤ 10 GB | ≥ 1 nm | ≥ 25 μl |
| 10 GB <x ≤ 50 GB | ≥ 2 nm | ≥ 25 μl | |
| 50 gb <x ≤ 100 gb | ≥ 3 nm | ≥ 25 μl | |
| X> 100 GB | ≥ 4 nm | ||
| Pojedynczy pas | Na pas | ≥ 1,5 nm / pula biblioteki | ≥ 25 μl / pula biblioteki |
Oprócz stężenia i całkowitej ilości wymagany jest również odpowiedni wzór szczytowy.
UWAGA: Sekwencjonowanie pasów bibliotek o niskiej różnorodności wymaga Phix Spike-In, aby zapewnić solidne wywołanie podstawowe.
Zalecamy przesłanie bibliotek wstępnie nabranych jako próbek. Jeśli potrzebujesz BMKGENE do wykonania puli bibliotek, zapoznaj się z
Wymagania biblioteczne dla częściowego sekwencjonowania pasów.
Rozmiar biblioteki (mapa szczytowa)
Główny pik powinien znajdować się w granicach 300-450 pz.
Biblioteki powinny mieć jeden główny pik, brak zanieczyszczenia adaptera i bez dimerów starterów.
Przepływ pracy serwisowej




Biblioteka Raport QC
Raport na temat jakości biblioteki przedstawiono przed sekwencjonowaniem, oceną kwoty biblioteki i fragmentacji.
Sekwencjonowanie raportu QC
Tabela 1. Statystyki dotyczące sekwencjonowania danych.
| Próbka id | Bmkid | Raw odczyty | Surowe dane (BP) | Czyste odczyty (%) | Q20 (%) | P30 (%) | GC (%) |
| C_01 | BMK_01 | 22 870,120 | 6 861 036 000 | 96.48 | 99.14 | 94,85 | 36.67 |
| C_02 | BMK_02 | 14 717 867 | 4 415 360 100 | 96.00 | 98,95 | 93,89 | 37,08 |
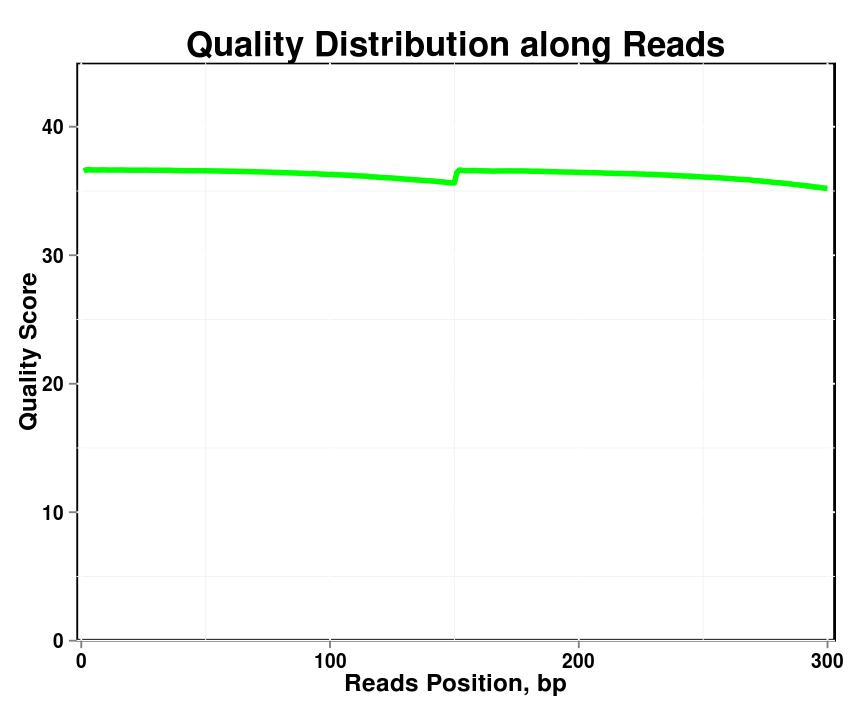
Rysunek 1. Rozkład jakości wzdłuż odczytów w każdej próbce
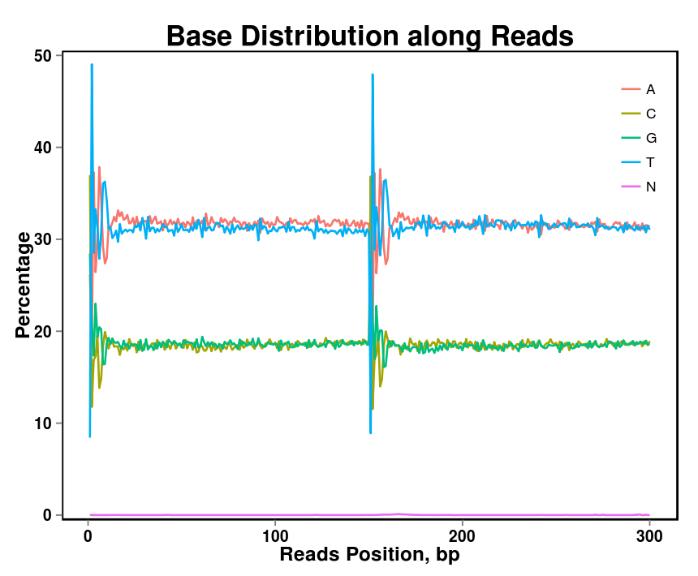
Rysunek 2. Podstawowy rozkład treści
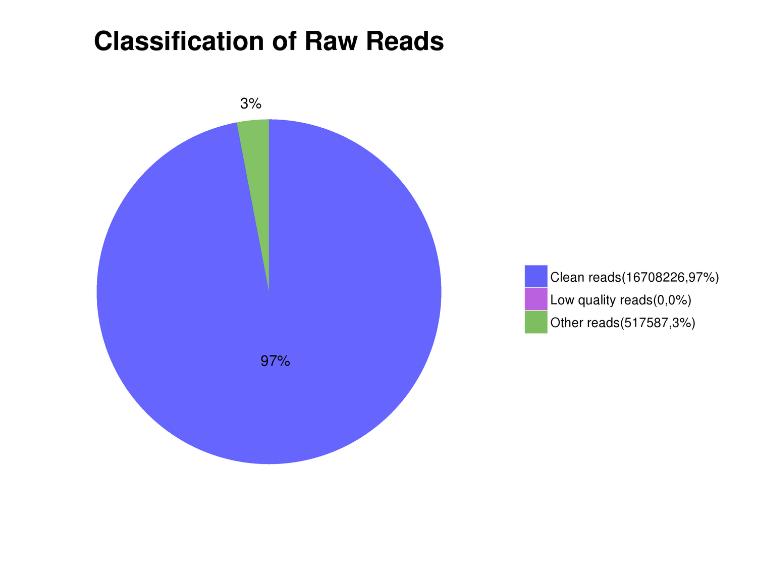
Rysunek 3. Rozkład zawartości odczytu w danych sekwencjonowania