Transkryptom przestrzenny BMKMANU S1000
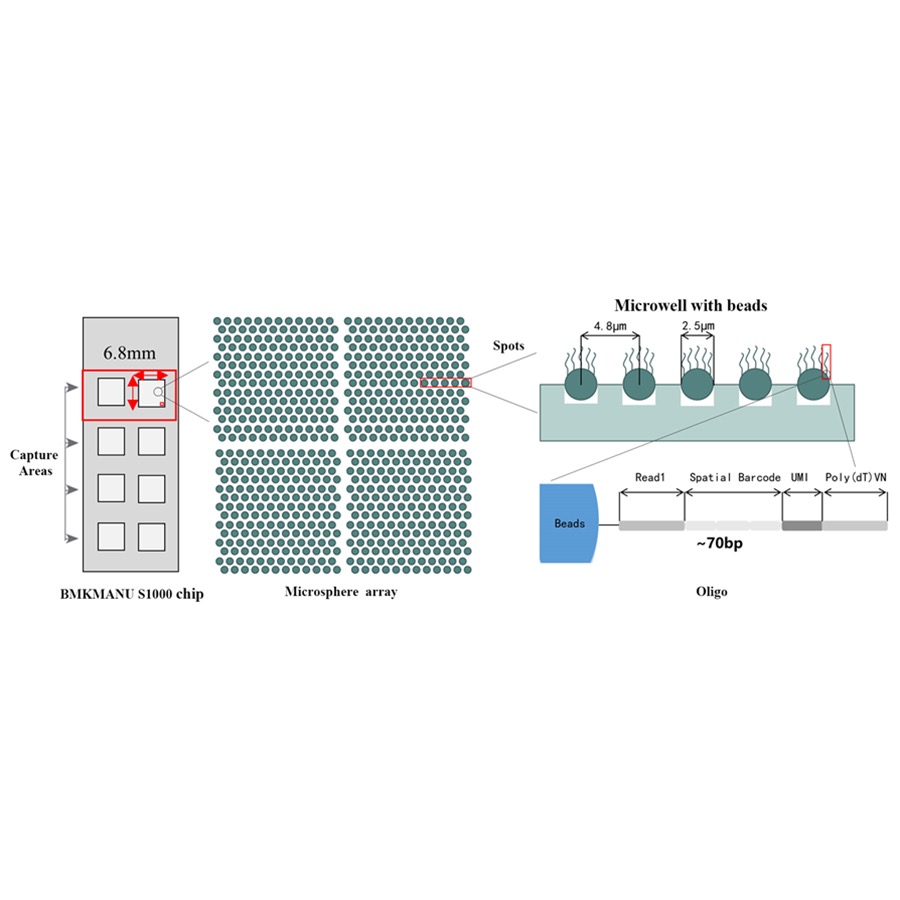
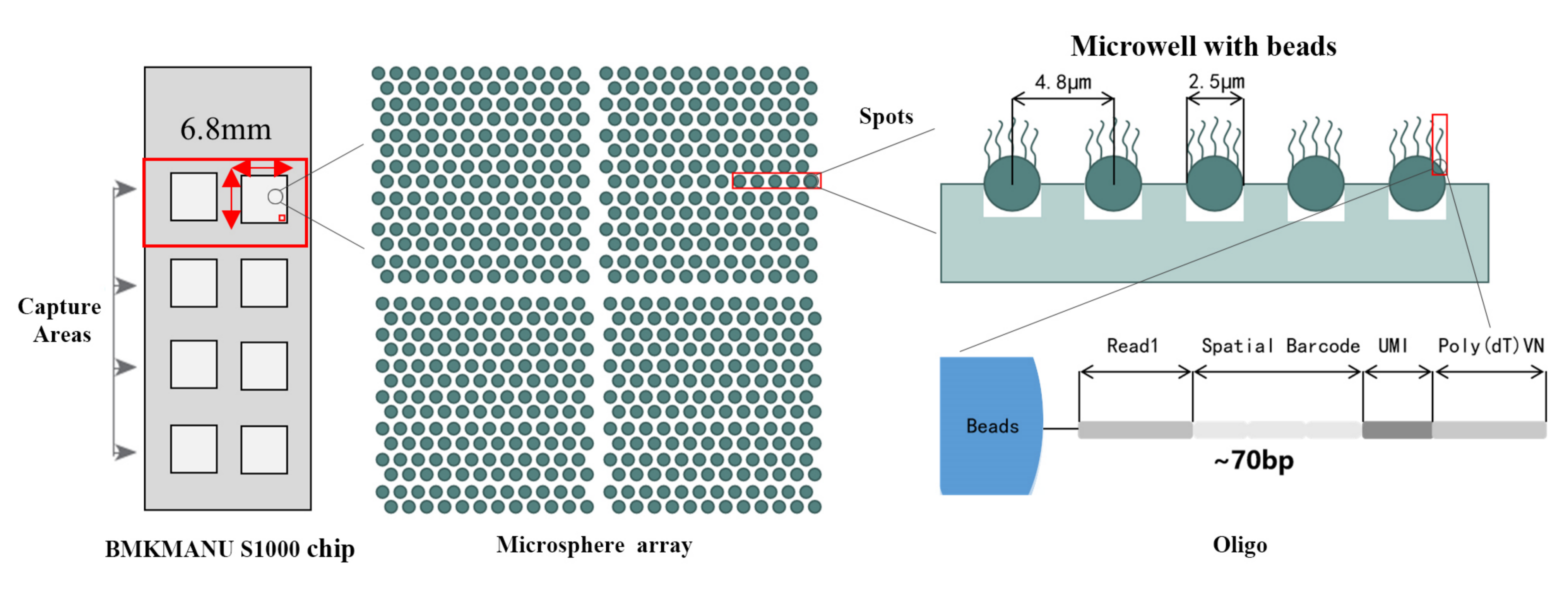
Schemat techniczny transkryptomu przestrzennego BMKMANU S1000
Cechy
● Rozdzielczość: 5 µM
● Średnica plamki: 2,5 µM
● Liczba spotów: około 2 miliony
● 3 możliwe formaty obszaru przechwytywania: 6,8 mm * 6,8 mm, 11 mm * 11 mm lub 15 mm * 20 mm
● Każda kulka z kodem kreskowym zawiera startery składające się z 4 sekcji:
ogon poli(dT) do starterów mRNA i syntezy cDNA
Unikalny identyfikator molekularny (UMI) korygujący błąd wzmocnienia
Przestrzenny kod kreskowy
Sekwencja wiązania startera sekwencjonującego częściowy odczyt 1
● Barwienie H&E i fluorescencyjne skrawków
● Możliwość wykorzystaniatechnologia segmentacji komórek: integracja barwienia H&E, barwienia fluorescencyjnego i sekwencjonowania RNA w celu określenia granic każdej komórki i prawidłowego przypisania ekspresji genów do każdej komórki.
Zalety BMKMANU S1000
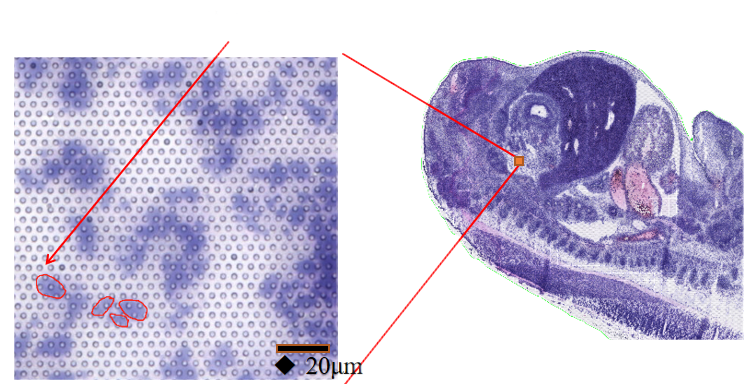
●Rozdzielczość subkomórkowa: Każdy obszar przechwytywania zawierał >2 miliony przestrzennych plamek z kodem kreskowym o średnicy 2,5 µm i odstępach między środkami plamek 5 µm, co umożliwiło przestrzenną analizę transkryptomu z rozdzielczością subkomórkową (5 µm).
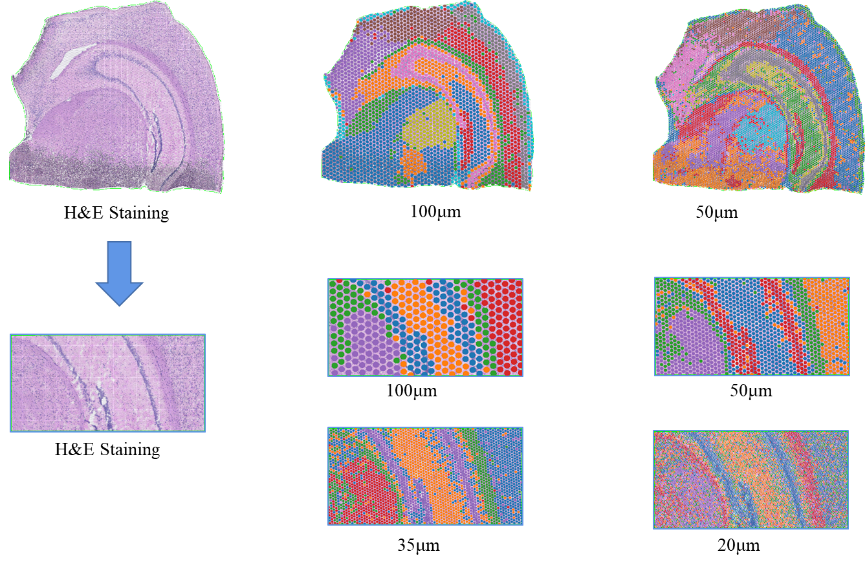
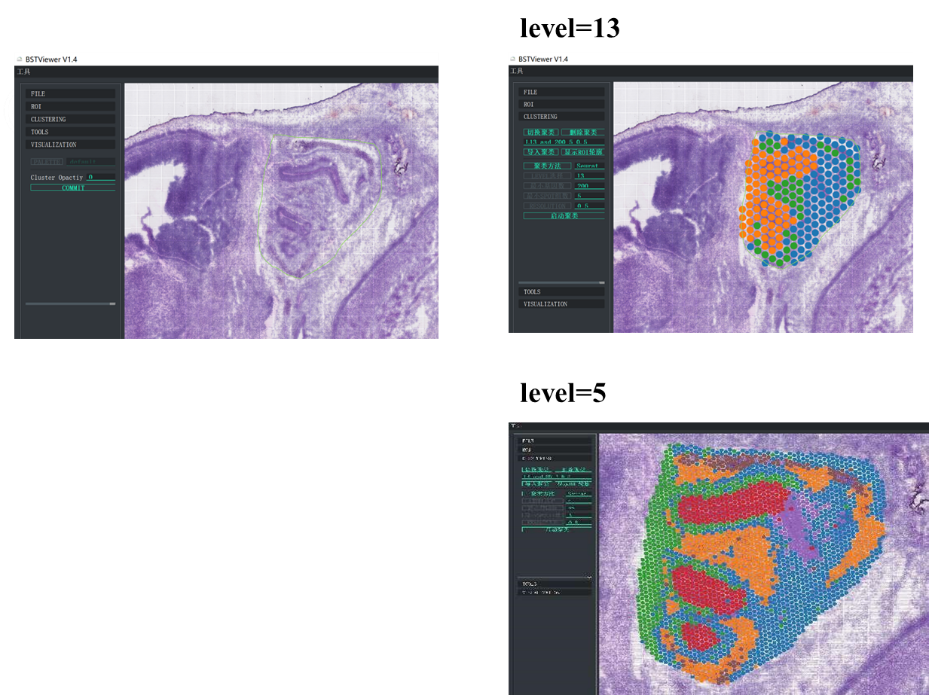
●Wielopoziomowa analiza rozdzielczości:Elastyczna analiza wielopoziomowa w zakresie od 100 μm do 5 μm w celu określenia różnych cech tkanek przy optymalnej rozdzielczości.
●Możliwość zastosowania technologii segmentacji komórek „Trzy w jednym slajdzie”:Łącząc barwienie fluorescencyjne, barwienie H&E i sekwencjonowanie RNA na jednym szkiełku, nasz algorytm analizy „trzy w jednym” umożliwia identyfikację granic komórek na potrzeby późniejszej transkryptomiki opartej na komórkach.
●Kompatybilny z wieloma platformami sekwencjonowania: Dostępne jest zarówno sekwencjonowanie NGS, jak i sekwencjonowanie z długim odczytem.
●Elastyczny projekt 1-8 aktywnych obszarów przechwytywania: Rozmiar obszaru przechwytywania jest elastyczny i można zastosować 3 formaty (6,8 mm * 6,8 mm, 11 mm * 11 mm i 15 mm * 20 mm)
●Usługa w jednym miejscu: Integruje wszystkie etapy oparte na doświadczeniu i umiejętnościach, w tym kriosekcje, barwienie, optymalizację tkanek, przestrzenne kodowanie kreskowe, przygotowanie bibliotek, sekwencjonowanie i bioinformatykę.
●Kompleksowa bioinformatyka i przyjazna dla użytkownika wizualizacja wyników:pakiet zawiera 29 analiz i ponad 100 wysokiej jakości danych liczbowych, w połączeniu z wykorzystaniem opracowanego przez nas oprogramowania do wizualizacji i dostosowywania podziału komórek i grupowania punktów.
●Indywidualna analiza i wizualizacja danych: dostępne dla różnych zleceń badawczych
●Wysoko wykwalifikowany zespół techniczny: z doświadczeniem w ponad 250 typach tkanek i ponad 100 gatunkach, w tym ludziach, myszach, ssakach, rybach i roślinach.
●Aktualizacje całego projektu w czasie rzeczywistym: z pełną kontrolą postępu eksperymentu.
●Opcjonalna analiza łączona z sekwencjonowaniem mRNA pojedynczych komórek
Specyfikacje usług
|
Próbka Wymagania
| Biblioteka |
Strategia sekwencjonowania
| Zalecane dane | Kontrola jakości |
| Próbki kriogeniczne z osadzeniem OCT, 3 bloki na próbkę | Biblioteka cDNA S1000 | Illumina PE150 (dostępne inne platformy) | 100 tys. odczytów PE na 100 uM ( 60-150 GB ) | RIN>7 |
Aby uzyskać więcej informacji na temat wskazówek dotyczących przygotowywania próbek i przebiegu usług, skontaktuj się z: aEkspert BMKGENE
Przebieg prac serwisowych
Na etapie przygotowania próbki przeprowadzana jest wstępna próba ekstrakcji masowego RNA, aby zapewnić uzyskanie wysokiej jakości RNA. Na etapie optymalizacji tkanki skrawki są barwione i wizualizowane, a warunki przepuszczalności dla uwalniania mRNA z tkanki są optymalizowane. Zoptymalizowany protokół jest następnie stosowany podczas konstruowania biblioteki, po czym następuje sekwencjonowanie i analiza danych.
Kompletny przepływ usług obejmuje aktualizacje w czasie rzeczywistym i potwierdzenia od klientów, co pozwala na utrzymanie responsywnej pętli informacji zwrotnej, zapewniającej płynną realizację projektu.
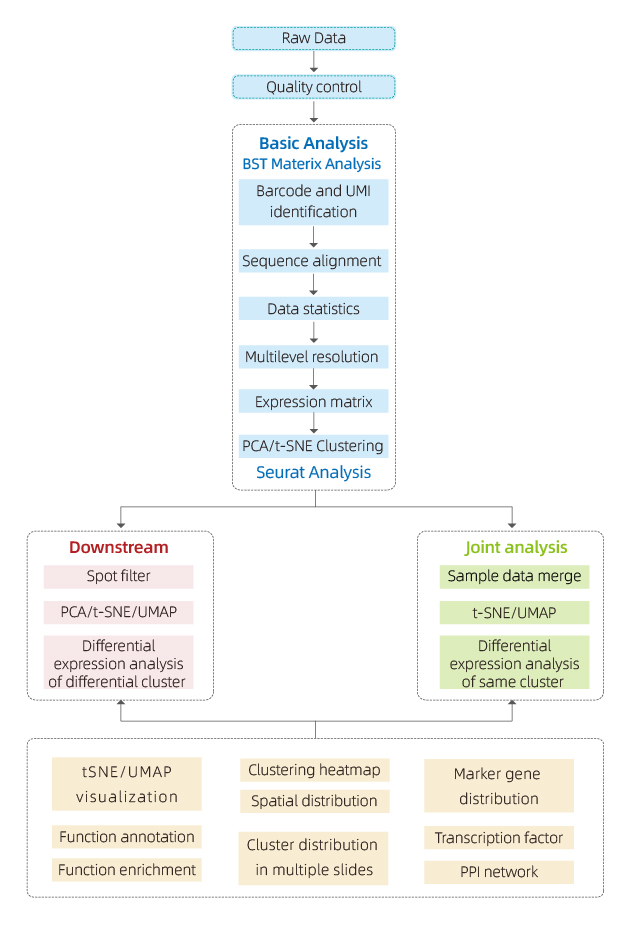
Dane generowane przez BMKMANU S1000 są analizowane przy użyciu oprogramowania „BSTMatrix”, które zostało niezależnie zaprojektowane przez BMKGENE, generując matrycę ekspresji genów. Na tej podstawie generowany jest standardowy raport obejmujący kontrolę jakości danych, analizę próbki wewnętrznej i analizę międzygrupową.
● Kontrola jakości danych:
Dane wyjściowe i dystrybucja wyników jakości
Wykrywanie genów na plamkę
Pokrycie tkanki
● Analiza próbki wewnętrznej:
Bogactwo genów
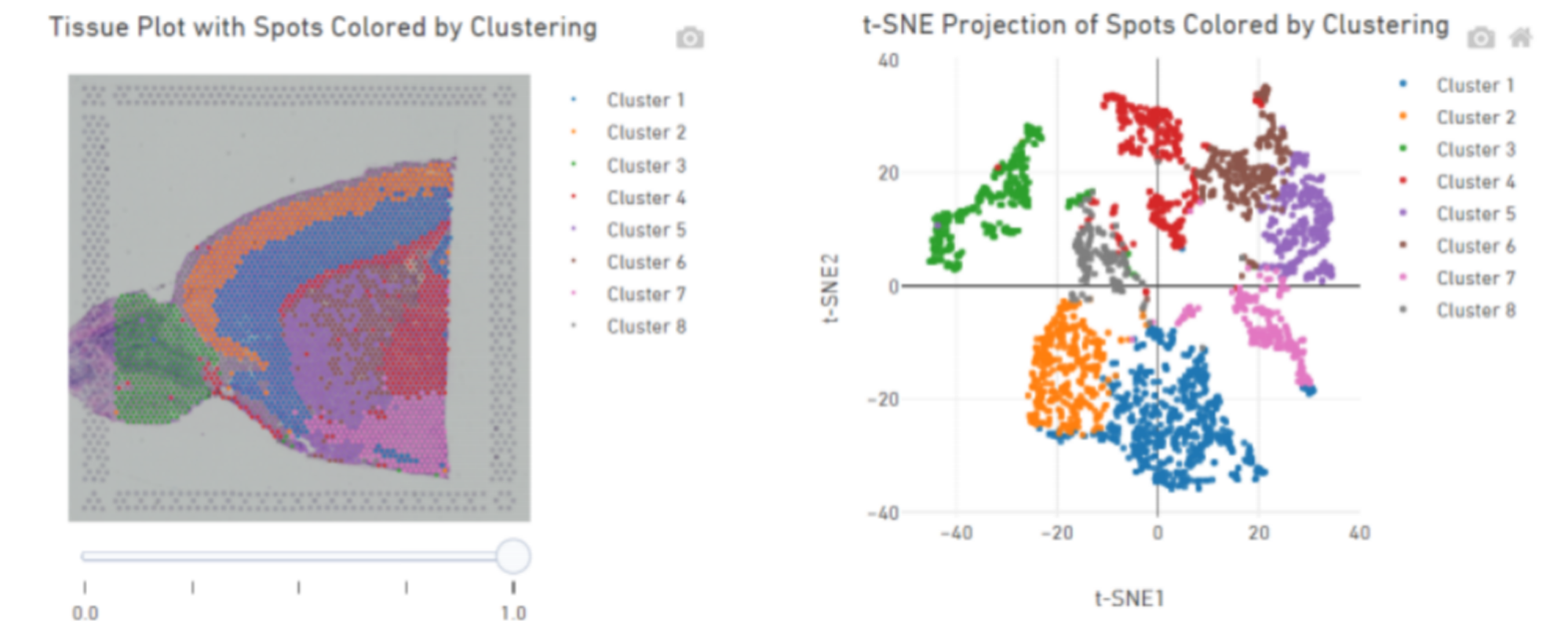
Grupowanie punktów, w tym analiza zredukowanych wymiarów
Różnicowa analiza ekspresji pomiędzy klastrami: identyfikacja genów markerowych
Adnotacja funkcjonalna i wzbogacanie genów markerowych
● Analiza międzygrupowa:
Ponowne połączenie plam z obu próbek (np. chorych i kontrolnych) i ponowne skupienie
Identyfikacja genów markerowych dla każdego klastra
Adnotacja funkcjonalna i wzbogacanie genów markerowych
Wyrażenie różniczkowe tego samego skupienia między grupami
Dodatkowo w ramach projektu BMKGENE opracowano „BSTViewer” to przyjazne dla użytkownika narzędzie, które umożliwia użytkownikowi wizualizację ekspresji genów i grupowania plamek w różnych rozdzielczościach.
BMKGene opracowało oprogramowanie do wizualizacji przyjaznej dla użytkownika
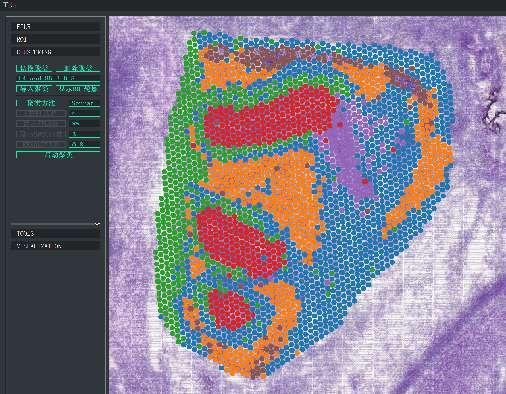
Klastrowanie punktów BSTViewer w rozdzielczości wielopoziomowej
BSTCellViewer: automatyczne i ręczne dzielenie komórek
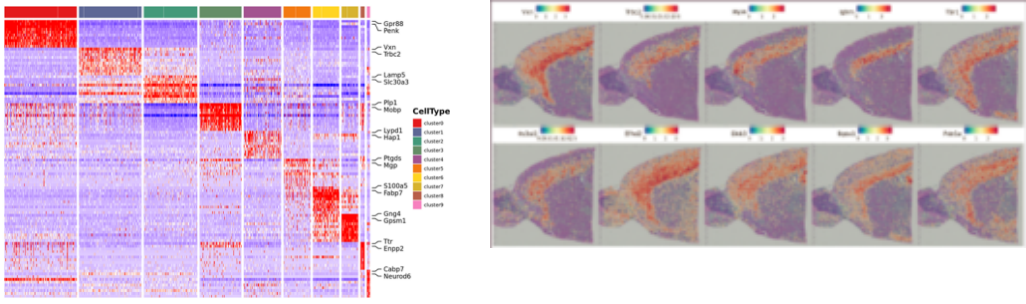
Analiza próbki wewnętrznej
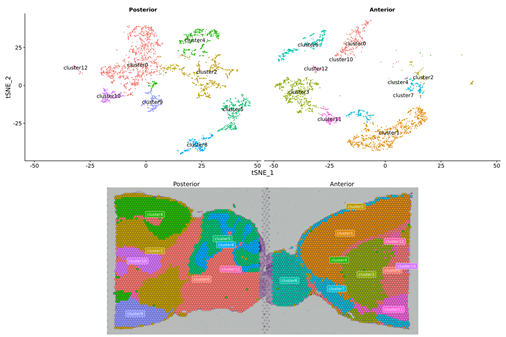
Grupowanie punktowe:
Identyfikacja i rozkład przestrzenny genów markerowych:
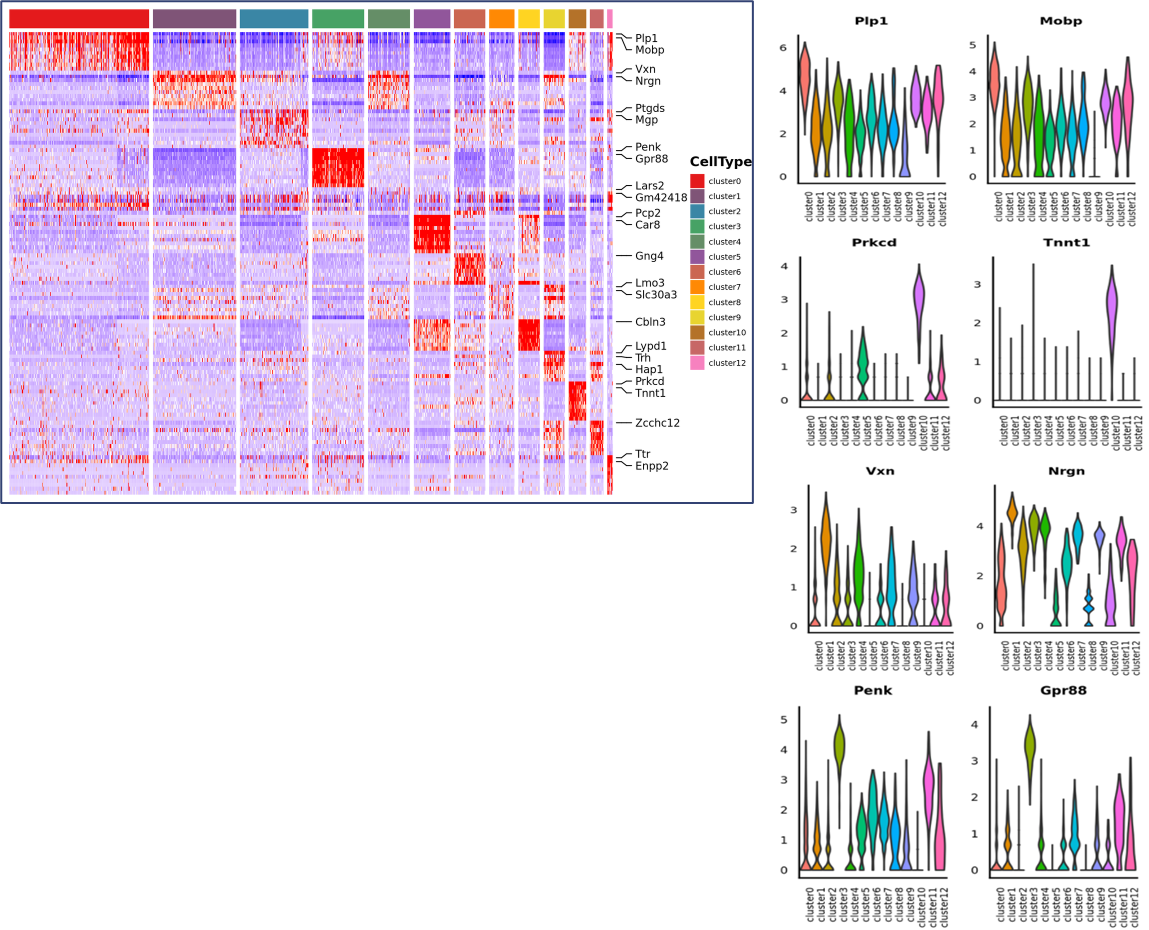
Analiza międzygrupowa
Kombinacja danych z obu grup i ponownego skupienia:
Geny markerowe nowych klastrów:
Zapoznaj się z postępami, jakie umożliwiają usługi transkryptomiki przestrzennej BMKGene z technologią BMKManu S1000 w tej wyróżnionej publikacji:
Song, X. i in. (2023) „Transkryptomika przestrzenna ujawnia indukowane światłem komórki chlorenchymy zaangażowane w promowanie regeneracji pędów kalusa pomidora”,Proceedings of National Academy of Sciences of the United States of America, 120(38), s. 1. e2310163120. doi: 10.1073/pnas.2310163120
Ty, Y. i in. (2023) „Systematyczne porównanie metod transkryptomii przestrzennej opartej na sekwencjonowaniu”,bioRxiv, P. 2023.12.03.569744. doi: 10.1101/2023.12.03.569744.