Sekwencjonowanie amplikonu 16S/18S/ITS – PacBio
Funkcje usługi
● Platforma sekwencjonowania: PacBio Revio
● Tryb sekwencjonowania: CCS (odczyt HiFi)
● Amplifikacja regionu docelowego, a następnie tandemowe łączenie amplikonów przed przygotowaniem biblioteki dzwonków HiFi SMRT
Zalety serwisu
●Wyższa rozdzielczość taksonomiczna: Tsekwencjonowanie krótkiego amplikonu Hana,umożliwiając wyższe wskaźniki klasyfikacji OTU na poziomie gatunku.
●Bardzo dokładne wywołanie podstawowe: Sekwencjonowanie trybu PacBio CCS (odczyt HiFi).
●Bez izolacji: Szybka identyfikacja składu mikrobiologicznego w próbkach środowiskowych.
●Powszechnie stosowane: Różnorodne badania społeczności drobnoustrojów.
●Kompleksowa analiza bioinformatyczna: Najnowszy pakiet QIIME2 (ilościowy wgląd w ekologię drobnoustrojów) z różnorodnymi analizami pod kątem bazy danych, adnotacji, OTU/ASV.
●Rozległa wiedza specjalistyczna: Dzięki tysiącom projektów sekwencjonowania amplikonów prowadzonych co roku, BMKGENE wnosi ponad dziesięcioletnie doświadczenie, wysoko wykwalifikowany zespół analityczny, obszerną treść i doskonałe wsparcie posprzedażowe.
Specyfikacje usług
| Biblioteka | Strategia sekwencjonowania | Zalecane dane |
| Amplikon | PacBio Revio | Tagi 10/30/50 tys. (CCS) |
Przykładowe wymagania
| Stężenie (ng/µL) | Całkowita ilość (µg) | Objętość (µL) |
| ≥5 | ≥0,3 | ≥20 |
Zalecana dostawa próbek
Zamrozić próbki w ciekłym azocie na 3-4 godziny i przechowywać w ciekłym azocie lub w -80 stopniach z rezerwacją długoterminową. Wymagana jest wysyłka próbek z suchym lodem.
Przebieg prac serwisowych

Dostawa próbek

Budowa biblioteki

Sekwencjonowanie

Analiza danych

Usługi posprzedażowe
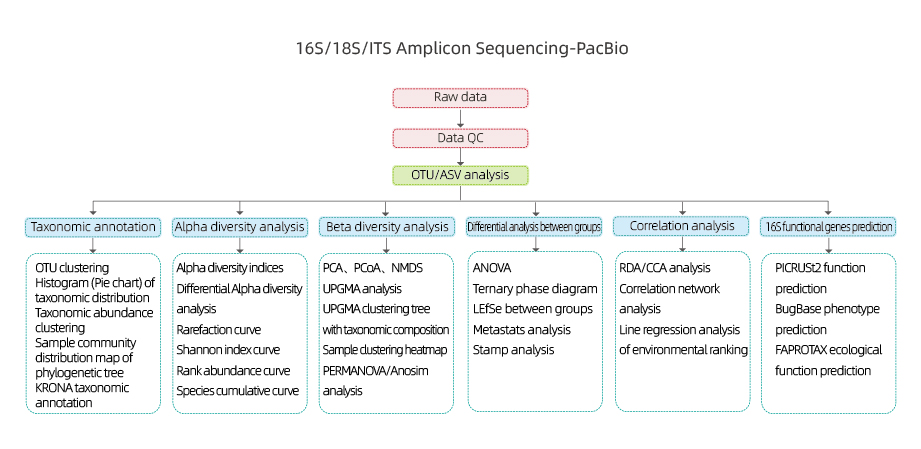
Zawiera następującą analizę:
●Kontrola jakości surowych danych
●Klustrowanie/odszumianie OTU (ASV)
●Adnotacja OTU
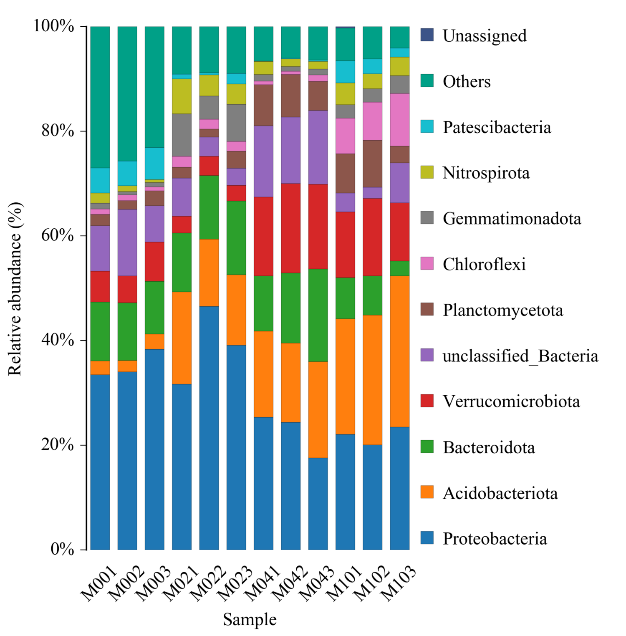
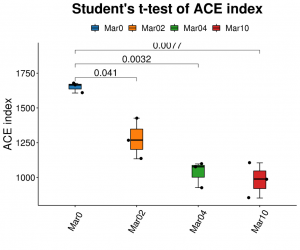
●Analiza różnorodności Alpha: wiele indeksów, w tym Shannon, Simpson i ACE.
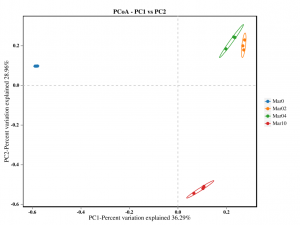
●Analiza różnorodności Beta
●Analiza międzygrupowa
●Analiza korelacji: pomiędzy czynnikami środowiskowymi a składem i różnorodnością OUT
● Przewidywanie genu funkcjonalnego 16S
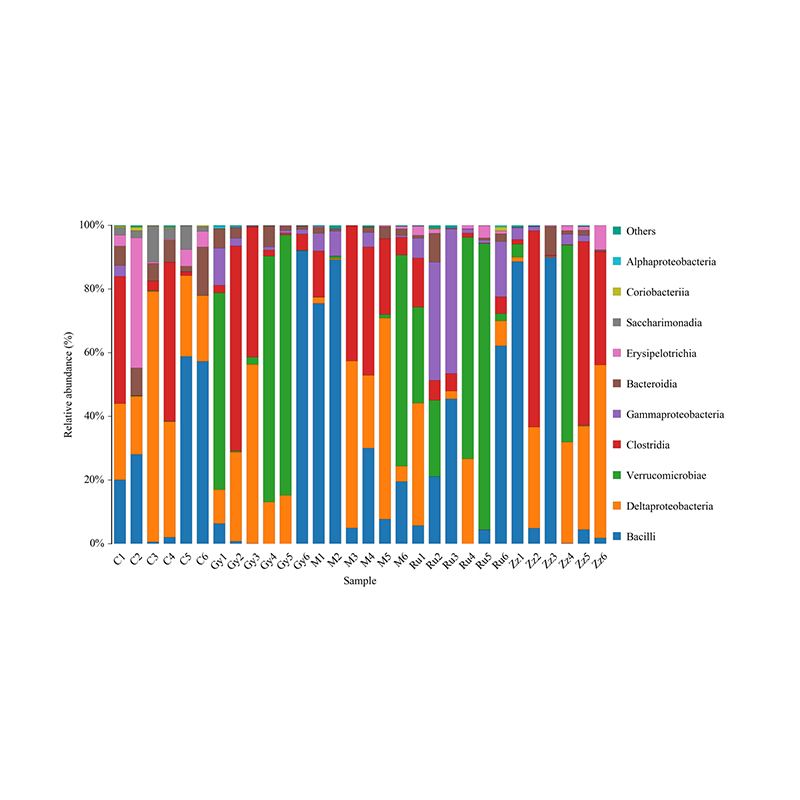
Histogram rozkładu taksonomicznego
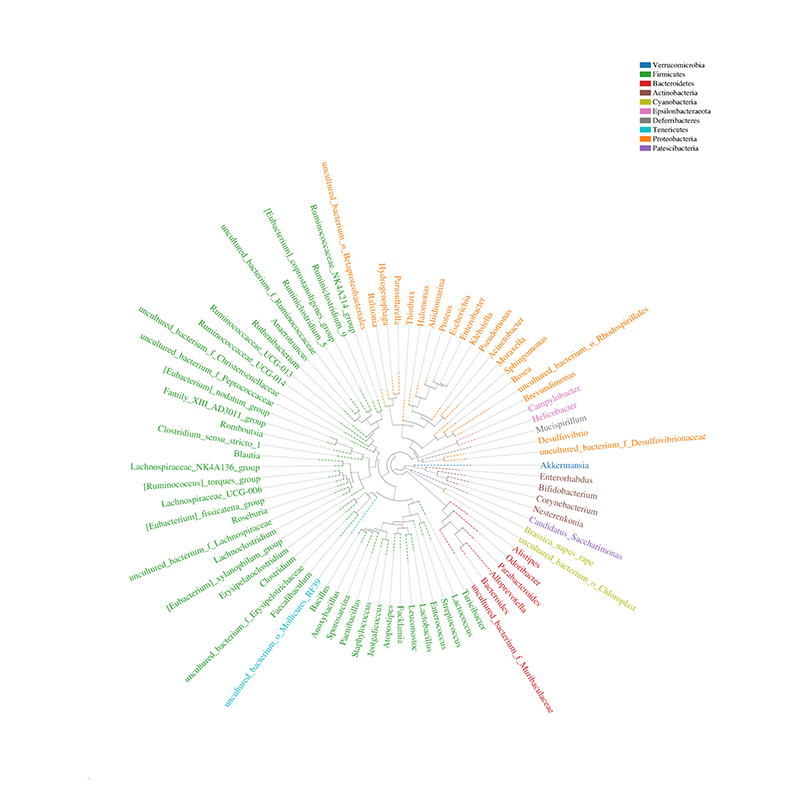
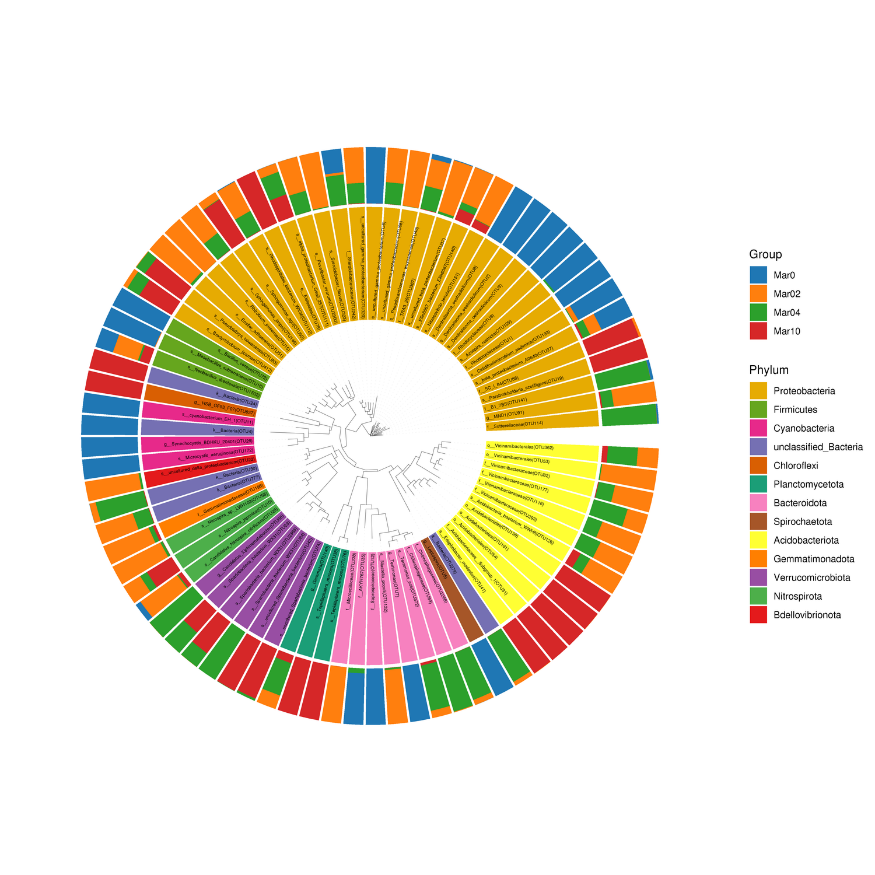
Drzewo filogenetyczne dystrybucji społeczności
Analiza różnorodności alfa: ACE
Analiza różnorodności beta: PCoA
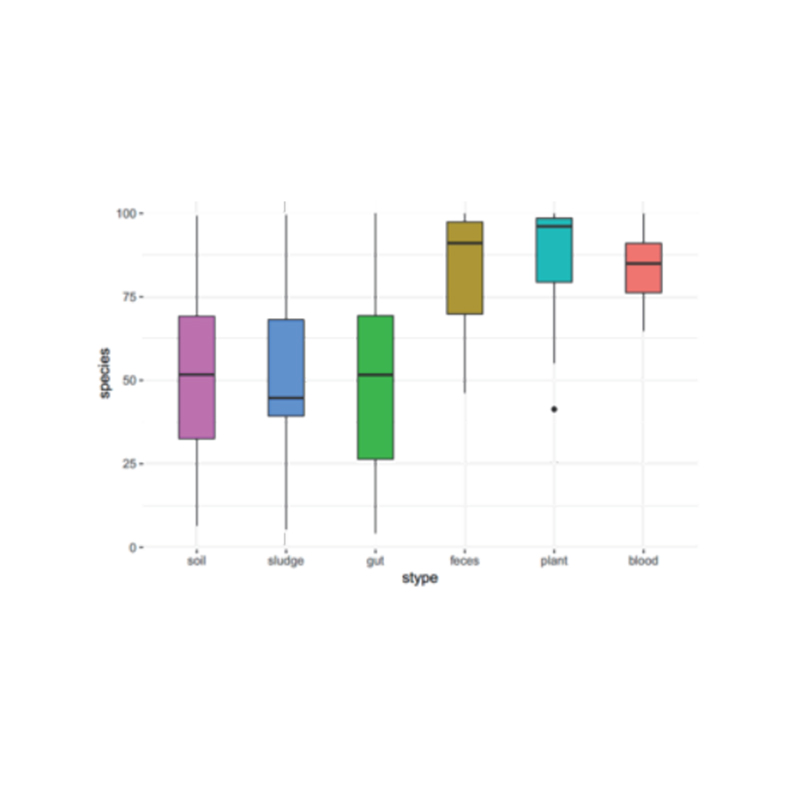
Analiza międzygrupowa: ANOVA
Odkryj postępy, jakie zapewniają usługi sekwencjonowania amplikonów BMKGene za pomocą PacBio, korzystając z wyselekcjonowanej kolekcji publikacji.
Gao, X. i Wang, H. (2023) „Analiza porównawcza profili bakteryjnych i funkcji żwacza podczas adaptacji do różnych fenologii (rezielone vs. trawiaste) u alpejskich merynosów z dwoma etapami wzrostu na alpejskiej łące”, Fermentacja, 9( 1), s. 1 16. doi: 10.3390/FERMENTACJA9010016/S1.
Li, S. i in. (2023) „Przechwytywanie ciemnej materii drobnoustrojów w glebach pustynnych za pomocą metagenomiki opartej na kulturomice i analizy o wysokiej rozdzielczości”, npj Biofilms and Microbiomes 2023 9:1, 9(1), s. 1–14. doi: 10.1038/s41522-023-00439-8.
Mu, L. i in. (2022) „Wpływ soli kwasów tłuszczowych na charakterystykę fermentacji, różnorodność bakteryjną i stabilność tlenową mieszanej kiszonki przygotowanej z lucerny, słomy ryżowej i otrębów pszennych”, Journal of the Science of Food and Agriculture, 102(4), s. 1475– 1487. doi: 10.1002/JSFA.11482.
Yang, J. i in. (2023) „Interakcja między biomarkerami stresu oksydacyjnego a mikroflorą jelitową w działaniu przeciwutleniającym ekstraktów z Sonchus brachyotus DC”. w: Stres oksydacyjny jelit wywołany oksazolonem u dorosłych danio pręgowanych, Przeciwutleniacze 2023, tom. 12, s. 192, 12 ust. 1, s. 12. 192. doi: 10.3390/ANTIOX12010192.