Sekwencjonowanie amplikonu 16S/18S/ITS-NGS
Funkcje usługi
● Platforma sekwencjonowania: Illumina NovaSeq.
● Amplifikacja krótkich regionów 16S, 18S i ITS, wśród innych celów amplifikacji.
● Elastyczny wybór amplikonu.
● Doświadczenia z poprzednich projektów z wieloma celami amplifikacji.
Zalety serwisu
●Bez izolacji:Szybka identyfikacja składu mikrobiologicznego w próbkach środowiskowych.
●Wysoka rozdzielczość: W słabo występujących składnikach w próbkach środowiskowych.
●Szeroko stosowane: Różnorodne badania społeczności drobnoustrojów.
●Kompleksowa analiza bioinformatyczna: Najnowszy pakiet QIIME2 (ilościowy wgląd w ekologię drobnoustrojów) z różnorodnymi analizami pod kątem bazy danych, adnotacji, OTU/ASV.
●Rozległa wiedza specjalistyczna: Dzięki 150 tysiącom projektów sekwencjonowania amplikonów prowadzonych rocznie, BMKGENE wnosi ponad dziesięcioletnie doświadczenie, wysoko wykwalifikowany zespół analityczny, obszerną treść i doskonałe wsparcie posprzedażowe.
Specyfikacje usług
| Biblioteka | Strategia sekwencjonowania | Zalecane dane |
| Amplikon | Illumina PE250 | Tagi 50/100/300 tys. (odczyt par) |
Wymagania serwisowe
| Stężenie (ng/µL) | Całkowita kwota (ng) | Objętość (µL) |
| ≥1 | ≥200 | ≥20 |
● Gleba/szlam: 1-2g
● Treść jelitowa-zwierzęca: 0,5-2g
● Treść jelitowa-owada: 0,1-0,25g
● Powierzchnia rośliny (wzbogacony osad): 0,1-0,5g
● Osad wzbogacony bulionem fermentacyjnym): 0,1-0,5g
● Odchody (duże zwierzęta): 0,5-2g
● Odchody (myszy): 3-5 ziaren
● Płyn do płukania pęcherzyków płucnych: bibuła filtracyjna
● Wymaz z pochwy: 5-6 wymazów
● Wymaz ze skóry/narządów płciowych/śliny/tkanek miękkich jamy ustnej/wymaz z gardła/wymaz z odbytu: 2-3 wymazy
● Mikroorganizmy powierzchniowe: bibuła filtracyjna
● Zbiornik wodny/powietrze/biofilm: bibuła filtracyjna
● Endofity: 1-2g
● Płytka nazębna: 0,5-1g
Przebieg prac serwisowych

Dostawa próbek

Budowa biblioteki

Sekwencjonowanie

Analiza danych

Usługi posprzedażowe
Zawiera następującą analizę:
- Kontrola jakości surowych danych
- Klastrowanie OTU / usuwanie szumów (ASV)
- Adnotacja OTU
- Analiza różnorodności alfa: wiele indeksów, w tym Shannon, Simpson i ACE.
- Analiza różnorodności beta
- Analiza międzygrupowa
- Analiza korelacji: pomiędzy czynnikami środowiskowymi a składem i różnorodnością OUT
- Przewidywanie genu funkcjonalnego 16S
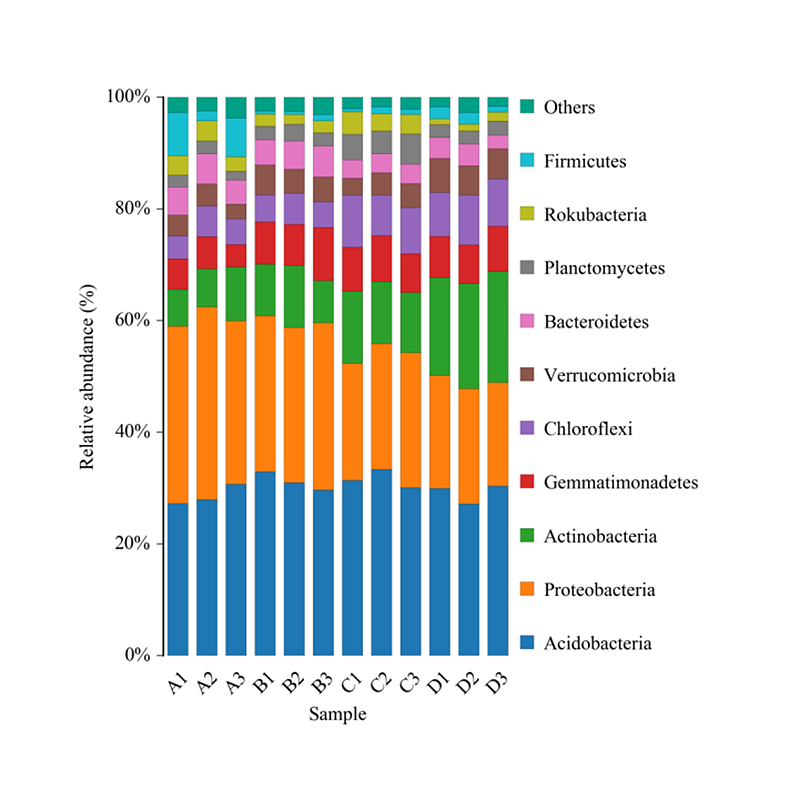
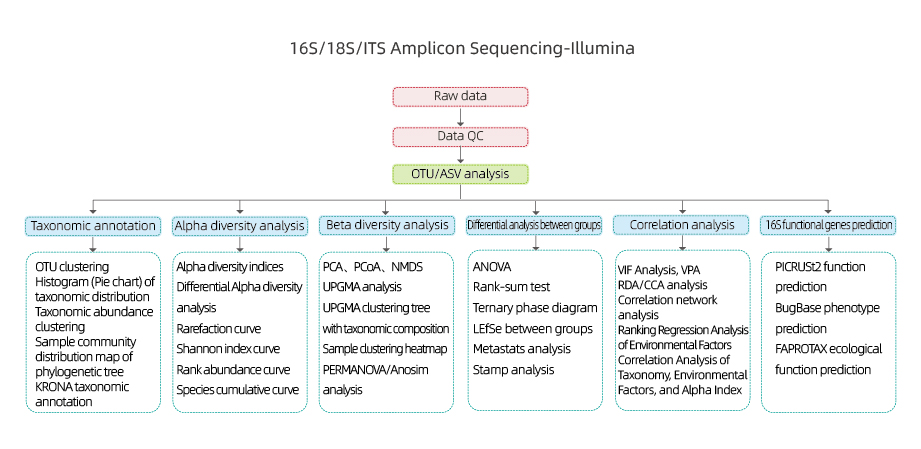
Histogram rozkładu taksonomicznego
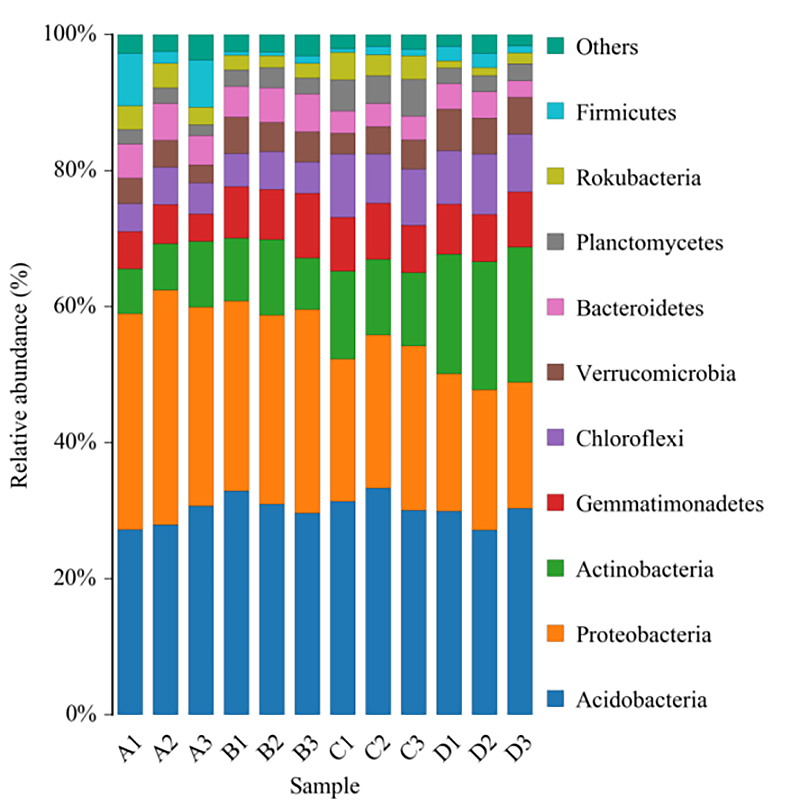
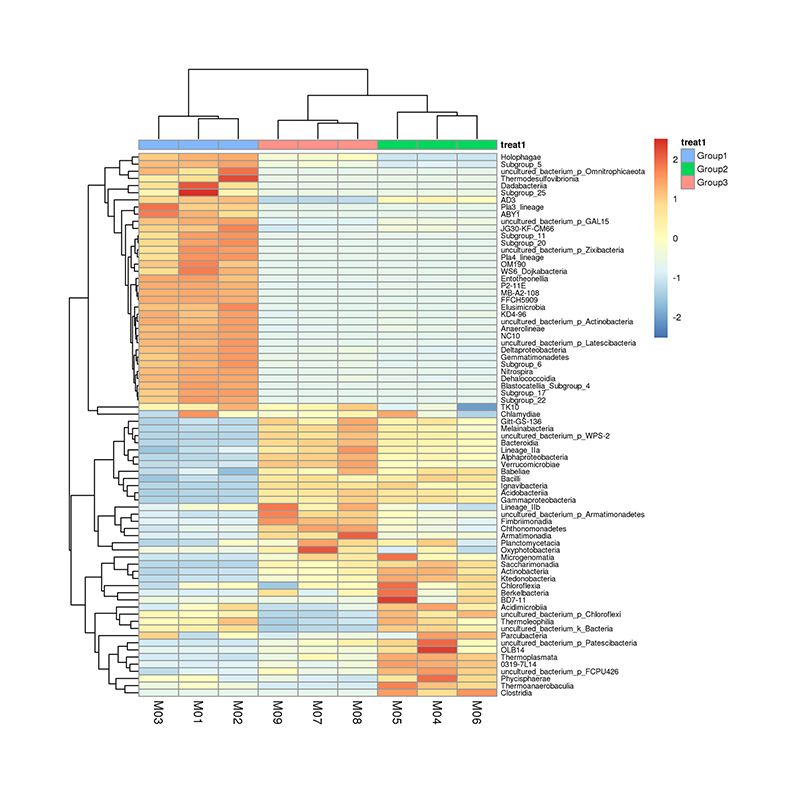
Mapa ciepła grupująca liczebność taksonomiczną
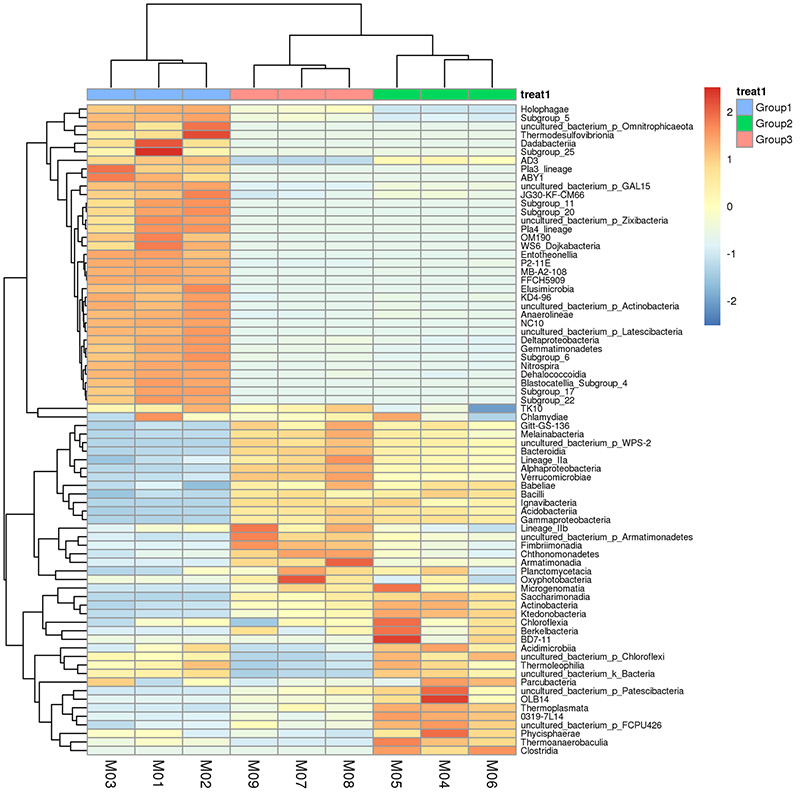
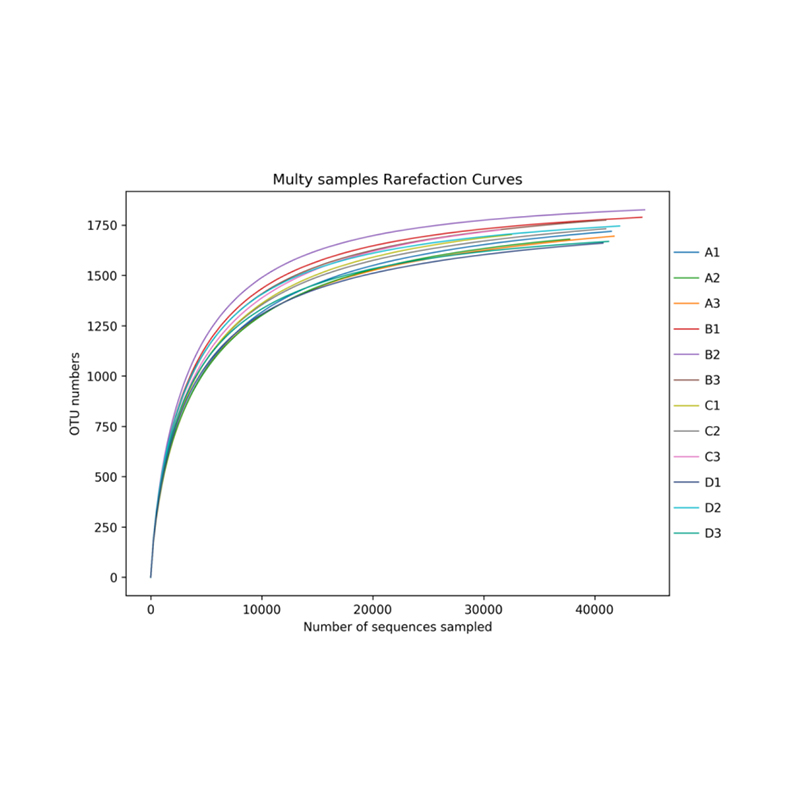
Analiza różnorodności alfa: krzywa rozrzedzenia
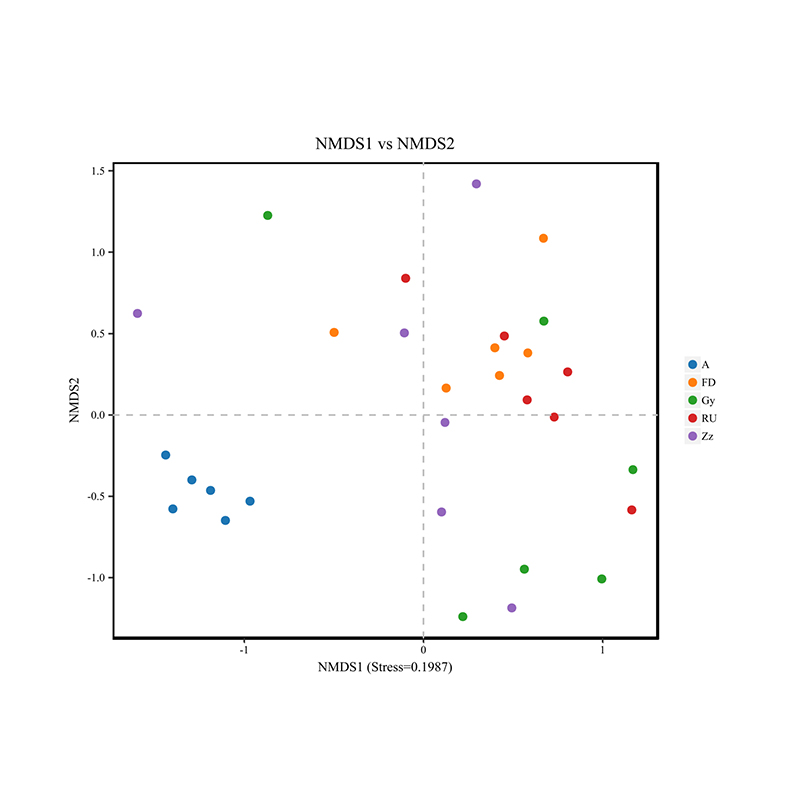
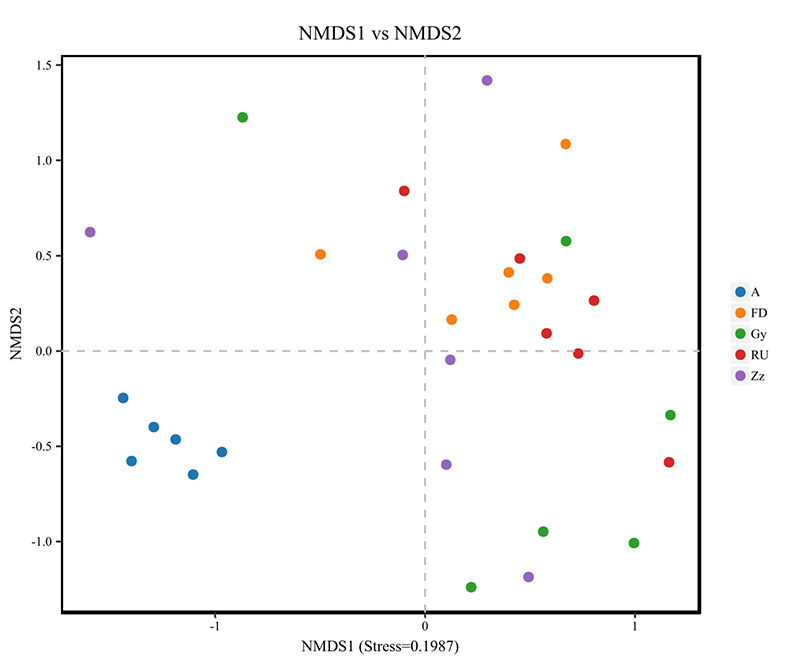
analiza różnorodności beta: NMDS
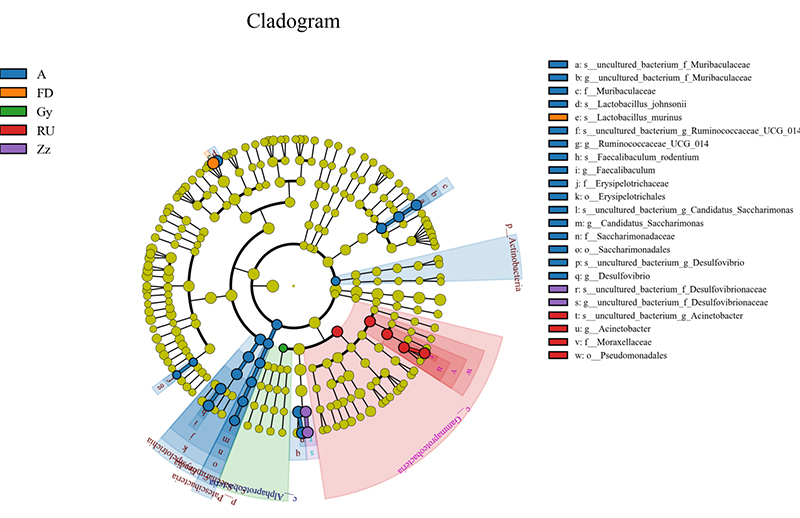
Analiza międzygrupowa: odkrycie biomarkera LEFSE
Zapoznaj się z postępami, jakie umożliwiają usługi sekwencjonowania amplikonów BMKGene za pomocą firmy Illumina, poprzez wyselekcjonowany zbiór publikacji.
Dong, C. i in. (2022) „Montaż, rdzeń mikroflory i funkcja ryzosfery gleby i mikroflory kory u Eucommia ulmoides”, Frontiers in Microbiology, 13. doi: 10.3389/FMICB.2022.855317/FULL.
Li, Y. i in. (2023) „Syntetyczny przeszczep konsorcjów bakteryjnych w leczeniu bakteryjnego zapalenia pochwy wywołanego przez Gardnerella pochwy u myszy”, Microbiome, 11(1), s. 1–14. doi: 10.1186/s40168-023-01497-y
Yang, J., Fu, Y. i Liu, H. (2022) „Mikrobiomy pyłu powietrznego zebrane podczas naziemnego eksperymentu z zamkniętym bioregeneracyjnym podtrzymywaniem życia „Lunar Palace 365””, Environmental Microbiomes, 17(1), s. 1–20. doi: 10.1186/S40793-022-00399-0/RYSUNKI/8.
Yin, S. i in. (2022) „Zależna od surowca obfitość funkcjonalnych genów związanych z transformacją azotu kontroluje utratę azotu podczas kompostowania”, Bioresource Technology, 361, s. 127678. doi: 10.1016/J.BIORTECH.2022.127678.