10x genomika wizja przestrzenna transkryptom
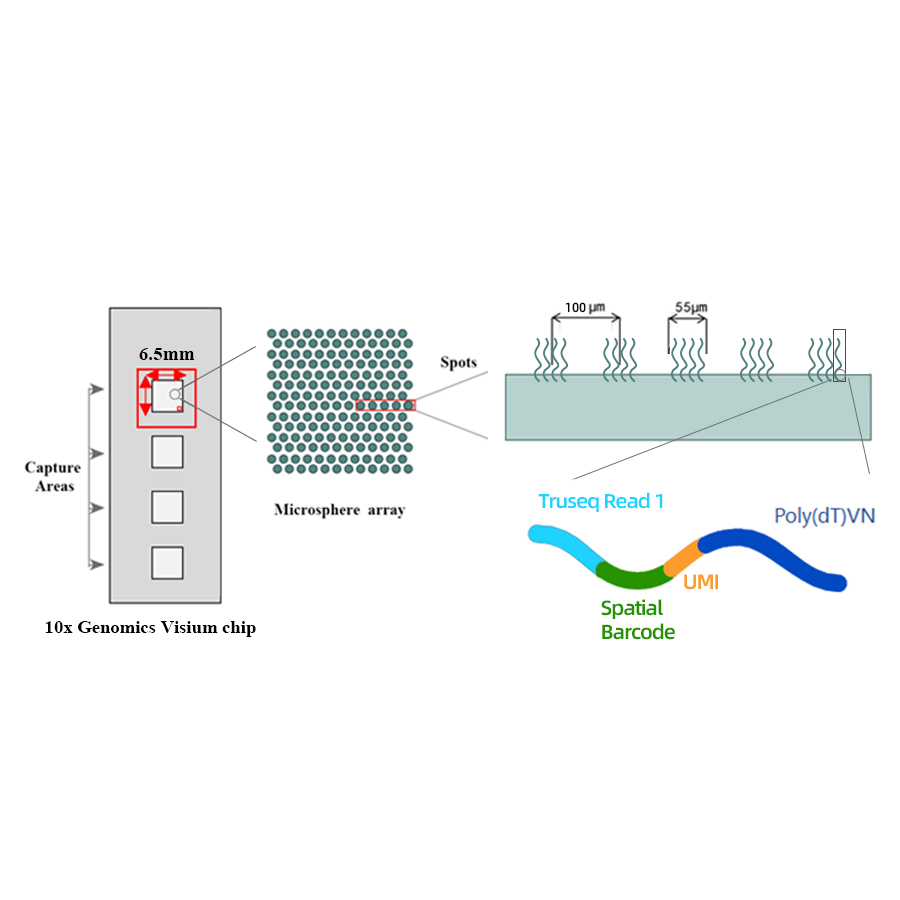
Schemat techniczny

Cechy
● Rozdzielczość: 100 µm
● Średnica punktowa: 55 µm
● Liczba miejsc: 4992
● Obszar przechwytywania: 6,5 x 6,5 mm
● Każde kod kodowania jest załadowany starterami złożonymi z 4 sekcji:
- Poly (DT) ogon do gruntowania mRNA i syntezy cDNA
- Unikalny identyfikator molekularny (UMI) w celu skorygowania stronniczości wzmocnienia
- Kod kreskowy przestrzenny
- Sekwencja wiązania częściowego startera sekwencjonowania odczytu 1
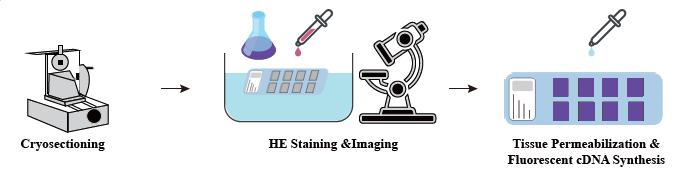
● Barwienie H&E sekcji
Zalety
●Usługa jednokrotna: Integruje wszystkie doświadczenia i kroki oparte na umiejętnościach, w tym sekcje krio, barwienie, optymalizację tkanek, kod kreskowy przestrzenny, przygotowanie biblioteki, sekwencjonowanie i bioinformatyka.
● Wysoko wykwalifikowany zespół techniczny: z doświadczeniem w ponad 250 typach tkanek i ponad 100 gatunkach, w tym ludzkich, myszy, ssakach, rybach i roślinach.
●Aktualizacja w czasie rzeczywistym na temat całego projektu: z pełną kontrolą postępu eksperymentalnego.
●Kompleksowa standardowa bioinformatyka:Pakiet obejmuje 29 analiz i ponad 100 wysokiej jakości liczb.
●Dostosowywana analiza danych i wizualizacja: Dostępne dla różnych żądań badań.
●Opcjonalna analiza połączeń z sekwencjonowaniem mRNA jednokomórkowych
Specyfikacje
| Wymagania przykładowe | Biblioteka | Strategia sekwencjonowania | Zalecane dane | Kontrola jakości |
| Próbki krio-Egched-Egched (Optymalna średnica: ok. 6x6x6 mm³) 2 bloki na próbkę | Biblioteka cDNA 10x Visium | Illumina PE150 | 50K PE odczytuje na miejsce (60 GB) | Rin> 7 |
Aby uzyskać więcej informacji na temat przykładowych wskazówek dotyczących przygotowania i przepływu pracy, prosimy o rozmowę zBMKGENE EXTRIC
Przepływ pracy serwisowej
W fazie przygotowania próbki przeprowadza się początkowe badanie ekstrakcji luzem RNA, aby zapewnić, że można uzyskać wysokiej jakości RNA. Na etapie optymalizacji tkanki sekcje są zabarwione i wizualizowane, a warunki permeabilizacji do uwalniania mRNA z tkanki są zoptymalizowane. Zoptymalizowany protokół jest następnie stosowany podczas budowy biblioteki, a następnie sekwencjonowanie i analiza danych.
Pełny przepływ pracy usług obejmuje aktualizacje w czasie rzeczywistym i potwierdzenia klienta w celu utrzymania responsywnej pętli opinii, zapewniając płynne wykonanie projektu.

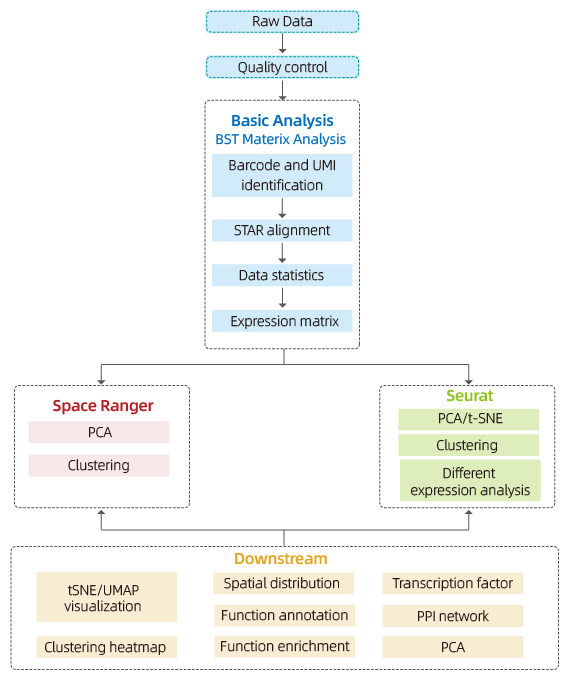
Obejmuje następującą analizę:
Kontrola jakości danych:
o Wyjście danych i rozkład jakości jakości
o Wykrywanie genów na miejsce
o Zakres tkanek
Analiza wewnętrznego próbki:
o bogactwo genów
o Klastrowanie punktowe, w tym analiza zmniejszonego wymiaru
o Analiza ekspresji między klastrami: Identyfikacja genów markerów
o Funkcjonalna adnotacja i wzbogacenie genów markerowych
Analiza międzygrupowa
o Ponowne kombinacja plam z obu próbek (np. Chore i kontroli) i ponowne klaster
o Identyfikacja genów markerów dla każdego klastra
o Funkcjonalna adnotacja i wzbogacenie genów markerowych
o Różnicowa ekspresja tego samego klastra między grupami
Analiza wewnętrznej próbki
Klastrowanie punktowe

Identyfikacja genów markerów i rozkład przestrzenny


Analiza międzygrupowa
Połączenie danych zarówno grup i ponownego klastra
Geny markerów nowych klastrów
Zbadaj postępy ułatwione przez BMKGene's Spatial transkryptomika usługi 10 -krotnej wizji w tych przedstawionych publikacjach:
Chen, D. i in. (2023) „MTHL1, potencjalny homolog Drosophila gpcr adhezji ssaków, bierze udział w reakcjach przeciwnowotworowych na wstrzyknięte komórki onkogenne u much”,Materiały z National Academy of Sciences of the United States of America, 120 (30), s. 1 E2303462120. doi: /10.1073/pnas.2303462120
Chen, Y. i in. (2023) „Stal umożliwia wyznaczenie w wysokiej rozdzielczości czasoprzestrzennych danych transkryptomicznych”,Odprawy w bioinformatyce, 24 (2), s. 1–10. DOI: 10.1093/BIB/BBAD068.
Liu, C. i in. (2022) „Atlas czasoprzestrzenny organogenezy w rozwoju kwiatów orchidei”,Badanie kwasów nukleinowych, 50 (17), s. 9724–9737. doi: 10.1093/nar/gKAC773.
Wang, J. i in. (2023) „Integrowanie transkryptomiki przestrzennej i sekwencjonowanie RNA pojedynczego Nukleusa ujawnia potencjalne strategie terapeutyczne dla leczówki macicy”,International Journal of Biological Sciences, 19 (8), s. 2515–2530. doi: 10.7150/ijbs.83510.