-

Metagenomisk sekvensering -NGS
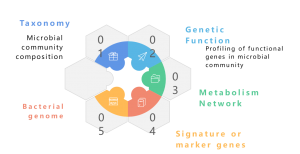
Et metagenom er en samling av det totale genetiske materialet til et blandet samfunn av organismer, for eksempel miljømessige og menneskelige metagenomer. Den inneholder genomer av både dyrkbare og ukultiverbare mikroorganismer. Hagle metagenomisk sekvensering med NGS muliggjør studiet av disse intrikate genomiske landskapene innebygd i miljøprøver ved å gi mer enn taksonomisk profilering, og gir også granulær innsikt i artsmangfold, overflodsdynamikk og komplekse populasjonsstrukturer. Utover taksonomiske studier, tilbyr hagle metagenomikk også et funksjonelt genomisk perspektiv, som muliggjør utforskning av kodede gener og deres antatte roller i økologiske prosesser. Til slutt bidrar etableringen av korrelasjonsnettverk mellom genetiske elementer og miljøfaktorer til en helhetlig forståelse av det intrikate samspillet mellom mikrobielle samfunn og deres økologiske bakgrunn. Avslutningsvis står metagenomisk sekvensering som et sentralt instrument for å avdekke de genomiske forviklingene til forskjellige mikrobielle samfunn, og belyse de mangefasetterte forholdene mellom genetikk og økologi i disse komplekse økosystemene.
Plattformer: Illumina NovaSeq og DNBSEQ-T7
-

Metagenomisk sekvensering-TGS
Et metagenom er en samling av det genetiske materialet til et blandet samfunn av organismer, for eksempel miljømessige og menneskelige metagenomer. Den inneholder genomer av både dyrkbare og ukultiverbare mikroorganismer. Metagenomisk sekvensering muliggjør studiet av disse intrikate genomiske landskapene innebygd i økologiske prøver ved å gi mer enn taksonomisk profilering. Det tilbyr også et funksjonelt genomikkperspektiv ved å utforske de kodede genene og deres antatte roller i miljøprosesser. Mens tradisjonelle hagletilnærminger med Illumina-sekvensering har blitt mye brukt i metagenomiske studier, har bruken av Nanopore og PacBio langlest sekvensering endret feltet. Nanopore- og PacBio-teknologi forbedrer nedstrøms bioinformatiske analyser, spesielt metagenommontering, og sikrer mer kontinuerlige sammenstillinger. Rapporter indikerer at Nanopore-basert og PacBio-basert metagenomikk har vellykket generert komplette og lukkede bakterielle genomer fra komplekse mikrobiomer (Moss, EL, et al., Nature Biotech, 2020). Integrering av Nanopore-lesninger med Illumina-lesninger gir en strategisk tilnærming for feilretting, og reduserer Nanopores iboende lave nøyaktighet. Denne synergistiske kombinasjonen utnytter styrken til hver sekvenseringsplattform, og tilbyr en robust løsning for å overvinne potensielle begrensninger og fremme presisjonen og påliteligheten til metagenomiske analyser.
Plattform: Nanopore PromethION 48, Illumia og PacBio Revio
-

Helgenom bisulfitt sekvensering (WGBS)
Whole Genome Bisulfite Sequencing (WGBS) står som gullstandardmetodikken for dyptgående utforskning av DNA-metylering, nærmere bestemt den femte posisjonen i cytosin (5-mC), en sentral regulator for genuttrykk og cellulær aktivitet. Prinsippet som ligger til grunn for WGBS involverer bisulfittbehandling, som induserer omdannelsen av umetylerte cytosiner til uracil (C til U), mens metylerte cytosiner forblir uendret. Denne teknikken tilbyr enkeltbaseoppløsning, slik at forskere kan undersøke metylomet grundig og avdekke unormale metyleringsmønstre assosiert med forskjellige tilstander, spesielt kreft. Ved å bruke WGBS kan forskere få enestående innsikt i genomomfattende metyleringslandskap, og gi en nyansert forståelse av de epigenetiske mekanismene som ligger til grunn for ulike biologiske prosesser og sykdommer.
-

Analyse for Transposase-tilgjengelig kromatin med høy gjennomstrømningssekvensering (ATAC-seq)
ATAC-seq er en sekvenseringsteknikk med høy gjennomstrømning som brukes for genomomfattende kromatintilgjengelighetsanalyse. Bruken gir en dypere forståelse av de komplekse mekanismene for global epigenetisk kontroll over genuttrykk. Metoden bruker en hyperaktiv Tn5-transposase for å samtidig fragmentere og merke åpne kromatinregioner ved å sette inn sekvenseringsadaptere. Påfølgende PCR-amplifisering resulterer i opprettelsen av et sekvenseringsbibliotek, som muliggjør omfattende identifikasjon av åpne kromatinregioner under spesifikke rom-tid-forhold. ATAC-seq gir et helhetlig syn på tilgjengelige kromatinlandskap, i motsetning til metoder som utelukkende fokuserer på transkripsjonsfaktorbindingssteder eller spesifikke histonmodifiserte regioner. Ved å sekvensere disse åpne kromatinregionene avslører ATAC-seq regioner som er mer sannsynlige for aktive regulatoriske sekvenser og potensielle transkripsjonsfaktorbindingssteder, og tilbyr verdifull innsikt i den dynamiske moduleringen av genuttrykk over genomet.
-

16S/18S/ITS Amplicon Sequencing-PacBio
16S og 18S rRNA-genene, sammen med Internal Transcribed Spacer (ITS)-regionen, fungerer som pivotale molekylære fingeravtrykksmarkører på grunn av deres kombinasjon av svært konserverte og hypervariable regioner, noe som gjør dem til uvurderlige verktøy for å karakterisere prokaryote og eukaryote organismer. Amplifisering og sekvensering av disse regionene tilbyr en isolasjonsfri tilnærming for å undersøke den mikrobielle sammensetningen og mangfoldet på tvers av ulike økosystemer. Mens Illumina-sekvensering typisk retter seg mot korte hypervariable regioner som V3-V4 av 16S og ITS1, har det blitt demonstrert at overlegen taksonomisk merknad er oppnåelig ved å sekvensere hele lengden av 16S, 18S og ITS. Denne omfattende tilnærmingen resulterer i høyere prosentandeler av nøyaktig klassifiserte sekvenser, og oppnår et oppløsningsnivå som strekker seg til artsidentifikasjon. PacBios Single-Molecule Real-Time (SMRT) sekvenseringsplattform skiller seg ut ved å gi svært nøyaktige lange avlesninger (HiFi) som dekker full-lengde amplicons, og konkurrerer med presisjonen til Illumina-sekvensering. Denne evnen lar forskere oppnå en uovertruffen fordel - en panoramautsikt over det genetiske landskapet. Den utvidede dekningen hever oppløsningen i artsannotering betydelig, spesielt innenfor bakterie- eller soppsamfunn, noe som muliggjør en dypere forståelse av vanskelighetene til mikrobielle populasjoner.
-

16S/18S/ITS Amplicon Sequencing-NGS
Amplikonsekvensering med Illumina-teknologi, spesifikt rettet mot de genetiske markørene 16S, 18S og ITS, er en kraftig metode for å avdekke fylogenien, taksonomien og artsoverfloden i mikrobielle samfunn. Denne tilnærmingen innebærer å sekvensere de hypervariable regionene til husholdningsgenetiske markører. Opprinnelig introdusert som et molekylært fingeravtrykk avWoeses et ali 1977 har denne teknikken revolusjonert mikrobiomprofilering ved å muliggjøre isolasjonsfrie analyser. Gjennom sekvensering av 16S (bakterier), 18S (sopp) og Internal Transscribed Spacer (ITS, fungi), kan forskere identifisere ikke bare rikelige arter, men også sjeldne og uidentifiserte. Amplikonsekvensering, som er bredt tatt i bruk som et sentralt verktøy, har blitt medvirkende til å identifisere differensielle mikrobielle sammensetninger på tvers av forskjellige miljøer, inkludert menneskelig munn, tarm, avføring og videre.
-

Re-sekvensering av hele genomet av bakterier og sopp
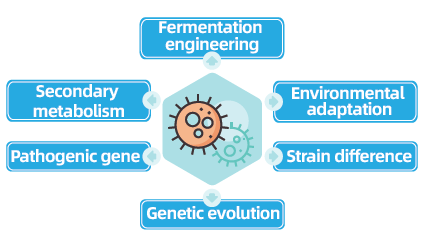
Bakterielle og sopplige helgenomresekvenseringsprosjekter er sentrale for å fremme mikrobiell genomikk ved å muliggjøre komplettering og sammenligning av mikrobielle genomer. Dette letter fermenteringsteknikk, optimalisering av industrielle prosesser og utforskning av sekundære metabolismeveier. Videre er sopp- og bakteriell re-sekvensering avgjørende for å forstå miljøtilpasning, optimalisere stammer og avsløre genetisk evolusjonsdynamikk, med brede implikasjoner innen medisin, landbruk og miljøvitenskap.
-

PacBio-full-lengde 16S/18S/ITS Amplicon Sequencing
Amplicon (16S/18S/ITS)-plattformen er utviklet med mange års erfaring innen mikrobiell mangfoldsprosjektanalyse, som inneholder standardisert grunnleggende analyse og personalisert analyse: grunnleggende analyse dekker det vanlige analyseinnholdet i gjeldende mikrobiell forskning, analyseinnholdet er rikt og omfattende, og analyseresultater presenteres i form av prosjektrapporter; Innholdet i personalisert analyse er mangfoldig. Prøver kan velges og parametere kan settes fleksibelt i henhold til den grunnleggende analyserapporten og forskningsformålet, for å realisere personlige krav. Windows-operativsystem, enkelt og raskt.
-

PacBio-transkriptom i full lengde (ikke-referanse)
Ved å ta Pacific Biosciences (PacBio) Isoform-sekvenseringsdata som input, er denne appen i stand til å identifisere transkripsjonssekvenser i full lengde (uten montering). Ved å kartlegge sekvenser i full lengde mot referansegenom, kan transkripsjoner optimaliseres av kjente gener, transkripsjoner, kodende regioner, etc. I dette tilfellet kan mer nøyaktig identifikasjon av mRNA-strukturer, som alternativ spleising, etc, oppnås. Fellesanalyse med NGS-transkriptomsekvenseringsdata muliggjør mer omfattende annotering og mer nøyaktig kvantifisering i uttrykk på transkripsjonsnivå, noe som i stor grad gagner nedstrøms differensialekspresjon og funksjonell analyse.
-

Reduced Representation Bisulfite Sequencing (RRBS)
Reduced Representation Bisulfite Sequencing (RRBS) har dukket opp som et kostnadseffektivt og effektivt alternativ til Whole Genome Bisulfite Sequencing (WGBS) i DNA-metyleringsforskning. Mens WGBS gir omfattende innsikt ved å undersøke hele genomet med enkeltbaseoppløsning, kan den høye kostnaden være en begrensende faktor. RRBS demper denne utfordringen strategisk ved selektivt å analysere en representativ del av genomet. Denne metodikken er avhengig av berikelse av CpG-øyrike regioner ved MspI-spalting etterfulgt av størrelsesvalg av 200-500/600 bps fragmenter. Følgelig blir bare regioner proksimalt til CpG-øyene sekvensert, mens de med fjerne CpG-øyer er ekskludert fra analysen. Denne prosessen, kombinert med bisulfitt-sekvensering, tillater høyoppløselig deteksjon av DNA-metylering, og sekvenseringstilnærmingen, PE150, fokuserer spesifikt på endene av innsatsene i stedet for midten, noe som øker effektiviteten til metyleringsprofilering. RRBS er et uvurderlig verktøy som muliggjør kostnadseffektiv DNA-metyleringsforskning og fremmer kunnskap om epigenetiske mekanismer.
-

Prokaryotisk RNA-sekvensering
RNA-sekvensering styrker den omfattende profileringen av alle RNA-transkripsjoner i celler under spesifikke forhold. Denne banebrytende teknologien fungerer som et potent verktøy som avslører intrikate genuttrykksprofiler, genstrukturer og molekylære mekanismer assosiert med ulike biologiske prosesser. RNA-sekvensering, som er bredt tatt i bruk innen grunnleggende forskning, klinisk diagnostikk og medikamentutvikling, og gir innsikt i vanskelighetene med cellulær dynamikk og genetisk regulering. Vår prokaryote RNA-prøvebehandling er skreddersydd for prokaryote transkriptomer, som involverer rRNA-uttømming og retningsbestemt bibliotekforberedelse.
Plattform: Illumina NovaSeq
-

Metatranskriptomsekvensering
Ved å utnytte Illumina-sekvenseringsteknologien avslører BMKGENEs metatranskriptomsekvenseringstjeneste det dynamiske genuttrykket til et mangfoldig utvalg av mikrober, som spenner fra eukaryoter til prokaryoter og virus, i naturlige miljøer som jord, vann, hav, avføring og tarm. Vår omfattende tjeneste gir forskere mulighet til å fordype seg i komplekse mikrobielle samfunns komplette genekspresjonsprofiler. Utover taksonomisk analyse, letter vår metatranskriptomsekvenseringstjeneste utforskning av funksjonell berikelse, og kaster lys over differensielt uttrykte gener og deres roller. Avdekk et vell av biologisk innsikt mens du navigerer i det komplekse landskapet av genuttrykk, taksonomisk mangfold og funksjonell dynamikk innenfor disse mangfoldige miljønisjene.
-

De novo Fungal Genome Assembly
BMKGENE tilbyr allsidige løsninger for soppgenomer, som imøtekommer ulike forskningsbehov og ønsket genomkompletthet. Bruk av kortlest Illumina-sekvensering alene tillater generering av et utkast til genom. Short-reads og long-read sekvensering ved bruk av Nanopore eller Pacbio kombineres for et mer raffinert soppgenom med lengre contigs. Dessuten forbedrer integrering av Hi-C-sekvensering mulighetene ytterligere, noe som muliggjør oppnåelse av et komplett genom på kromosomnivå.