Verminderde representatie bisulfietsequencing (RRBS)
Servicefuncties
● Vereist een referentiegenoom.
● Lambda-DNA wordt gebruikt om de bisulfietomzettingsefficiëntie te monitoren.
● De efficiëntie van de MspI-vertering wordt ook gemonitord.
● Dubbele enzymvertering voor plantenmonsters.
● Sequencing op Illumina NovaSeq.
Servicevoordelen
●Kosteneffectief en efficiënt alternatief voor WGBS: waardoor de analyse tegen lagere kosten en met lagere monstervereisten kan worden uitgevoerd.
●Compleet platform:bieden one-stop uitstekende service, van monsterverwerking, bibliotheekconstructie en sequencing tot bio-informatica-analyse.
●Uitgebreide expertise: nu RRBS-sequencingprojecten met succes zijn voltooid voor een breed scala aan soorten, brengt BMKGENE meer dan tien jaar ervaring, een zeer bekwaam analyseteam, uitgebreide inhoud en uitstekende ondersteuning na de verkoop met zich mee.
Servicespecificaties
| Bibliotheek | Sequentiestrategie | Aanbevolen gegevensuitvoer | Kwaliteitscontrole |
| Met MspI gedigereerde en met Bisulfiet behandelde bibliotheek | Illumina PE150 | 8 GB | Q30 ≥ 85% Bisulfietconversie > 99% MspI snijefficiëntie > 95% |
Voorbeeldvereisten
| Concentratie (ng/μL) | Totaal bedrag (µg) |
| |
| Genomisch DNA | ≥ 30 | ≥ 1 | Beperkte afbraak of vervuiling |
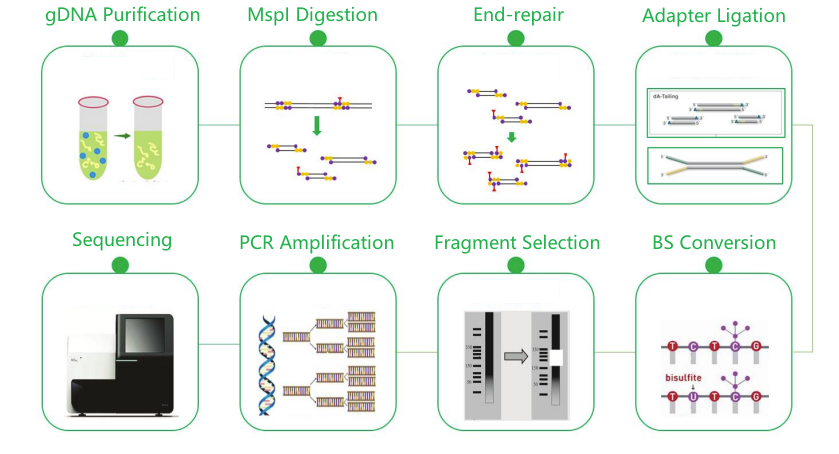
Servicewerkstroom

Levering van monsters

Bouw van bibliotheek

Sequencing

Gegevensanalyse
After-sales diensten
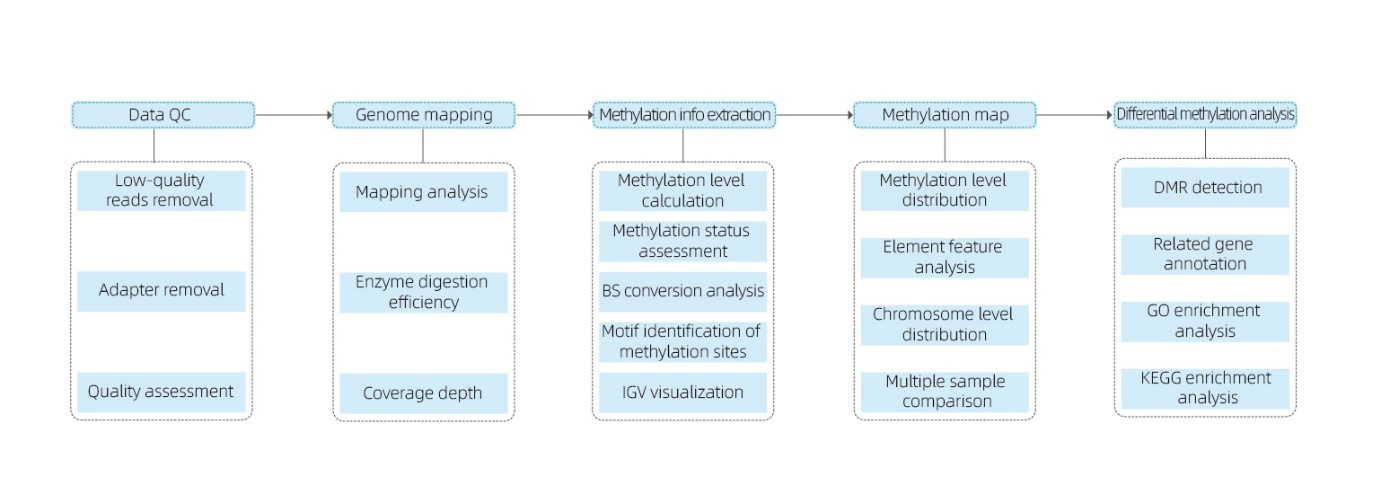
Bevat de volgende analyse:
● Kwaliteitscontrole van ruwe sequenties;
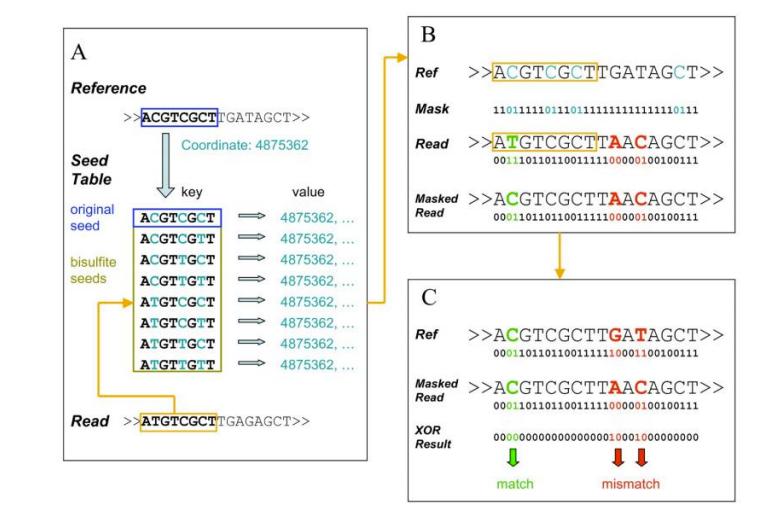
● In kaart brengen van het referentiegenoom;
● Detectie van 5mC-gemethyleerde basen en motiefidentificatie;
● Analyse van de methylatieverdeling en monstervergelijking;
● Analyse van differentieel gemethyleerde regio's (DMR's);
● Functionele annotatie van genen geassocieerd met DMR's.
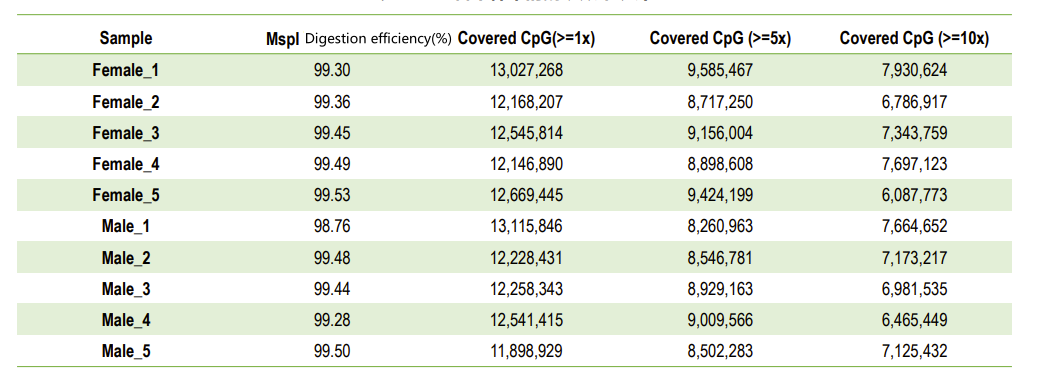
Kwaliteitscontrole: verteringsefficiëntie (bij het in kaart brengen van het genoom)
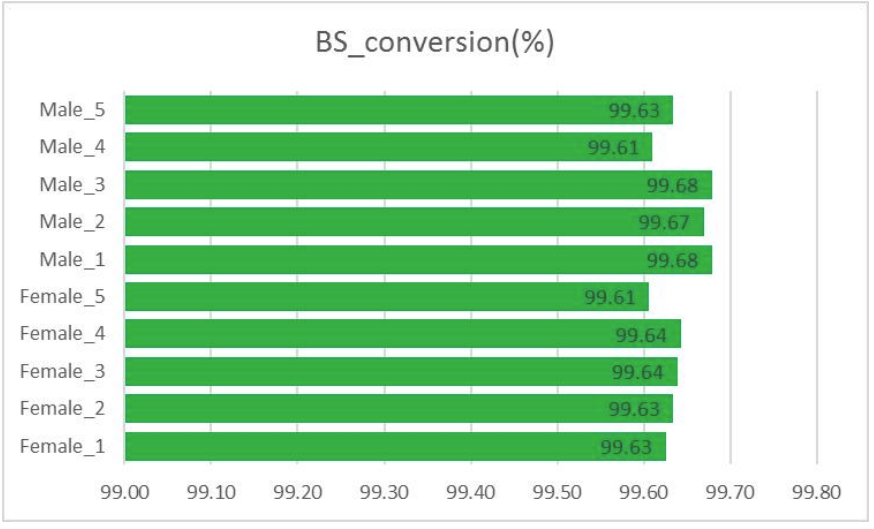
Kwaliteitscontrole: bisulfietconversie (bij methylatie-informatie-extractie)
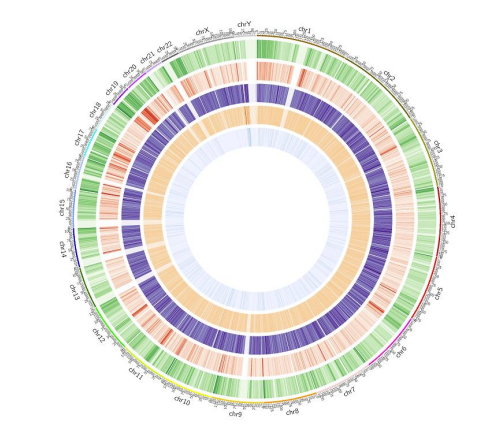
Methylatiekaart: 5mC-methylatie-genoombrede distributie
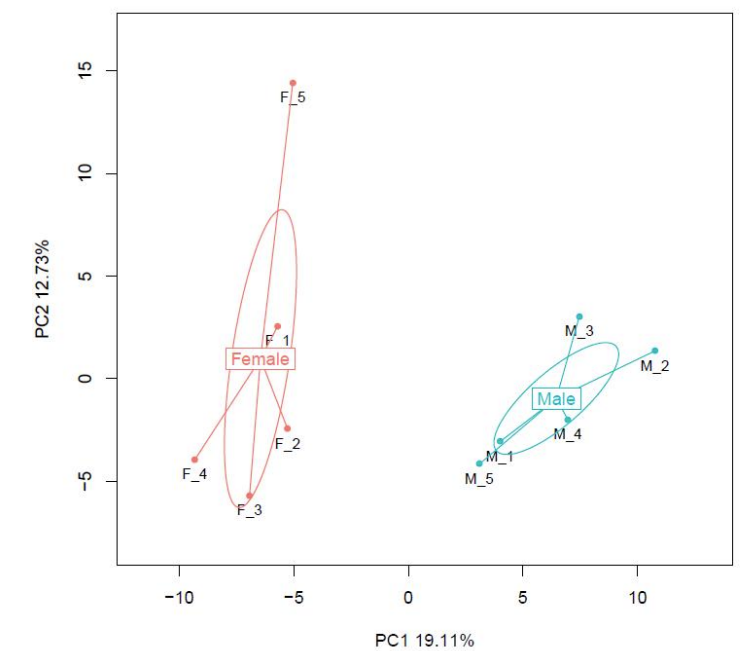
Voorbeeldvergelijking: analyse van hoofdcomponenten
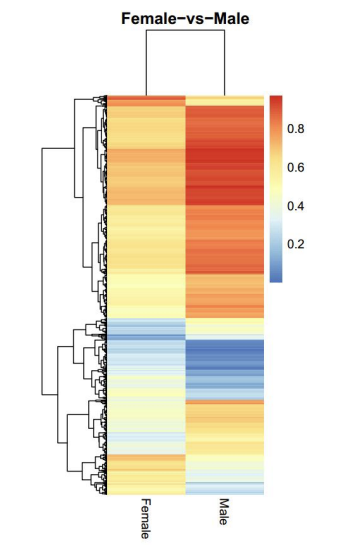
Analyse van differentieel gemethyleerde regio's (DMR's): heatmap
Ontdek de onderzoeksvooruitgang die wordt mogelijk gemaakt door de bisulfietsequencingdiensten van BMKGene via een samengestelde verzameling publicaties.
Li, Z. et al. (2022) 'Hoogwaardige herprogrammering in Leydig-achtige cellen door CRISPR-activatie en paracriene factoren',PNAS-nexus, 1(4). doi: 10.1093/PNASNEXUS/PGAC179.
Tian, H. et al. (2023) 'Genoombrede DNA-methylatieanalyse van de lichaamssamenstelling bij Chinese monozygote tweelingen',Europees tijdschrift voor klinisch onderzoek, 53(11), p. e14055. doi: 10.1111/ECI.14055.
Wu, Y. et al. (2022) 'DNA-methylatie en taille-heupverhouding: een epigenoombrede associatiestudie bij Chinese monozygote tweelingen',Journal of Endocrinologisch Onderzoek, 45(12), blz. 2365–2376. doi: 10.1007/S40618-022-01878-4.