-

Metagenomische sequencing-NGS
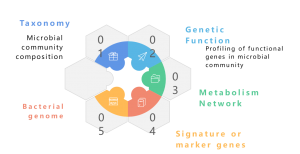
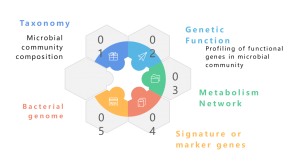
Een metagenoom is een verzameling van het totale genetische materiaal van een gemengde gemeenschap van organismen, zoals milieu- en menselijke metagenomen. Het bevat genomen van zowel kweekbare als niet-kweekbare micro-organismen. Shotgun-metagenomische sequencing met NGS maakt de studie mogelijk van deze ingewikkelde genomische landschappen ingebed in milieumonsters door meer te bieden dan alleen taxonomische profilering, en ook gedetailleerde inzichten te geven in de soortendiversiteit, de dynamiek van de overvloed en complexe populatiestructuren. Naast taxonomische studies biedt shotgun-metagenomica ook een functioneel genomicsperspectief, waardoor de verkenning van gecodeerde genen en hun vermeende rol in ecologische processen mogelijk wordt gemaakt. Ten slotte draagt het opzetten van correlatienetwerken tussen genetische elementen en omgevingsfactoren bij aan een holistisch begrip van de ingewikkelde wisselwerking tussen microbiële gemeenschappen en hun ecologische achtergrond. Concluderend is metagenomische sequencing een cruciaal instrument voor het ontrafelen van de genomische ingewikkeldheden van diverse microbiële gemeenschappen, waardoor de veelzijdige relaties tussen genetica en ecologie binnen deze complexe ecosystemen worden belicht.
Platformen: Illumina NovaSeq en DNBSEQ-T7
-

Metagenomische sequencing-TGS
Een metagenoom is een verzameling genetisch materiaal van een gemengde gemeenschap van organismen, zoals milieu- en menselijke metagenomen. Het bevat genomen van zowel kweekbare als niet-kweekbare micro-organismen. Metagenomische sequencing maakt de studie mogelijk van deze ingewikkelde genomische landschappen ingebed in ecologische monsters door meer te bieden dan alleen taxonomische profilering. Het biedt ook een functioneel genomicsperspectief door de gecodeerde genen en hun vermeende rol in omgevingsprocessen te onderzoeken. Terwijl traditionele shotgun-benaderingen met Illumina-sequencing op grote schaal zijn gebruikt in metagenomische studies, heeft de komst van Nanopore en PacBio long-read sequencing het veld veranderd. Nanopore- en PacBio-technologie verbeteren de stroomafwaartse bio-informatica-analyses, met name de assemblage van metagenoom, waardoor meer continue assemblages worden gegarandeerd. Uit rapporten blijkt dat op Nanopore en PacBio gebaseerde metagenomica met succes complete en gesloten bacteriële genomen uit complexe microbiomen hebben gegenereerd (Moss, EL, et al., Nature Biotech, 2020). Het integreren van Nanopore-lezingen met Illumina-lezingen biedt een strategische aanpak voor foutcorrectie, waardoor de inherente lage nauwkeurigheid van Nanopore wordt verzacht. Deze synergetische combinatie maakt gebruik van de sterke punten van elk sequencingplatform en biedt een robuuste oplossing om potentiële beperkingen te overwinnen en de precisie en betrouwbaarheid van metagenomische analyses te bevorderen.
Platform: Nanopore PromethION 48, Illumia en PacBio Revio
-

16S/18S/ITS Amplicon-sequencing-PacBio
De 16S- en 18S-rRNA-genen dienen, samen met de Internal Transcribed Spacer (ITS) -regio, als cruciale markers voor moleculaire vingerafdrukken vanwege hun combinatie van sterk geconserveerde en hypervariabele regio's, waardoor ze van onschatbare waarde zijn voor het karakteriseren van prokaryotische en eukaryotische organismen. Amplificatie en sequencing van deze regio's bieden een isolatievrije aanpak voor het onderzoeken van de microbiële samenstelling en diversiteit in verschillende ecosystemen. Hoewel Illumina-sequencing zich doorgaans richt op korte hypervariabele gebieden zoals V3-V4 van 16S en ITS1, is aangetoond dat superieure taxonomische annotatie haalbaar is door de volledige lengte van 16S, 18S en ITS te sequencen. Deze alomvattende aanpak resulteert in hogere percentages nauwkeurig geclassificeerde sequenties, waardoor een resolutieniveau wordt bereikt dat zich uitstrekt tot soortidentificatie. PacBio's Single-Molecule Real-Time (SMRT) sequencingplatform onderscheidt zich door het bieden van zeer nauwkeurige lange metingen (HiFi) die de amplicons over de volledige lengte bestrijken, wat de precisie van Illumina-sequencing evenaart. Deze mogelijkheid stelt onderzoekers in staat een ongeëvenaard voordeel te behalen: een panoramisch beeld van het genetische landschap. De uitgebreide dekking verhoogt de resolutie in soortannotatie aanzienlijk, vooral binnen bacteriële of schimmelgemeenschappen, waardoor een dieper inzicht in de complexiteit van microbiële populaties mogelijk wordt.
-

16S/18S/ITS Amplicon-sequencing-NGS
Amplicon-sequencing met Illumina-technologie, specifiek gericht op de genetische markers 16S, 18S en ITS, is een krachtige methode voor het ontrafelen van de fylogenie, taxonomie en soortenrijkdom binnen microbiële gemeenschappen. Deze aanpak omvat het sequencen van de hypervariabele gebieden van genetische markers voor huishoudelijk gebruik. Oorspronkelijk geïntroduceerd als een moleculaire vingerafdruk doorWoeses et alin 1977 heeft deze techniek een revolutie teweeggebracht in de profilering van het microbioom door isolatievrije analyses mogelijk te maken. Door de sequentiebepaling van 16S (bacteriën), 18S (schimmels) en Internal Transcribed Spacer (ITS, schimmels) kunnen onderzoekers niet alleen overvloedige soorten identificeren, maar ook zeldzame en ongeïdentificeerde soorten. Amplicon-sequencing wordt algemeen aanvaard als een cruciaal hulpmiddel en is een instrument geworden bij het onderscheiden van verschillende microbiële samenstellingen in verschillende omgevingen, waaronder de menselijke mond, darmen, ontlasting en daarbuiten.
-

Hersequencing van het hele genoom van bacteriën en schimmels
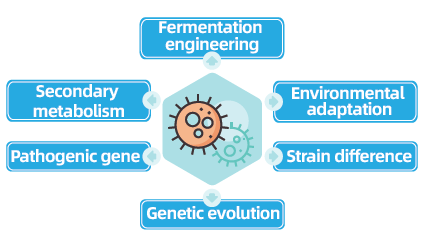
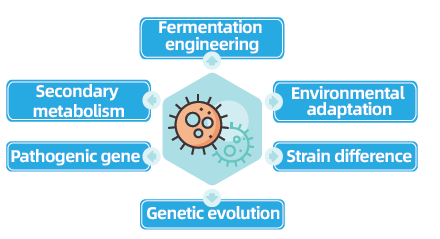
Hersequencing-projecten voor het hele genoom van bacteriën en schimmels zijn van cruciaal belang voor het bevorderen van microbiële genomica door de voltooiing en vergelijking van microbiële genomen mogelijk te maken. Dit vergemakkelijkt de fermentatietechniek, de optimalisatie van industriële processen en de verkenning van secundaire metabolismeroutes. Bovendien is het opnieuw rangschikken van schimmels en bacteriën cruciaal voor het begrijpen van aanpassing aan het milieu, het optimaliseren van stammen en het onthullen van de genetische evolutiedynamiek, met brede implicaties in de geneeskunde, de landbouw en de milieuwetenschappen.
-

Prokaryotische RNA-sequencing
RNA-sequencing maakt de uitgebreide profilering van alle RNA-transcripten in cellen onder specifieke omstandigheden mogelijk. Deze geavanceerde technologie dient als een krachtig hulpmiddel en onthult ingewikkelde genexpressieprofielen, genstructuren en moleculaire mechanismen die verband houden met diverse biologische processen. RNA-sequencing wordt op grote schaal toegepast in fundamenteel onderzoek, klinische diagnostiek en de ontwikkeling van geneesmiddelen en biedt inzicht in de fijne kneepjes van cellulaire dynamiek en genetische regulatie. Onze prokaryotische RNA-monsterverwerking is op maat gemaakt voor prokaryotische transcriptomen, waarbij rRNA-uitputting en directionele bibliotheekvoorbereiding betrokken zijn.
Platform: Illumina NovaSeq
-

Metatranscriptoomsequencing
Door gebruik te maken van de Illumina-sequencing-technologie onthult de metatranscriptoomsequencing-service van BMKGENE de dynamische genexpressie van een diverse reeks microben, van eukaryoten tot prokaryoten en virussen, in natuurlijke omgevingen zoals bodem, water, zee, ontlasting en de darmen. Onze uitgebreide service stelt onderzoekers in staat zich te verdiepen in de volledige genexpressieprofielen van complexe microbiële gemeenschappen. Naast taxonomische analyse vergemakkelijkt onze metatranscriptoomsequencing-service het onderzoek naar functionele verrijking, waarbij licht wordt geworpen op differentieel tot expressie gebrachte genen en hun rol. Ontdek een schat aan biologische inzichten terwijl je navigeert door de complexe landschappen van genexpressie, taxonomische diversiteit en functionele dynamiek binnen deze diverse ecologische niches.
-

De novo Fungal Genome Assembly
BMKGENE biedt veelzijdige oplossingen voor schimmelgenomen, tegemoetkomend aan diverse onderzoeksbehoeften en gewenste volledigheid van het genoom. Alleen al het gebruik van short-read Illumina-sequencing maakt het genereren van een conceptgenoom mogelijk. Short-reads en long-read sequencing met behulp van Nanopore of Pacbio worden gecombineerd voor een verfijnder schimmelgenoom met langere contigs. Bovendien verbetert de integratie van Hi-C-sequencing de mogelijkheden verder, waardoor het mogelijk wordt een compleet genoom op chromosoomniveau te bereiken.
-

De novo bacteriële genoomassemblage
Wij bieden een complete service voor de assemblage van bacteriële genoomen, waarbij we garanderen dat er geen hiaten zijn. Dit is mogelijk door long-read sequencing-technologieën te integreren, zoals Nanopore en PacBio voor assemblage en short-read sequencing met Illumina voor assemblagevalidatie en foutcorrectie van ONT-lezingen. Onze service biedt de volledige bio-informatische workflow, van assemblage, functionele annotatie en geavanceerde bio-informatica-analyse, waarmee specifieke onderzoeksdoelen worden vervuld. Deze dienst maakt de ontwikkeling mogelijk van nauwkeurige referentiegenomen voor verschillende genetische en genomische studies. Bovendien vormt het de basis voor toepassingen zoals stamoptimalisatie, genetische manipulatie en ontwikkeling van microbiële technologie, waardoor betrouwbare en hiaatvrije genomische gegevens worden gegarandeerd die cruciaal zijn voor het bevorderen van wetenschappelijke inzichten en biotechnologische innovatie.