Op Hi-C gebaseerde genoomassemblage
Servicefuncties
● Sequencing op Illumina NovaSeq met PE150.
● Voor service zijn weefselmonsters nodig, in plaats van geëxtraheerde nucleïnezuren, om te crosslinken met formaldehyde en de DNA-eiwitinteracties te behouden.
● Het Hi-C-experiment omvat het beperken en repareren van de kleverige uiteinden met biotine, gevolgd door circularisatie van de resulterende stompe uiteinden terwijl de interacties behouden blijven. Het DNA wordt vervolgens met streptavidinekorrels naar beneden getrokken en gezuiverd voor daaropvolgende bereiding van de bibliotheek.
Servicevoordelen

Overzicht van Hi-C
(Lieberman-Aiden E et al.,Wetenschap, 2009)
●Het elimineren van de behoefte aan genetische populatiegegevens:Hi-C vervangt de essentiële informatie die nodig is voor contig-verankering.
●Hoge markerdichtheid:wat leidt tot een hoge contig-verankeringsverhouding van meer dan 90%.
●Uitgebreide expertise en publicatierecords:BMKGene heeft ruime ervaring met meer dan 2000 gevallen van Hi-C Genome Assembly van 1000 verschillende soorten en verschillende patenten. Meer dan 200 gepubliceerde gevallen hebben een cumulatieve impactfactor van meer dan 2000.
●Hooggekwalificeerd bio-informaticateam:Met interne patenten en softwareauteursrechten voor Hi-C-experimenten en data-analyse maakt de zelfontwikkelde visualisatiedatasoftware het handmatig verplaatsen, omkeren, intrekken en opnieuw uitvoeren van blokken mogelijk.
●Ondersteuning na verkoop:Onze inzet gaat verder dan de voltooiing van het project met een after-sales serviceperiode van 3 maanden. Gedurende deze tijd bieden we projectopvolging, hulp bij het oplossen van problemen en vraag- en antwoordsessies om eventuele vragen met betrekking tot de resultaten te beantwoorden.
●Uitgebreide annotatie: we gebruiken meerdere databases om de genen functioneel te annoteren met geïdentificeerde variaties en de bijbehorende verrijkingsanalyse uit te voeren, waardoor inzichten worden verkregen over meerdere onderzoeksprojecten.
Servicespecificaties
| Voorbereiding bibliotheek | Sequentiestrategie | Aanbevolen gegevensuitvoer | Kwaliteitscontrole |
| Hi-C-bibliotheek | Illumina NovaSeq PE150 | 100x | Q30 ≥ 85% |
Voorbeeldvereisten
| Weefsel | Vereist bedrag |
| Dierlijke ingewanden | ≥ 2 gram |
| Dierlijke spier | |
| Zoogdierenbloed | ≥ 2 ml |
| Gevogelte/visbloed | |
| Plant-vers blad | ≥ 3 gram |
| Gekweekte cellen | ≥ 1x107 |
| Insect | ≥ 2 gram |
Servicewerkstroom

Experimentontwerp

Levering van monsters

Bouw van bibliotheek

Sequencing

Gegevensanalyse

After-sales diensten
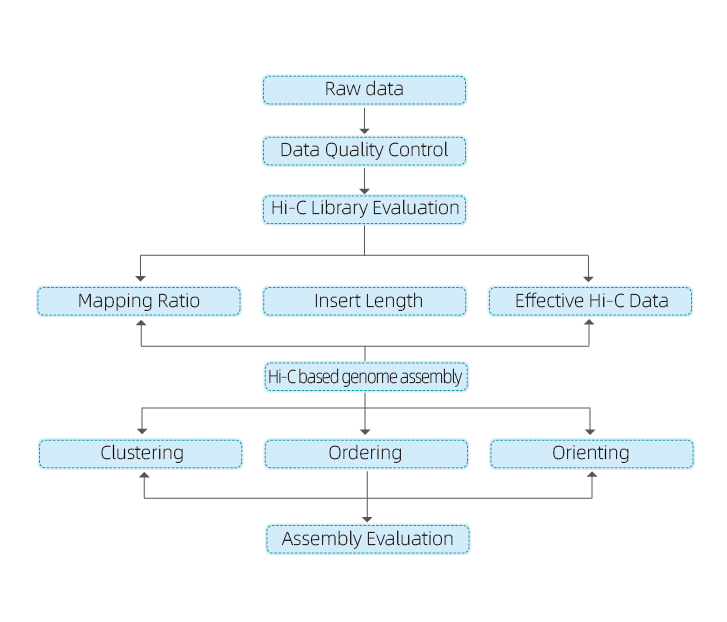
1) Kwaliteitscontrole van ruwe gegevens
2) QC van de Hi-C-bibliotheek: schatting van geldige Hi-C-interacties
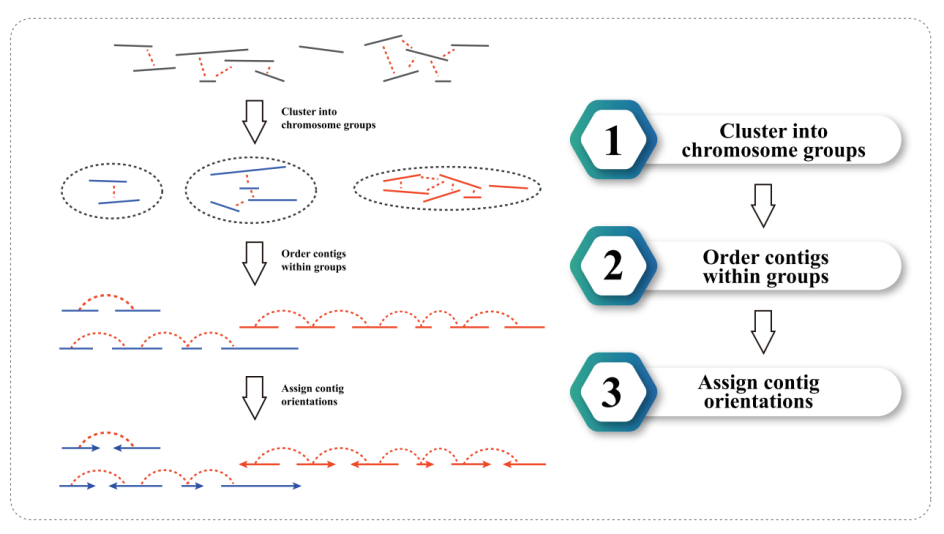
3) Hi-C-assemblage: clustering van contigs in groepen, gevolgd door contig-ordening binnen elke groep en het toewijzen van contig-oriëntatie
4) Hi-C-evaluatie
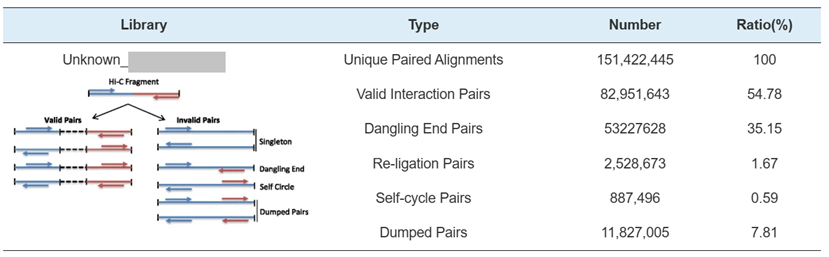
Hi-C Library QC – schatting van geldige Hi-C-interactieparen
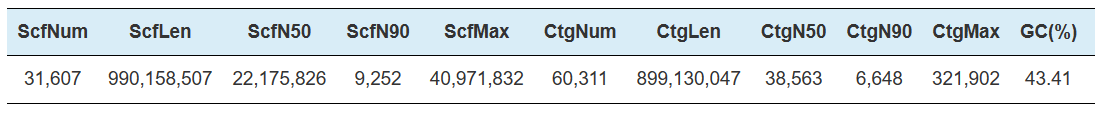
Hi-C Assembly – statistieken
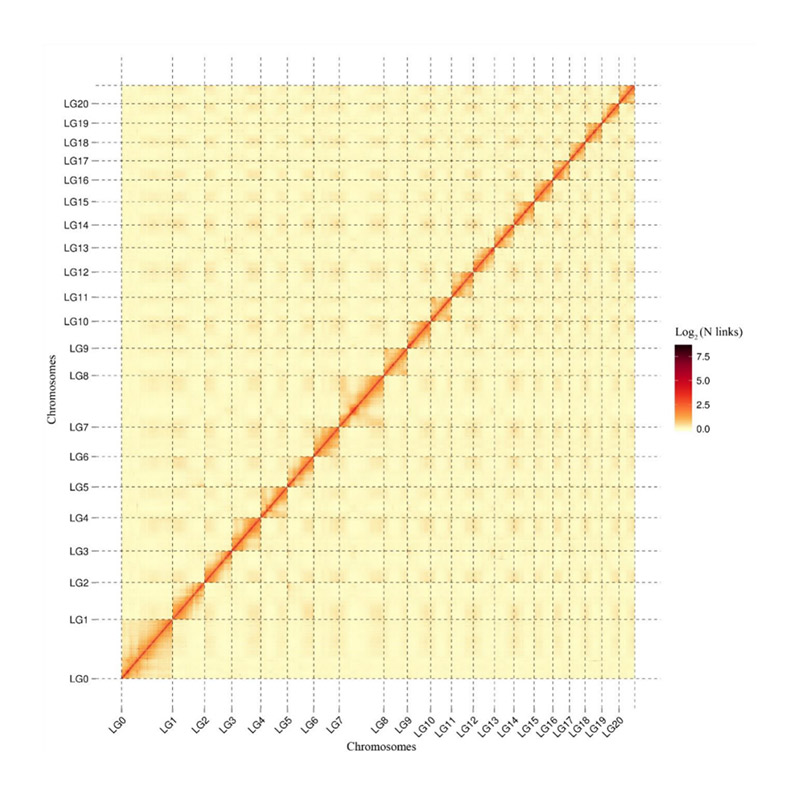
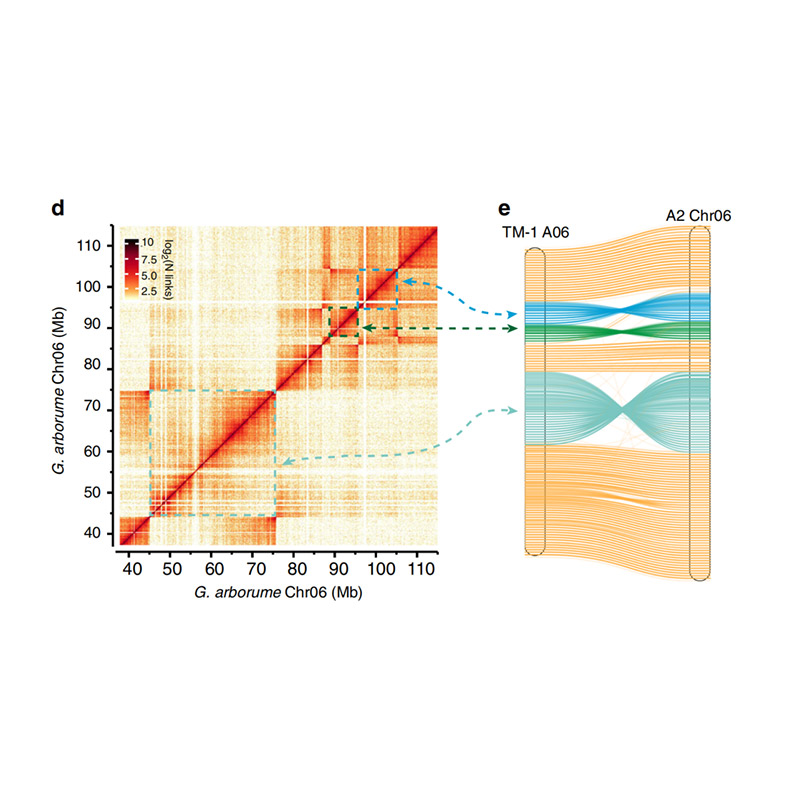
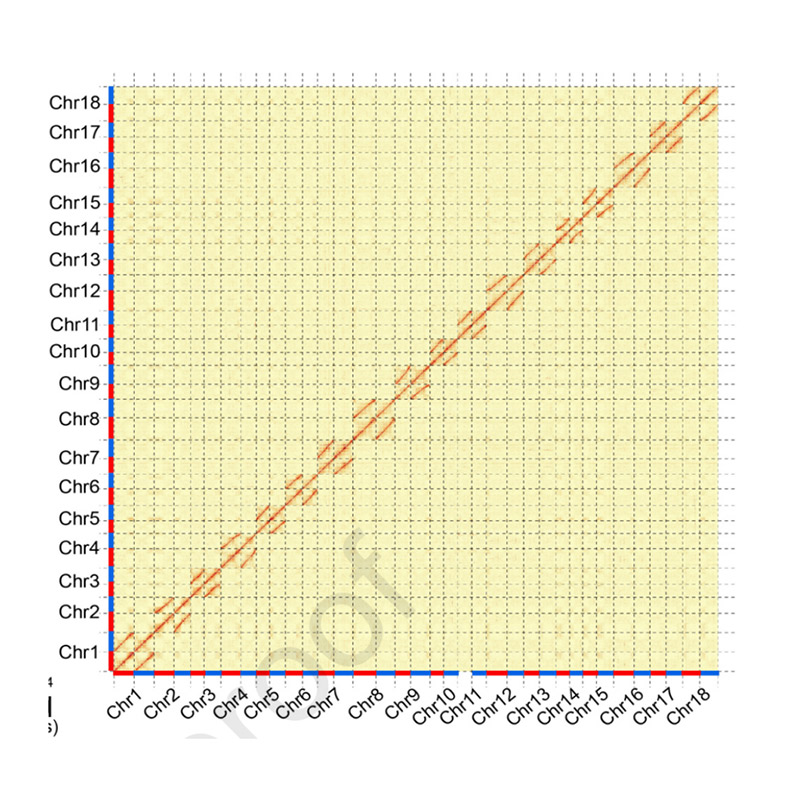
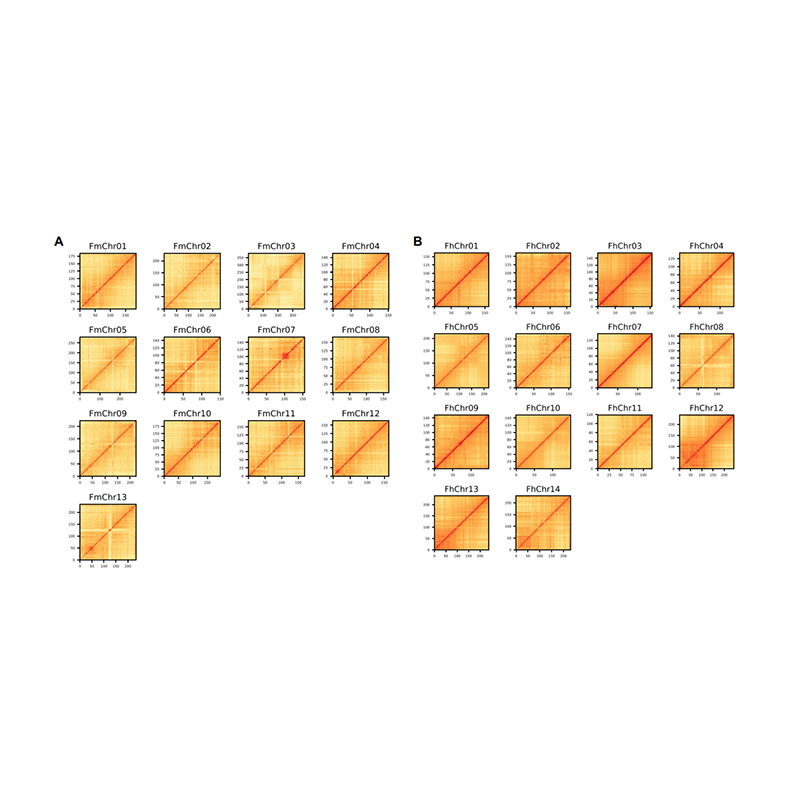
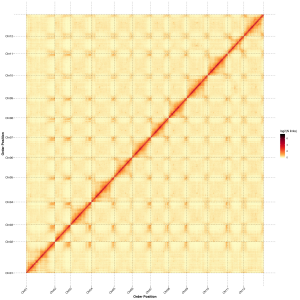
Evaluatie na montage – heatmap van signaalintensiteit tussen bins
Ontdek de vooruitgang die wordt mogelijk gemaakt door de Hi-C-assemblagediensten van BMKGene via een samengestelde verzameling publicaties.
Tian, T. et al. (2023) 'Genoomassemblage en genetische dissectie van een prominent droogteresistent maïskiemplasma', Nature Genetics 2023 55:3, 55(3), pp. 496–506. doi: 10.1038/s41588-023-01297-y.
Wang, ZL et al. (2020) 'Een assemblage op chromosomenschaal van het Aziatische honingbij Apis cerana-genoom', Frontiers in Genetics, 11, p. 524140. doi: 10.3389/FGENE.2020.00279/BIBTEX.
Zhang, F. et al. (2023) 'Onthullende evolutie van de biosynthese van tropaan-alkaloïden door twee genomen in de Solanaceae-familie te analyseren', Nature Communications 2023 14:1, 14(1), pp. 1–18. doi: 10.1038/s41467-023-37133-4.
Zhang, X. et al. (2020) 'Genomen van de banyanboom en bestuiverwesp bieden inzicht in de co-evolutie van vijgenwesp', Cell, 183(4), pp. 875-889.e17. doi: 10.1016/J.CELL.2020.09.043