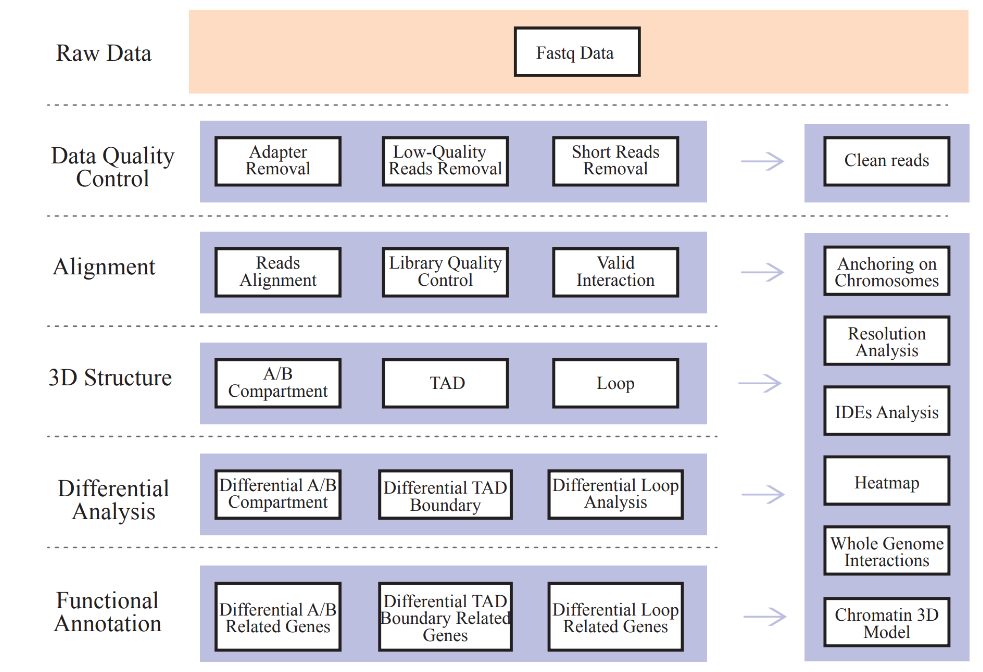
Op Hi-C gebaseerde chromatine-interactie
Servicefuncties
● Sequencing op Illumina NovaSeq met PE150.
● Voor service zijn weefselmonsters nodig, in plaats van geëxtraheerde nucleïnezuren, om te crosslinken met formaldehyde en de DNA-eiwitinteracties te behouden.
● Het Hi-C-experiment omvat het beperken en repareren van de kleverige uiteinden met biotine, gevolgd door circularisatie van de resulterende stompe uiteinden terwijl de interacties behouden blijven. Het DNA wordt vervolgens met streptavidinekorrels naar beneden getrokken en gezuiverd voor daaropvolgende bereiding van de bibliotheek.
Servicevoordelen
●Optimaal restrictie-enzymontwerp: om een hoge Hi-C-efficiëntie op verschillende soorten te garanderen met tot 93% geldige interactieparen.
●Uitgebreide expertise en publicatierecords:BMKGene heeft ruime ervaring met >2000 Hi-C-sequencingprojecten van 800 verschillende soorten en verschillende patenten. Meer dan 100 gepubliceerde cases met een cumulatieve impactfactor van meer dan 900.
●Hooggekwalificeerd bio-informaticateam:met interne patenten en softwareauteursrechten voor Hi-C-experimenten en data-analyse en een zelf ontwikkelde visualisatiedatasoftware.
●Ondersteuning na verkoop:Onze inzet gaat verder dan de voltooiing van het project met een after-sales serviceperiode van 3 maanden. Gedurende deze tijd bieden we projectopvolging, hulp bij het oplossen van problemen en vraag- en antwoordsessies om eventuele vragen met betrekking tot de resultaten te beantwoorden.
●Uitgebreide annotatie: we gebruiken meerdere databases om de genen functioneel te annoteren met geïdentificeerde variaties en de bijbehorende verrijkingsanalyse uit te voeren, waardoor inzichten worden verkregen over meerdere onderzoeksprojecten.
Servicespecificaties
| Bibliotheek | Sequentiestrategie | Aanbevolen gegevensuitvoer | Hi-C-signaalresolutie |
| Hi-C-bibliotheek | Illumina PE150 | Chromatinelus: 150x TAD: 50x | Chromatinelus: 10Kb TAD: 40Kb |
Servicevereisten
| Voorbeeldtype | Vereist bedrag |
| Dierlijk weefsel | ≥2g |
| Volbloed | ≥2 ml |
| Schimmels | ≥1g |
| Plant-jong weefsel | 1 g/aliquot, 2-4 porties aanbevolen |
| Gekweekte cellen | ≥1x107 |
Bevat de volgende analyse:
● Ruwe gegevens QC;
● Mapping en Hi-C bibliotheek QC: geldige interactieparen en Interaction Decay Exponents (IDE's);
● Genoombrede interactieprofilering: cis/trans-analyse en Hi-C-interactiekaart;
● Analyse van de distributie van A/B-compartimenten;
● Identificatie van TAD's en chromatinelussen;
● Differentiële analyse van 3D-chromatinestructuurelementen tussen monsters en overeenkomstige functionele annotatie van geassocieerde genen.
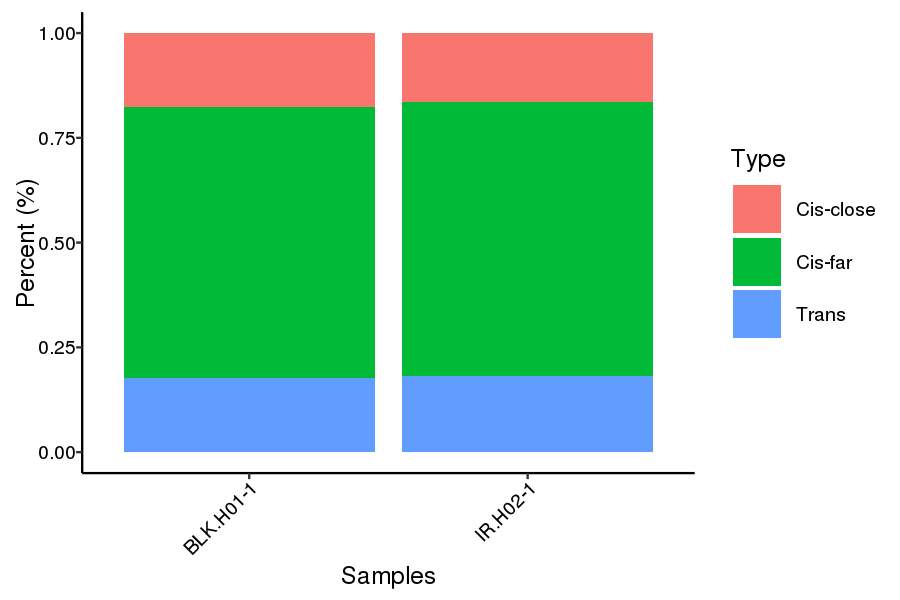
Cis- en trans-proportionele distributie
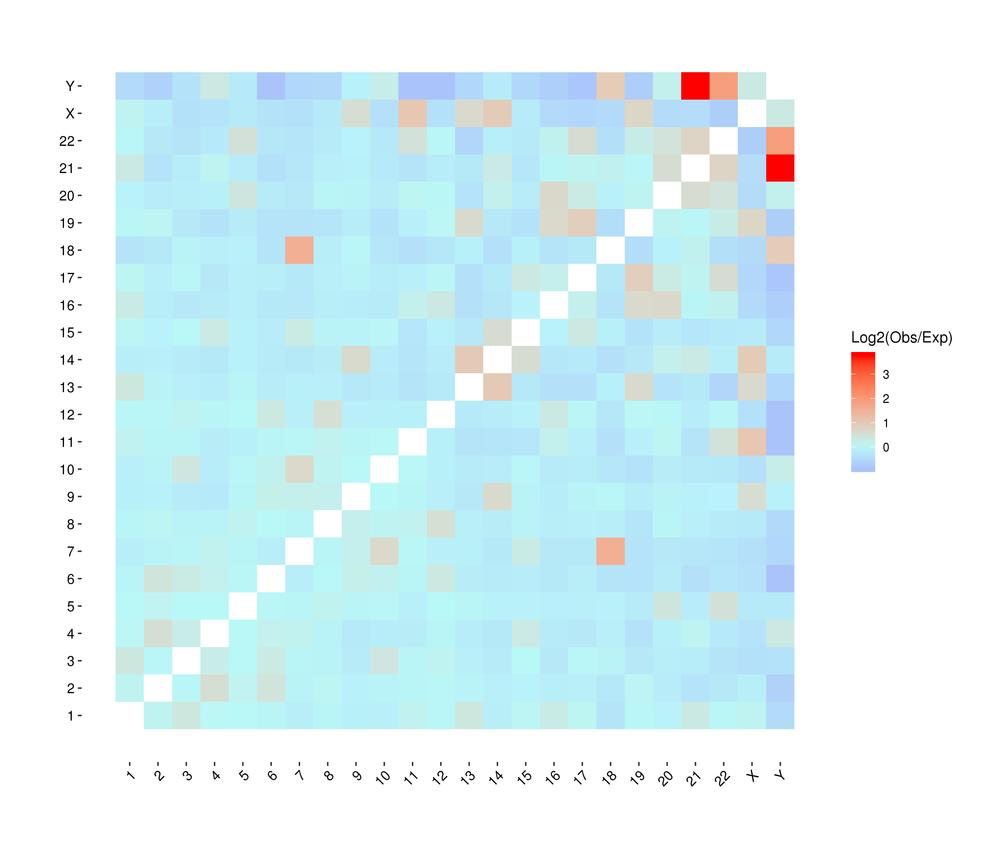
Heatmap van chromosomale interacties tussen monsters
Genoombrede distributie van A/B-compartimenten
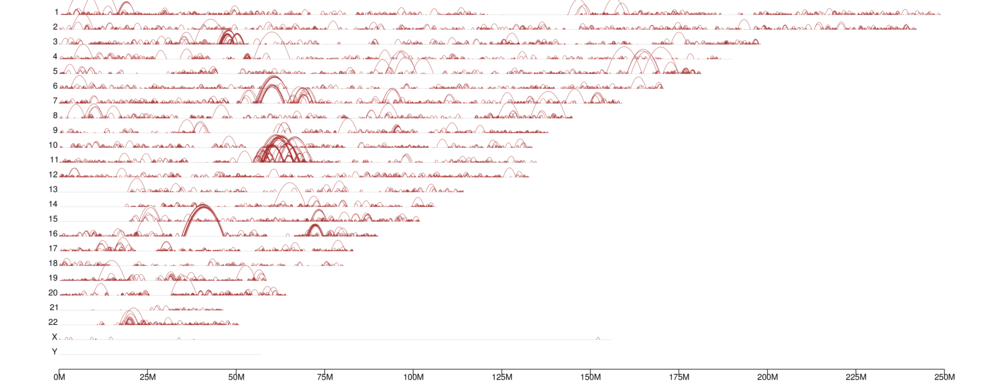
Genoombrede distributie van chromatinelussen
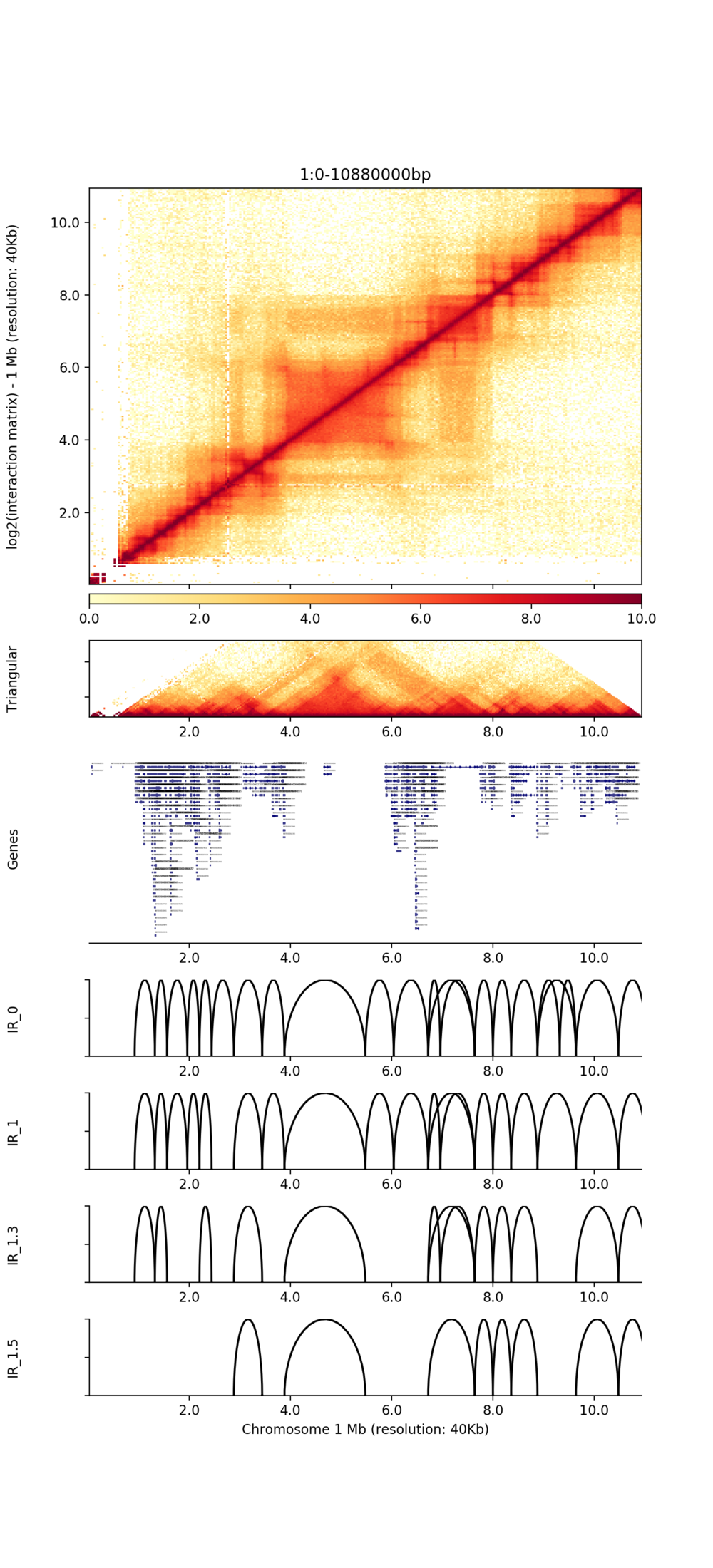
Visualisatie van TAD's
Ontdek de onderzoeksvooruitgang die wordt mogelijk gemaakt door de Hi-C-sequencingdiensten van BMKGene via een samengestelde verzameling publicaties.
Meng, T. et al. (2021) 'Een vergelijkende geïntegreerde multi-omics-analyse identificeert CA2 als een nieuw doelwit voor akkoorden',Neuro-Oncologie, 23(10), blz. 1709–1722. doi: 10.1093/NEUONC/NOAB156.
Xu, L. et al. (2021) '3D-desorganisatie en herschikking van het genoom bieden inzicht in de pathogenese van NAFLD door geïntegreerde Hi-C-, Nanopore- en RNA-sequencing',Acta Pharmaceutica Sinica B, 11(10), blz. 3150–3164. doi: 10.1016/J.APSB.2021.03.022.