DNBSEQ vooraf gemaakte bibliotheken
Functies
●Platform:MGI-DNBSEQ-T7
●Sequencing modi:PE150
●Overdracht van Illumina -bibliotheken naarMGI:het inschakelen van sequencing van hoge datavolumes tegen lage kosten.
●Kwaliteitscontrole van bibliotheken vóór sequencing.
●Sequencing Data QC en levering:Levering van QC -rapport en onbewerkte gegevens in FASTQ -indeling na demultiplexing en filteren Q30 leest.
Voordelen
●Veelzijdigheid van sequencing -services:De klant kan ervoor kiezen om te sequence op een rijstrook of hoeveelheid gegevens.
●Hoge gegevensuitvoer:1500 GB/rijstrook
●Levering van Sequencing QC -rapport:Met kwaliteitsstatistieken, gegevensnauwkeurigheid en algehele prestaties van het sequencing -project.
●Volwassen sequencingproces:met korte ommekeertijd.
●Rigoureuze kwaliteitscontrole: We implementeren strikte QC-vereisten om de levering van consistent hoogwaardige resultaten te garanderen.
Voorbeeldvereisten
| Gegevensbedrag (x) | Concentratie (qpcr/nm) | Volume | |
| Gedeeltelijke rijstrook
| X ≤ 10 GB | ≥ 1 nm | ≥ 25 ul |
| 10 GB <x ≤ 50 GB | ≥ 2 nm | ≥ 25 ul | |
| 50 GB <x ≤ 100 GB | ≥ 3 nm | ≥ 25 ul | |
| X> 100 GB | ≥ 4 nm | ||
| Enkele rijstrook | Per baan | ≥ 1,5 nm / bibliotheekpool | ≥ 25 ul / bibliotheekpool |
Naast concentratie en totale hoeveelheid is ook een geschikt piekpatroon vereist.
OPMERKING: Lane-sequencing van bibliotheken met lage diversiteit vereist Phix Spike-in om een robuuste basenaanvallen te garanderen.
We raden aan om pre-gepoolde bibliotheken in te dienen als monsters. Als u bmkgene nodig heeft om bibliotheekpools uit te voeren, raadpleeg dan de
Bibliotheekvereisten voor gedeeltelijke rijstrooksequencing.
Bibliotheekgrootte (piekkaart)
De belangrijkste piek moet binnen 300-450 bp zijn.
Bibliotheken moeten een enkele hoofdpiek hebben, geen adapterverontreiniging en geen primer -dimeren.
Serviceworkflow




Bibliotheek QC -rapport
Een rapport over de kwaliteit van de bibliotheek wordt verstrekt vóór sequencing, het beoordelen van bibliotheekbedrag en fragmentatie.
Sequencing QC -rapport
Tabel 1. Statistieken over sequentiegegevens.
| Voorbeeld -ID | BMKID | Raw leest | Ruwe gegevens (BP) | Schone lezingen (%) | Q20 (%) | Q30 (%) | GC (%) |
| C_01 | BMK_01 | 22.870,120 | 6.861.036.000 | 96.48 | 99.14 | 94.85 | 36.67 |
| C_02 | BMK_02 | 14.717.867 | 4.415,360.100 | 96,00 | 98,95 | 93.89 | 37.08 |
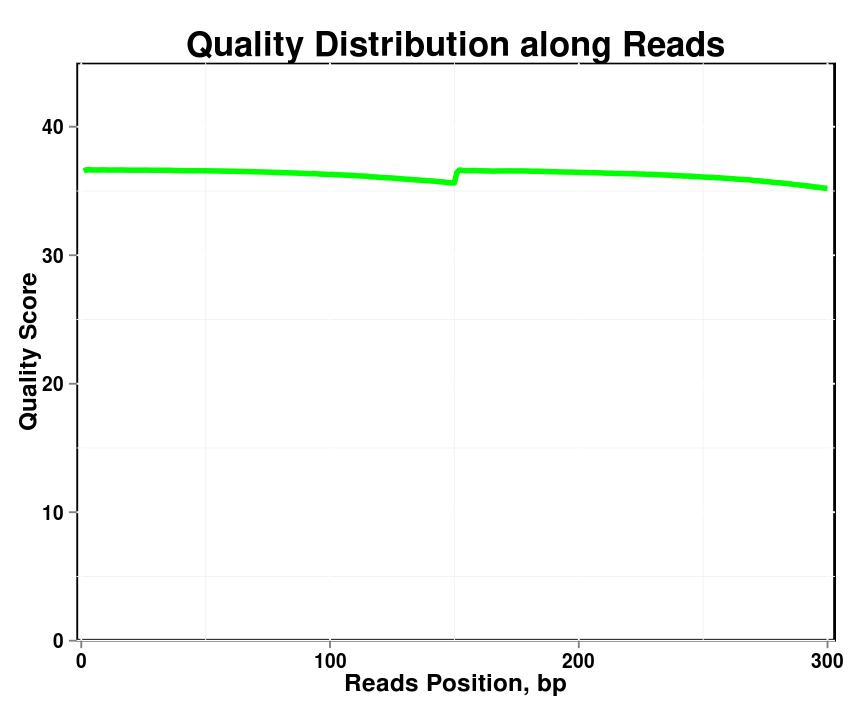
Figuur 1. Kwaliteitsverdeling langs leest in elk monster
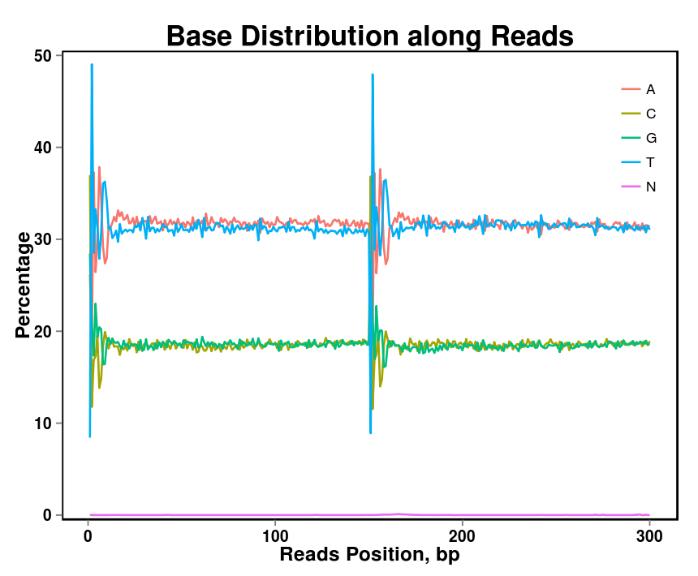
Figuur 2. Basisinhoudverdeling
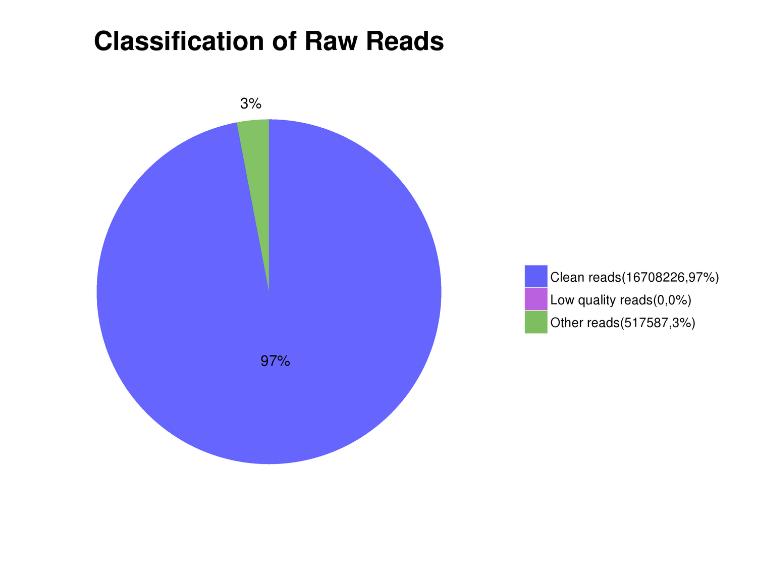
Figuur 3. Verdeling van leesinhoud in sequentiegegevens