BMKMANU S1000 Ruimtelijk transcriptoom
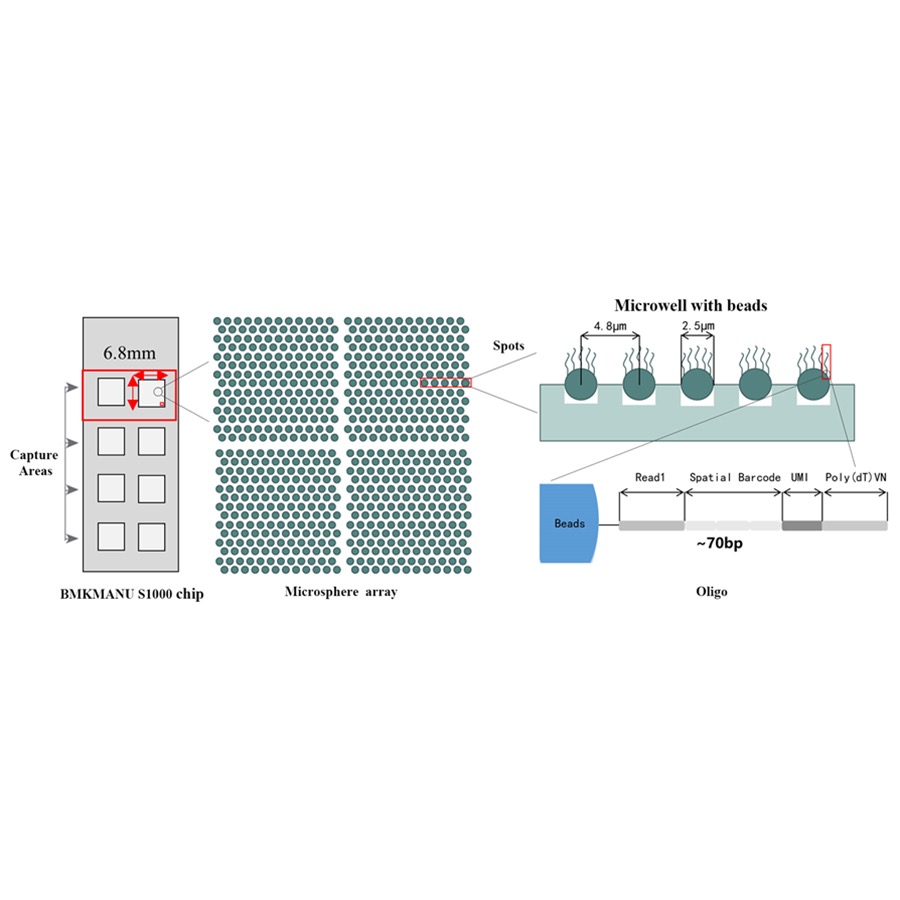
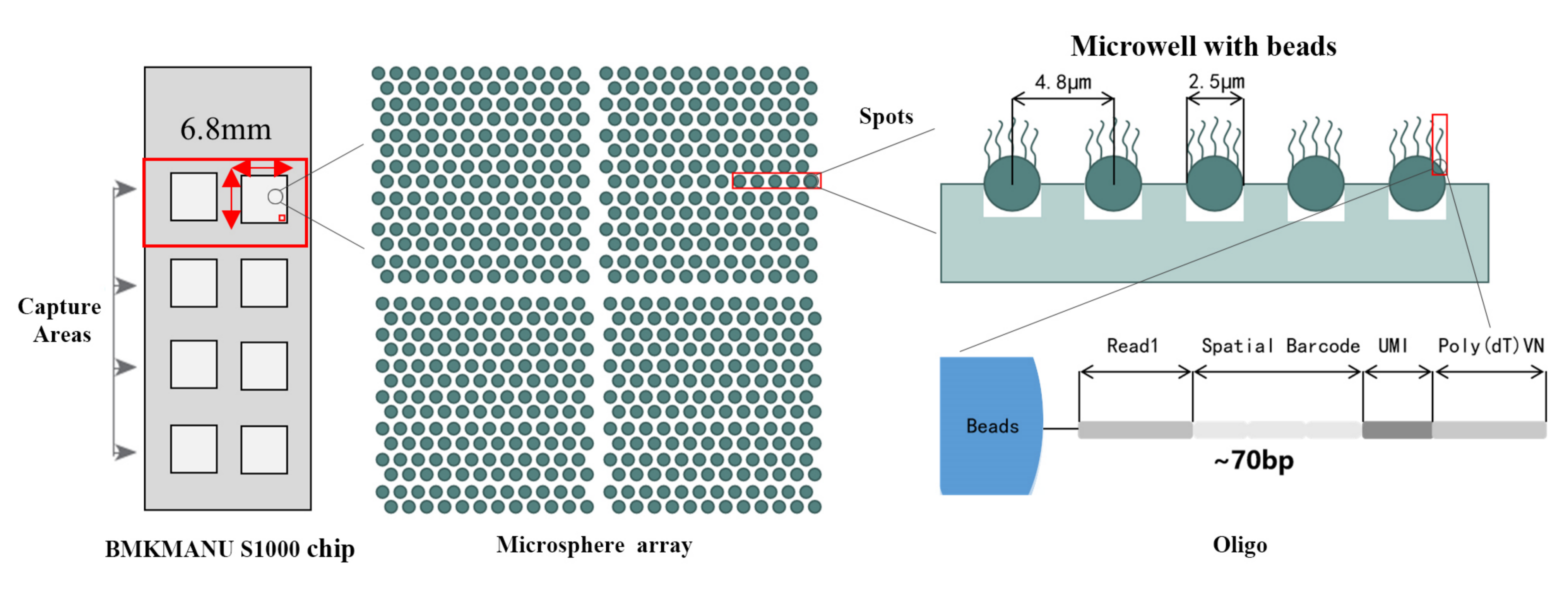
BMKMANU S1000 Ruimtelijk transcriptoom technisch schema
Functies
● Resolutie: 5 µM
● Spotdiameter: 2,5 µM
● Aantal plekken: ongeveer 2 miljoen
● 3 mogelijke formaten voor opnamegebieden: 6,8 mm * 6,8 mm, 11 mm * 11 mm of 15 mm * 20 mm
● Elke kraal met streepjescode is voorzien van primers die uit 4 secties bestaan:
poly(dT)-staart voor mRNA-priming en cDNA-synthese
Unieke Moleculaire Identificatie (UMI) om amplificatiebias te corrigeren
Ruimtelijke streepjescode
Bindende sequentie van gedeeltelijk gelezen 1-sequencingprimer
● H&E en fluorescerende kleuring van coupes
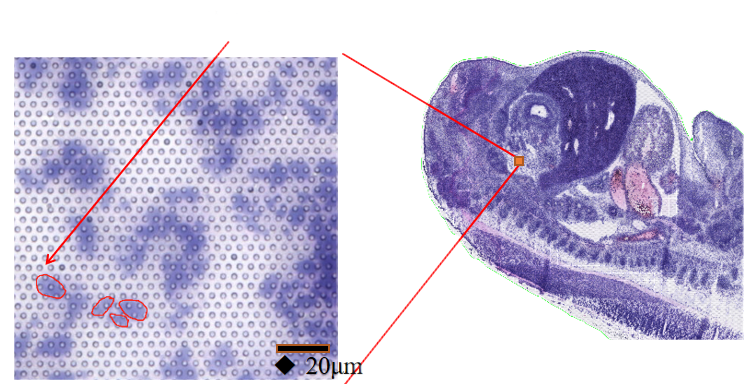
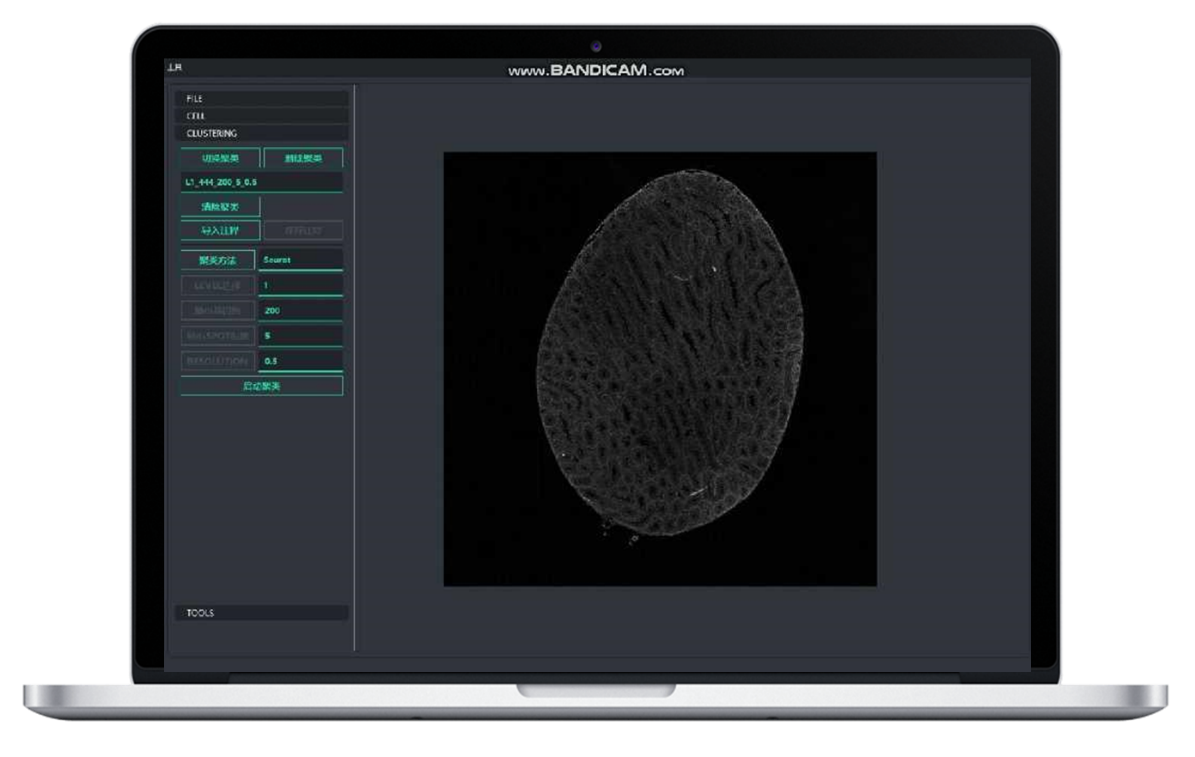
● Mogelijkheid tot gebruiktechnologie voor celsegmentatie: integratie van H&E-kleuring, fluorescerende kleuring en RNA-sequencing om de grenzen van elke cel te bepalen en genexpressie correct aan elke cel toe te wijzen.
Voordelen van BMKMANU S1000
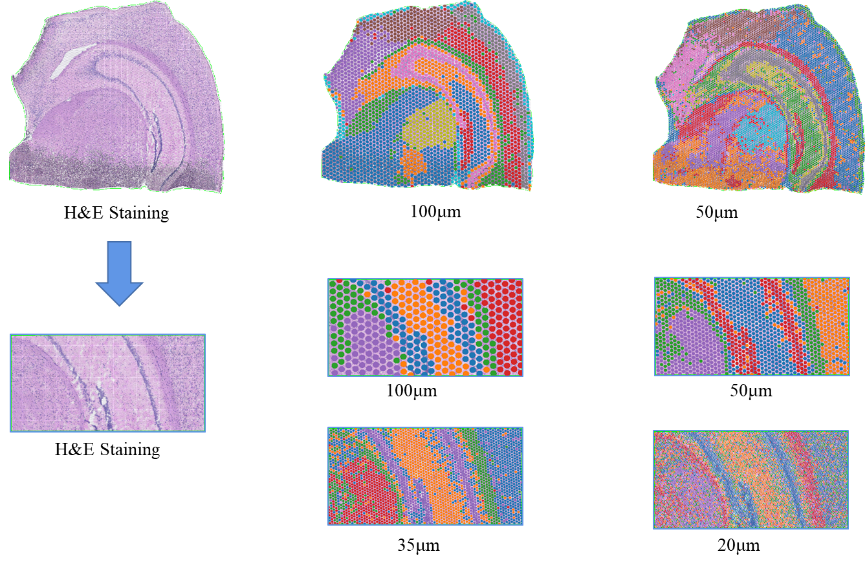
●Subcellulaire resolutie: Elk opnamegebied bevatte >2 miljoen ruimtelijke streepjescodevlekken met een diameter van 2,5 µm en een afstand van 5 µm tussen de vlekcentra, waardoor ruimtelijke transcriptoomanalyse met subcellulaire resolutie (5 µm) mogelijk was.
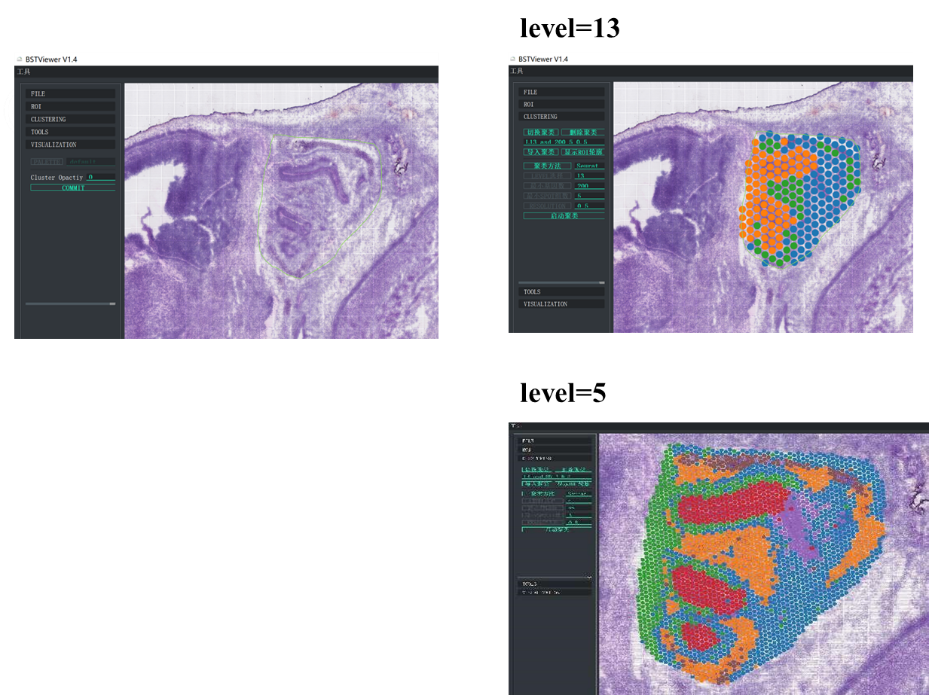
●Resolutieanalyse op meerdere niveaus:Flexibele analyse op meerdere niveaus, variërend van 100 μm tot 5 μm, om diverse weefselkenmerken met optimale resolutie op te lossen.
●Mogelijkheid om “Drie in één glaasje” celsegmentatietechnologie te gebruiken:Door fluorescentiekleuring, H&E-kleuring en RNA-sequencing op één objectglaasje te combineren, maakt ons "drie-in-één" analyse-algoritme de identificatie van celgrenzen mogelijk voor daaropvolgende celgebaseerde transcriptomics.
●Compatibel met meerdere sequencingplatforms: Zowel NGS als long-read sequencing beschikbaar.
●Flexibel ontwerp van 1-8 actieve opnamegebieden: De grootte van het opnamegebied is flexibel, waardoor het mogelijk is om 3 formaten te gebruiken (6,8 mm * 6,8 mm, 11 mm * 11 mm en 15 mm * 20 mm)
●One-stop-service: Het integreert alle op ervaring en vaardigheden gebaseerde stappen, inclusief cryo-sectie, kleuring, weefseloptimalisatie, ruimtelijke streepjescodes, bibliotheekvoorbereiding, sequencing en bio-informatica.
●Uitgebreide bio-informatica en gebruiksvriendelijke visualisatie van resultaten:Het pakket bevat 29 analyses en meer dan 100 cijfers van hoge kwaliteit, gecombineerd met het gebruik van in eigen huis ontwikkelde software om celsplitsing en spotclustering te visualiseren en aan te passen.
●Aangepaste data-analyse en visualisatie: beschikbaar voor verschillende onderzoeksaanvragen
●Hooggekwalificeerd technisch team: met ervaring in meer dan 250 weefseltypen en meer dan 100 soorten, waaronder mensen, muizen, zoogdieren, vissen en planten.
●Realtime updates voor het hele project: met volledige controle over de experimentele voortgang.
●Optionele gezamenlijke analyse met mRNA-sequencing van één cel
Servicespecificaties
|
Steekproef Vereisten
| Bibliotheek |
Sequentiestrategie
| Gegevens aanbevolen | Kwaliteitscontrole |
| OCT-ingebedde cryomonsters, 3 blokken per monster | S1000 cDNA-bibliotheek | Illumina PE150 (andere platforms beschikbaar) | 100K PE-metingen per 100 uM (60-150 GB) | RIN>7 |
Voor meer informatie over begeleiding bij monstervoorbereiding en serviceworkflow kunt u gerust contact opnemen met eenBMKGENE-expert
Servicewerkstroom
In de monstervoorbereidingsfase wordt een eerste bulk-RNA-extractieproef uitgevoerd om ervoor te zorgen dat RNA van hoge kwaliteit kan worden verkregen. In de weefseloptimalisatiefase worden de coupes gekleurd en gevisualiseerd en worden de permeabilisatieomstandigheden voor de afgifte van mRNA uit het weefsel geoptimaliseerd. Het geoptimaliseerde protocol wordt vervolgens toegepast tijdens de bibliotheekconstructie, gevolgd door sequencing en data-analyse.
De volledige serviceworkflow omvat realtime updates en klantbevestigingen om een responsieve feedbacklus te behouden en een soepele projectuitvoering te garanderen.
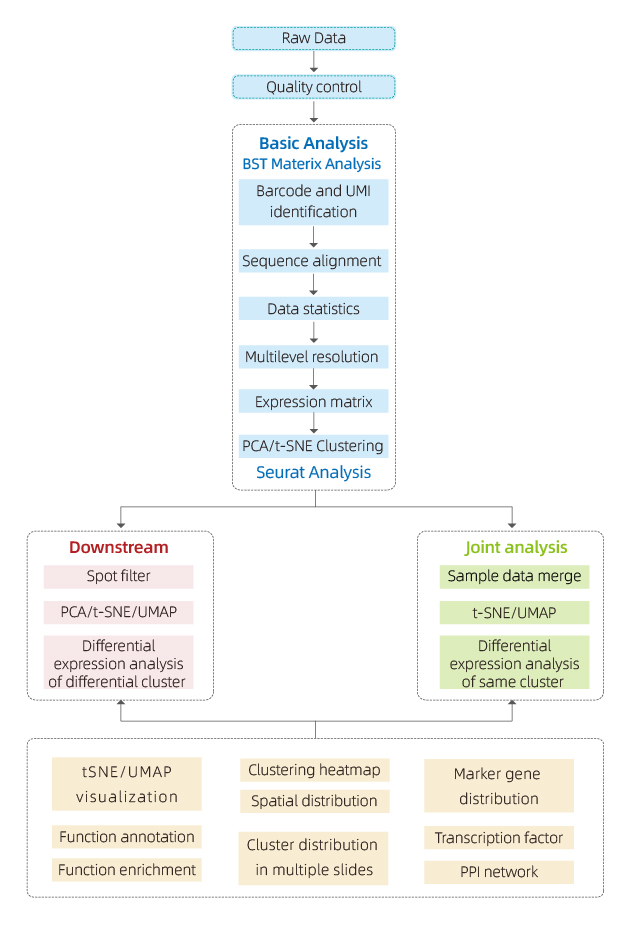
De door BMKMANU S1000 gegenereerde gegevens worden geanalyseerd met behulp van de software “BSTMatrix”, die onafhankelijk is ontworpen door BMKGENE en een genexpressiematrix genereert. Van daaruit wordt een standaardrapport gegenereerd met gegevenskwaliteitscontrole, analyse van de interne steekproef en analyse tussen groepen.
● Gegevenskwaliteitscontrole:
Gegevensuitvoer en distributie van kwaliteitsscores
Gendetectie per spot
Weefseldekking
● Analyse van binnenmonsters:
Genenrijkdom
Spotclustering, inclusief analyse van kleinere dimensies
Differentiële expressieanalyse tussen clusters: identificatie van markergenen
Functionele annotatie en verrijking van markergenen
● Intergroepsanalyse:
Opnieuw combineren van vlekken uit beide monsters (bijv. ziek en controle) en opnieuw clusteren
Identificatie van markergenen voor elk cluster
Functionele annotatie en verrijking van markergenen
Differentiële expressie van hetzelfde cluster tussen groepen
Daarnaast heeft BMKGENE “BSTViewer” ontwikkeld, een gebruiksvriendelijke tool waarmee de gebruiker de genexpressie kan visualiseren en clustering met verschillende resoluties kan spotten.
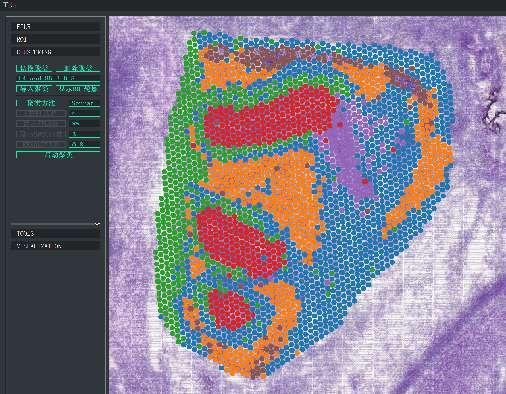
BMKGene ontwikkelde software voor gebruiksvriendelijke visualisatie
BSTViewer-spotclustering met resolutie op meerdere niveaus
BSTCellViewer: automatische en handmatige celsplitsing
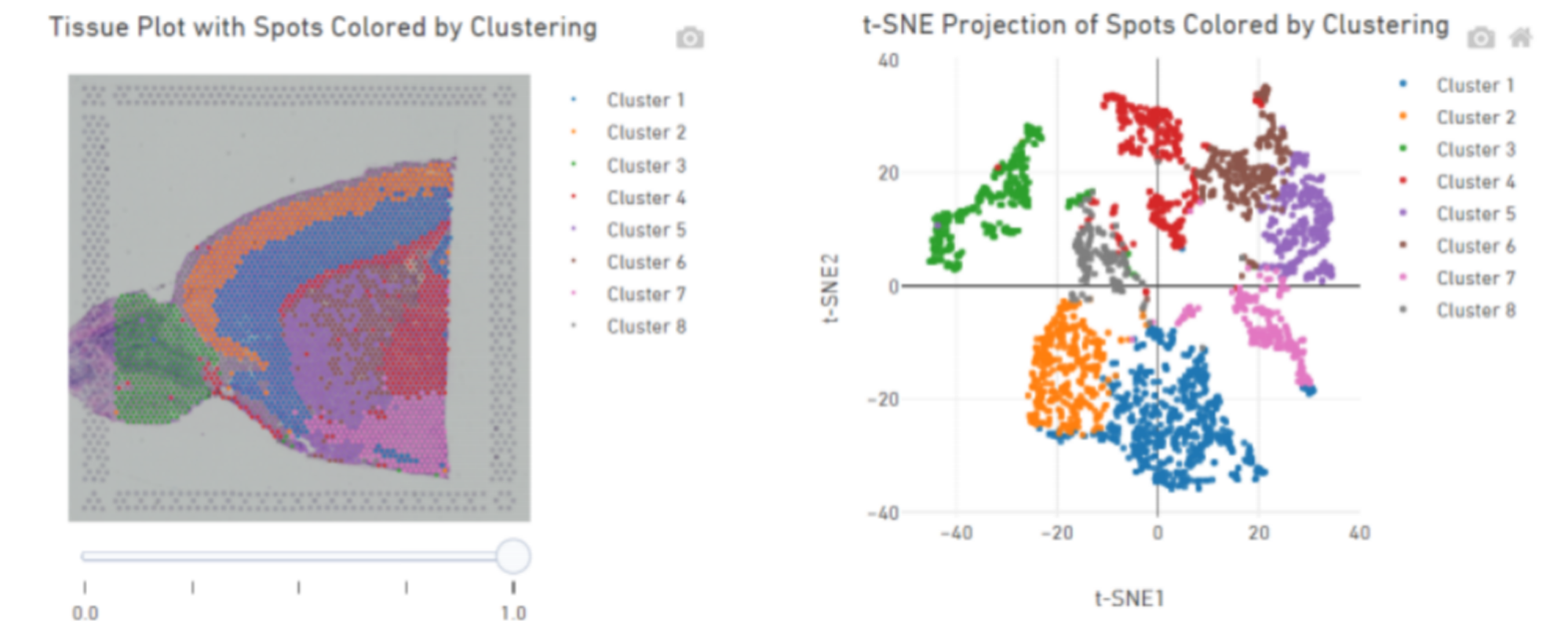
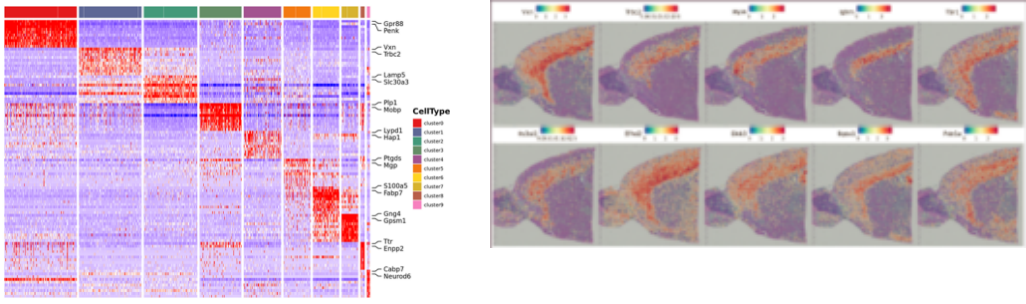
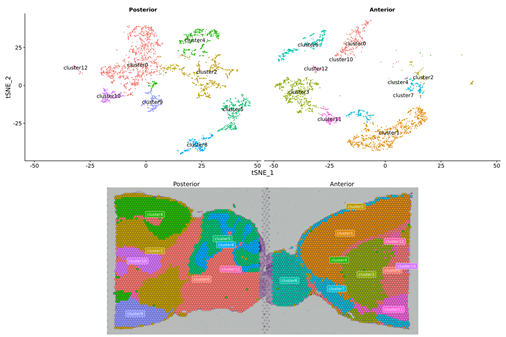
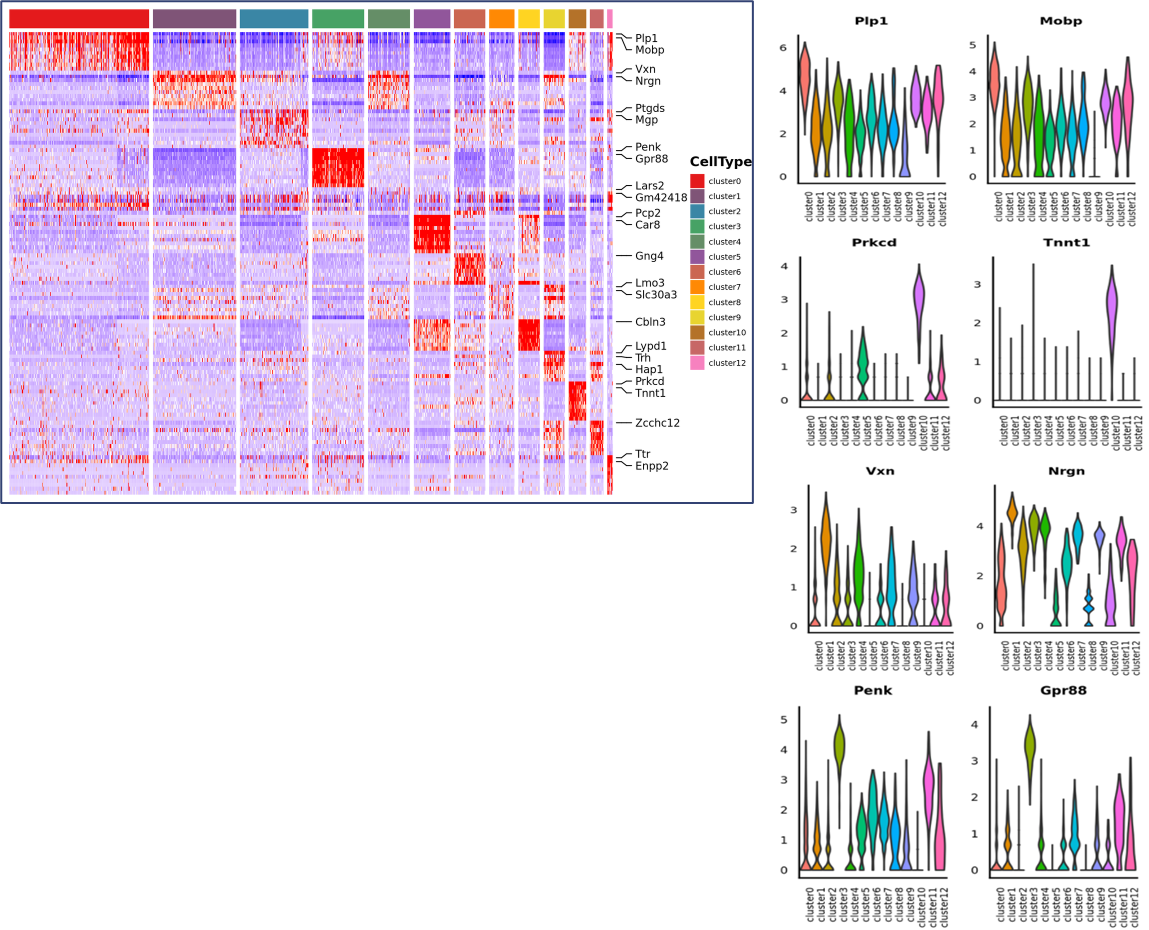
Analyse van binnenmonsters
Spotclustering:
Identificatie van markergenen en ruimtelijke distributie:
Intergroepsanalyse
Gegevenscombinatie van beide groepen en hercluster:
Markergenen van nieuwe clusters:
Ontdek de vooruitgang die wordt mogelijk gemaakt door de ruimtelijke transcriptomics-diensten van BMKGene met de BMKManu S1000-technologie in deze uitgelichte publicatie:
Song, X. et al. (2023) 'Ruimtelijke transcriptomics onthullen door licht geïnduceerde chlorenchymcellen die betrokken zijn bij het bevorderen van scheutregeneratie in tomatencallus',Proceedings van de National Academy of Sciences van de Verenigde Staten van Amerika, 120(38), p. e2310163120. doi: 10.1073/pnas.2310163120
Jij, Y. et al. (2023) 'Systematische vergelijking van op sequencing gebaseerde ruimtelijke transcriptomische methoden',bioRxiv, P. 2023.12.03.569744. doi: 10.1101/2023.12.03.569744.