BMKMANU S1000 स्थानिय ट्रान्सक्रिप्टोम
BMKMANU S1000 स्थानिय ट्रान्सक्रिप्टोम प्राविधिक योजना
सुविधाहरू
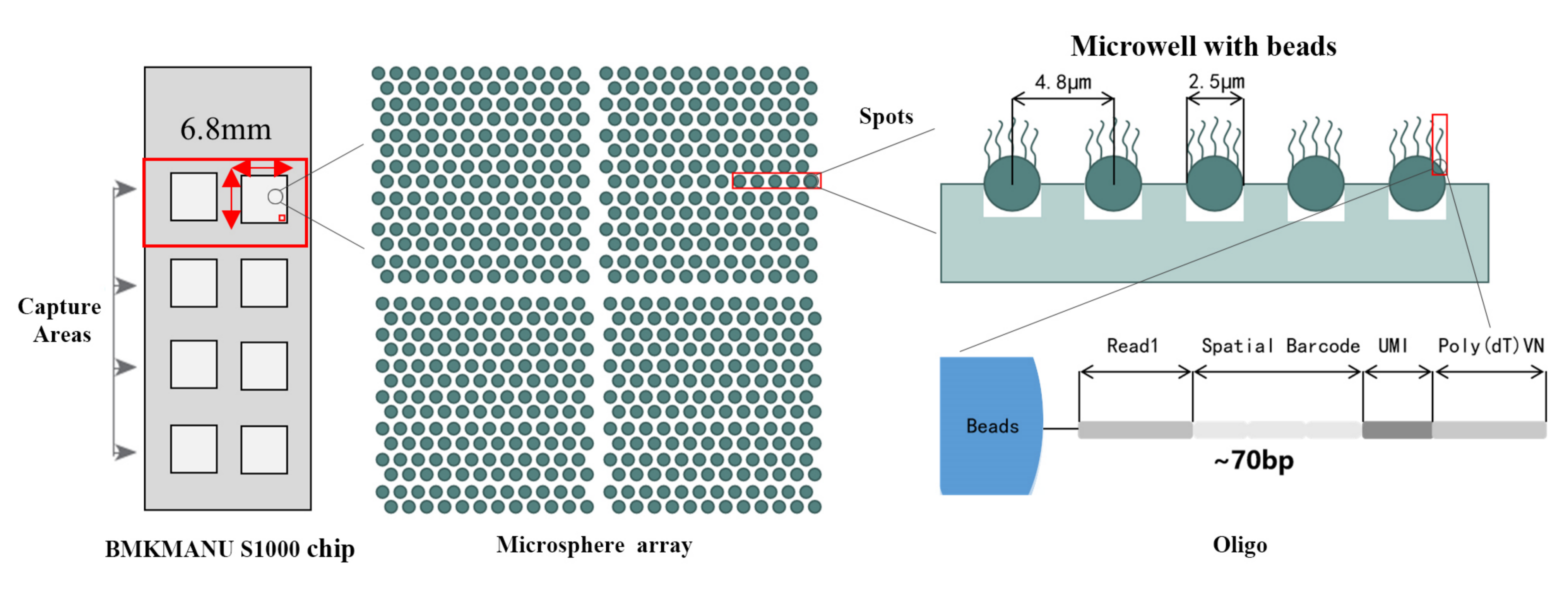
● रिजोल्युसन: 5 µM
● स्पट व्यास: 2.5 µM
● स्पटहरूको संख्या: लगभग 2 मिलियन
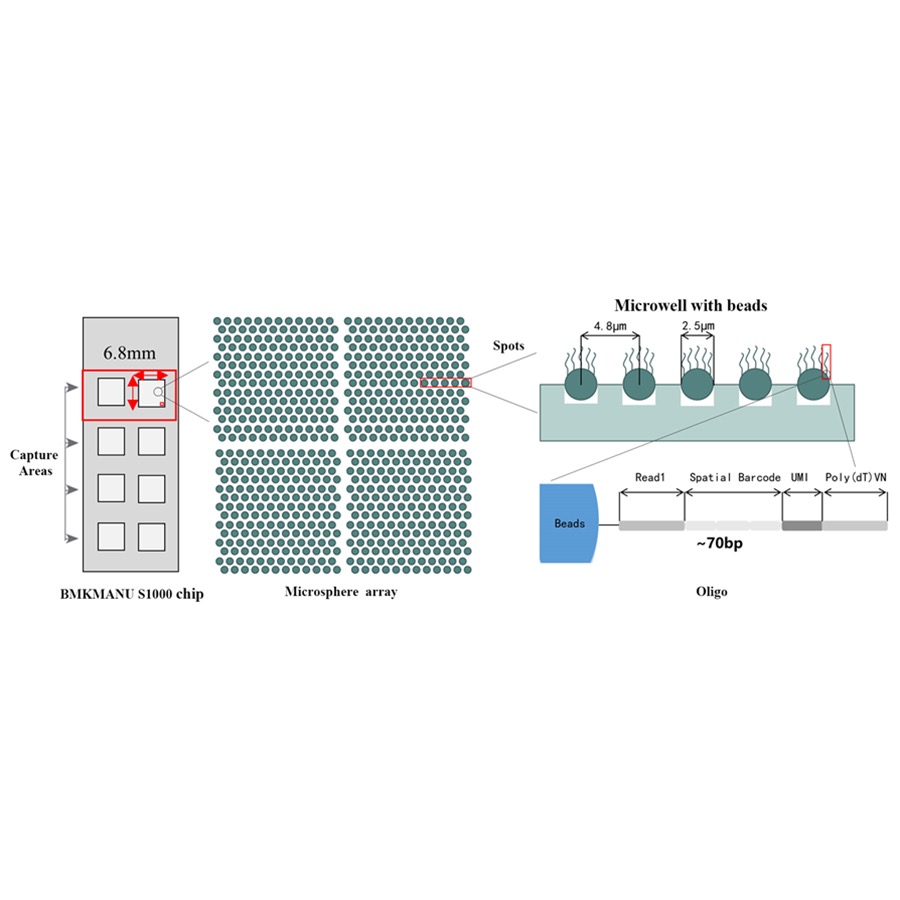
● 3 सम्भावित क्याप्चर क्षेत्र ढाँचाहरू: 6.8 मिमी * 6.8 मिमी, 11 मिमी * 11 मिमी वा 15 मिमी * 20 मिमी
● प्रत्येक बारकोड गरिएको मोती 4 खण्डहरू मिलेर बनेको प्राइमरहरूले भरिएको हुन्छ:
एमआरएनए प्राइमिङ र सीडीएनए संश्लेषणका लागि पाली(डीटी) टेल
प्रवर्धन पूर्वाग्रह सुधार गर्न अद्वितीय आणविक पहिचानकर्ता (UMI)
स्थानिय बारकोड
आंशिक पढिएको १ अनुक्रम प्राइमरको बाइन्डिङ अनुक्रम
● H&E र खण्डहरूको फ्लोरोसेन्ट दाग
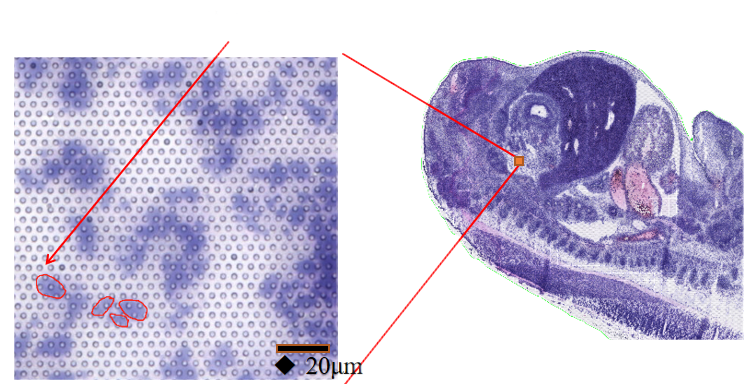
● प्रयोग गर्ने सम्भावनासेल विभाजन प्रविधि: H&E स्टेनिङ, फ्लोरोसेन्ट स्टेनिङ, र RNA अनुक्रमणिकाको एकीकरण प्रत्येक कक्षको सीमा निर्धारण गर्न र प्रत्येक कोषमा जीन अभिव्यक्ति सही रूपमा तोक्न।
BMKMANU S1000 का फाइदाहरू
●उप-सेलुलर रिजोल्युसन: प्रत्येक क्याप्चर क्षेत्रमा 2.5 µm व्यास र स्पट सेन्टरहरू बीच 5 µm को स्पेसिङका साथ>2 मिलियन स्थानिक बारकोड गरिएका स्पटहरू समावेश छन्, जसले उप-सेलुलर रिजोल्युसन (5 µm) को साथ स्थानिक ट्रान्सक्रिप्टोम विश्लेषण सक्षम पार्दै।
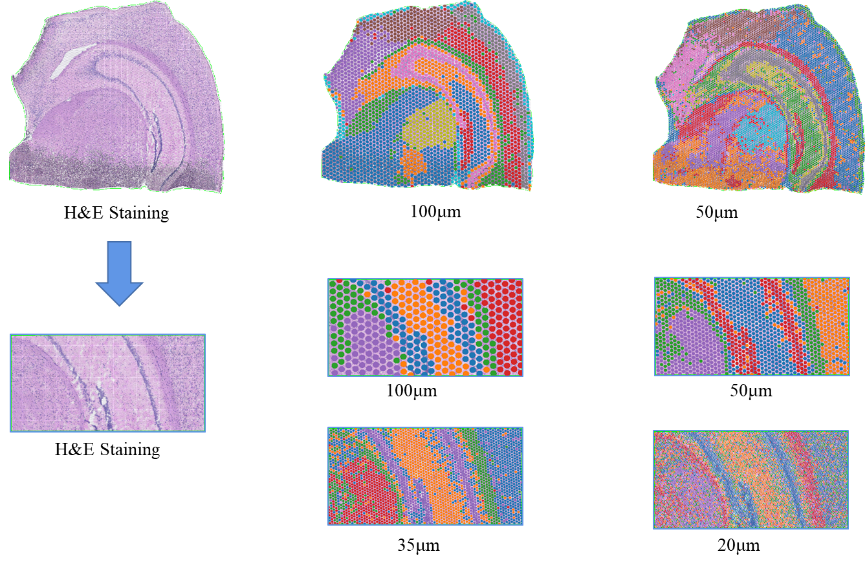
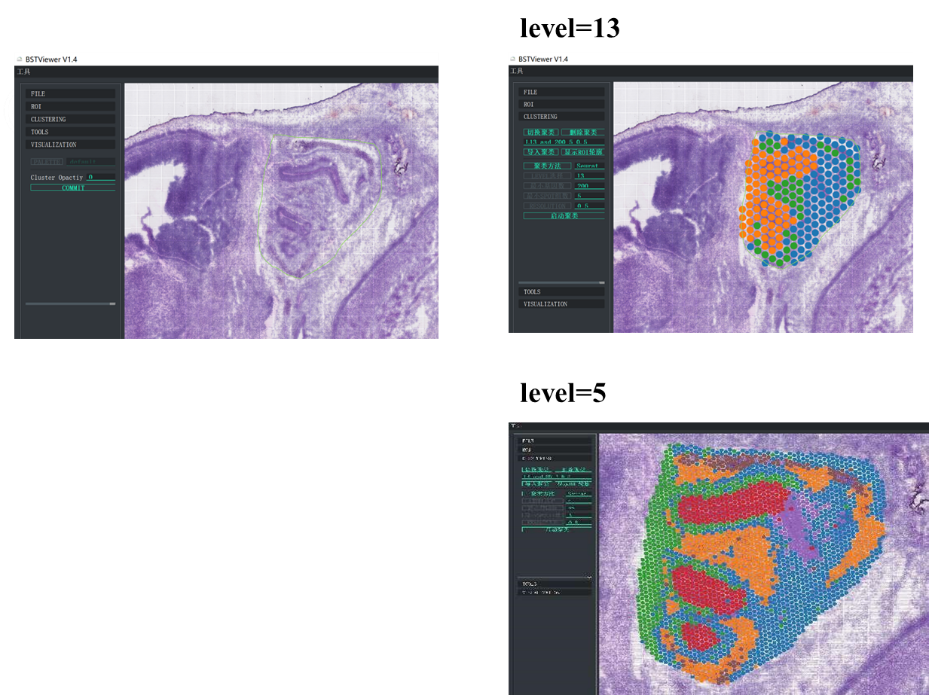
●बहु-स्तरीय संकल्प विश्लेषण:इष्टतम रिजोल्युसनमा विविध ऊतक सुविधाहरू समाधान गर्न 100 μm देखि 5 μm सम्मको लचिलो बहु-स्तर विश्लेषण।
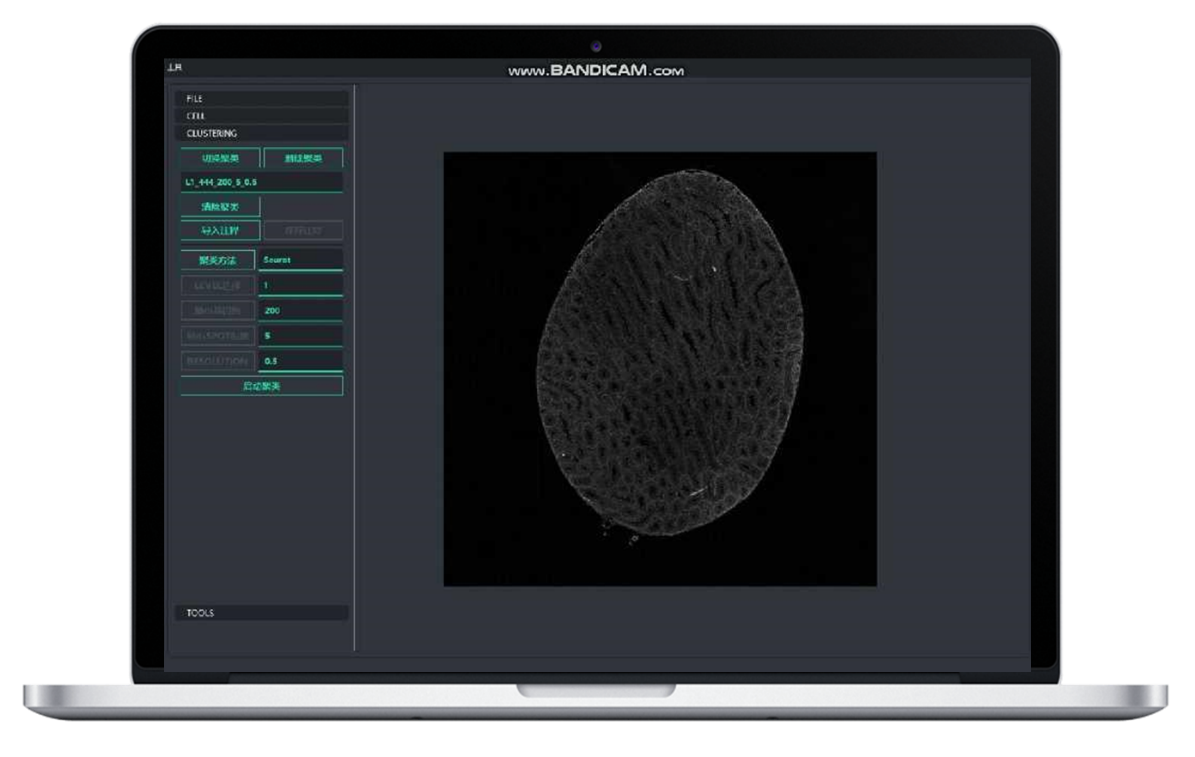
● "एक स्लाइडमा तीन" सेल विभाजन प्रविधि प्रयोग गर्ने सम्भावना:एकल स्लाइडमा फ्लोरोसेन्स स्टेनिङ, H&E स्टेनिङ, र RNA सिक्वेन्सिङको संयोजन गर्दै, हाम्रो "थ्री-इन-वन" विश्लेषण एल्गोरिदमले सेल-आधारित ट्रान्सक्रिप्टोमिक्सका लागि सेल सीमाहरू पहिचान गर्न सशक्त बनाउँछ।
●बहु अनुक्रमिक प्लेटफर्महरूसँग उपयुक्त: दुबै NGS र लामो-पढ्ने अनुक्रम उपलब्ध छ।
●1-8 सक्रिय क्याप्चर क्षेत्रको लचिलो डिजाइन: क्याप्चर क्षेत्रको आकार लचिलो छ, 3 ढाँचाहरू (6.8 मिमी * 6.8 मिमी।, 11 मिमी * 11 मिमी र 15 मिमी * 20 मिमी) प्रयोग गर्न सम्भव छ।
●एक-स्टप सेवा: यसले क्रायो-सेक्शनिङ, स्टेनिङ, टिस्यु अप्टिमाइजेसन, स्पेसियल बारकोडिङ, लाइब्रेरी तयारी, सिक्वेन्सिङ, र बायोइन्फर्मेटिक्सलगायत सबै अनुभव र सीपमा आधारित चरणहरूलाई एकीकृत गर्छ।
●व्यापक जैव सूचना विज्ञान र परिणामहरूको प्रयोगकर्ता-अनुकूल दृश्य:प्याकेजले सेल विभाजन र स्पट क्लस्टरिङको कल्पना र अनुकूलन गर्न इनहाउस विकसित सफ्टवेयरको प्रयोगसँग मिलेर २९ विश्लेषणहरू र १००+ उच्च गुणस्तरका आंकडाहरू समावेश गर्दछ।
●अनुकूलित डाटा विश्लेषण र दृश्य: विभिन्न अनुसन्धान अनुरोधहरूको लागि उपलब्ध
●उच्च दक्ष प्राविधिक टोली: मानव, मुसा, स्तनपायी, माछा र बिरुवाहरू सहित 250 भन्दा बढी टिश्यू प्रकारहरू र 100+ प्रजातिहरूमा अनुभवको साथ।
●सम्पूर्ण परियोजनामा वास्तविक-समय अद्यावधिकहरू: प्रयोगात्मक प्रगतिको पूर्ण नियन्त्रणको साथ।
●एकल-सेल mRNA अनुक्रमको साथ वैकल्पिक संयुक्त विश्लेषण
सेवा विशिष्टताहरू
|
नमूना आवश्यकताहरू
| पुस्तकालय |
अनुक्रम रणनीति
| Data सिफारिस गर्नुभयो | गुणस्तर नियन्त्रण |
| OCT-इम्बेडेड क्रायो नमूनाहरू, प्रति नमूना 3 ब्लकहरू | S1000 cDNA पुस्तकालय | Illumina PE150 (अन्य प्लेटफार्महरू उपलब्ध छन्) | 100K PE प्रति 100 uM पढिन्छ (६०-१५० जीबी) | RIN>7 |
नमूना तयारी मार्गदर्शन र सेवा कार्यप्रवाहको बारेमा थप विवरणहरूको लागि, कृपया एकसँग कुरा गर्न नहिचकिचाउनुहोस्BMKGENE विशेषज्ञ
सेवा कार्य प्रवाह
नमूना तयारी चरणमा, उच्च गुणस्तरको आरएनए प्राप्त गर्न सकिने सुनिश्चित गर्न प्रारम्भिक बल्क आरएनए निकासी परीक्षण गरिन्छ। टिस्यु अप्टिमाइजेसन चरणमा खण्डहरू दाग र दृश्यात्मक हुन्छन् र टिश्यूबाट एमआरएनए रिलिजको लागि पारगम्यीकरण अवस्थाहरू अनुकूलित हुन्छन्। अनुकूलित प्रोटोकल त्यसपछि पुस्तकालय निर्माण समयमा लागू हुन्छ, अनुक्रम र डेटा विश्लेषण पछि।
पूर्ण सेवा कार्यप्रवाहमा वास्तविक-समय अद्यावधिकहरू र क्लाइन्ट पुष्टिकरणहरू समावेश छन् उत्तरदायी प्रतिक्रिया लूप कायम राख्न, सहज परियोजना कार्यान्वयन सुनिश्चित गर्दै।
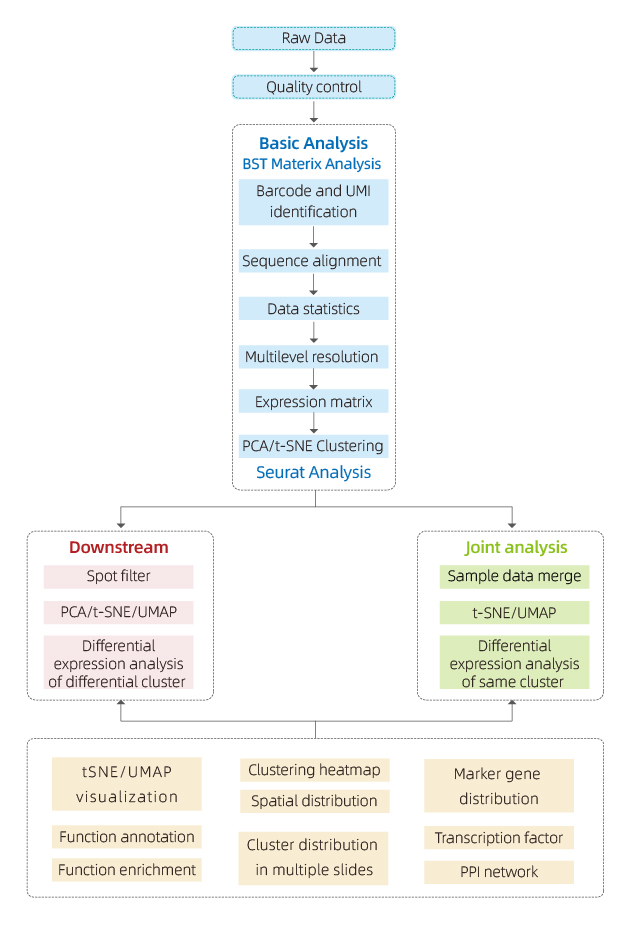
BMKMANU S1000 द्वारा उत्पन्न डाटा "BSTMatrix" सफ्टवेयर प्रयोग गरेर विश्लेषण गरिन्छ, जुन BMKGENE द्वारा स्वतन्त्र रूपमा डिजाइन गरिएको हो, जीन अभिव्यक्ति म्याट्रिक्स उत्पन्न गर्दछ। त्यहाँबाट, एक मानक रिपोर्ट उत्पन्न हुन्छ जसमा डेटा गुणस्तर नियन्त्रण, भित्री-नमूना विश्लेषण र अन्तर-समूह विश्लेषण समावेश हुन्छ।
● डाटा गुणस्तर नियन्त्रण:
डाटा आउटपुट र गुणस्तर स्कोर वितरण
प्रति स्थान जीन पत्ता लगाउने
टिश्यु कभरेज
● भित्री-नमूना विश्लेषण:
जीन समृद्धि
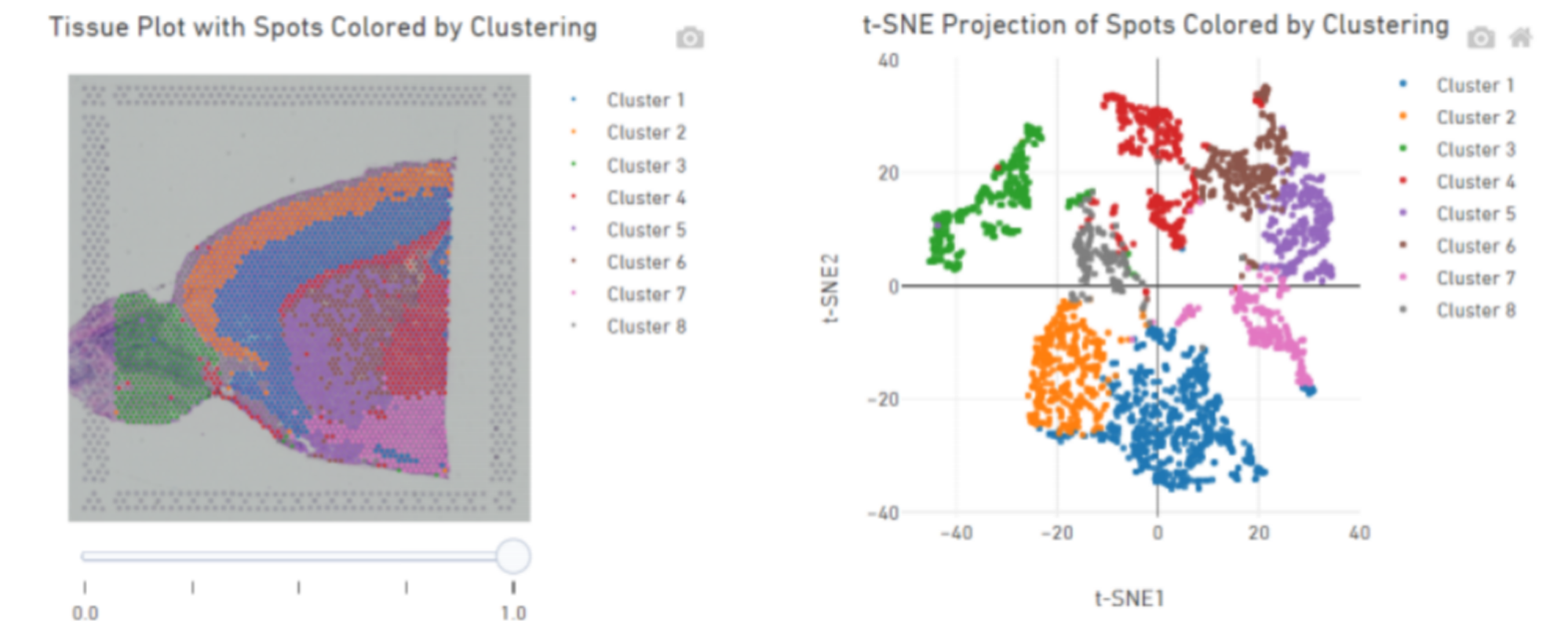
घटाइएको आयाम विश्लेषण सहित स्पट क्लस्टरिङ
क्लस्टरहरू बीचको भिन्न अभिव्यक्ति विश्लेषण: मार्कर जीनहरूको पहिचान
कार्यात्मक एनोटेशन र मार्कर जीन को संवर्धन
● अन्तर-समूह विश्लेषण:
दुबै नमूनाहरू (जस्तै रोग र नियन्त्रण) र पुन: क्लस्टरबाट स्पटहरूको पुन: संयोजन
प्रत्येक क्लस्टरको लागि मार्कर जीनहरूको पहिचान
कार्यात्मक एनोटेशन र मार्कर जीन को संवर्धन
समूहहरू बीच एउटै क्लस्टरको भिन्न अभिव्यक्ति
थप रूपमा, BMKGENE विकसित गरिएको "BSTViewer" एक प्रयोगकर्ता-अनुकूल उपकरण हो जसले प्रयोगकर्तालाई विभिन्न रिजोल्युसनहरूमा जीन अभिव्यक्ति र स्पट क्लस्टरिङको कल्पना गर्न सक्षम बनाउँछ।
BMKGene ले प्रयोगकर्ता मैत्री दृश्यका लागि सफ्टवेयर विकास गर्यो
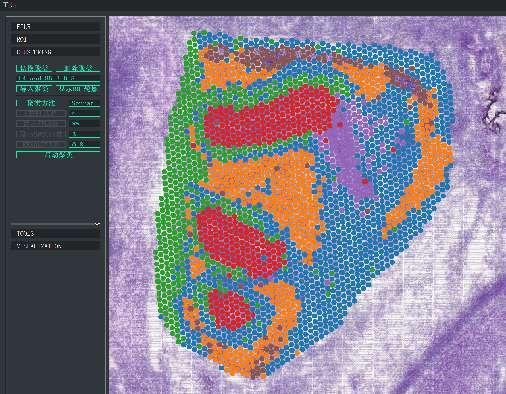
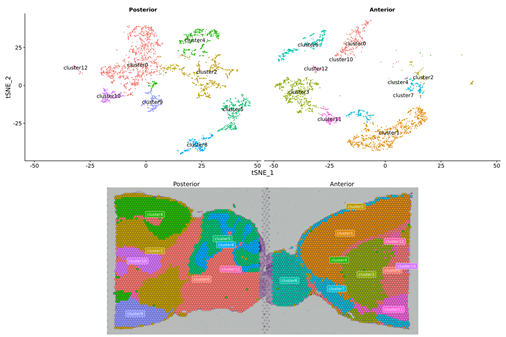
बहु-स्तर रिजोल्युसनमा BSTViewer स्पट क्लस्टरिङ
BSTCellViewer: स्वचालित र म्यानुअल सेल विभाजन
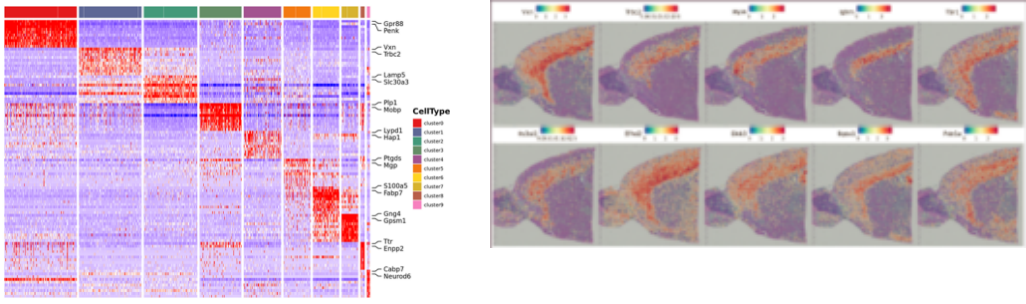
भित्री-नमूना विश्लेषण
स्पट क्लस्टरिङ:
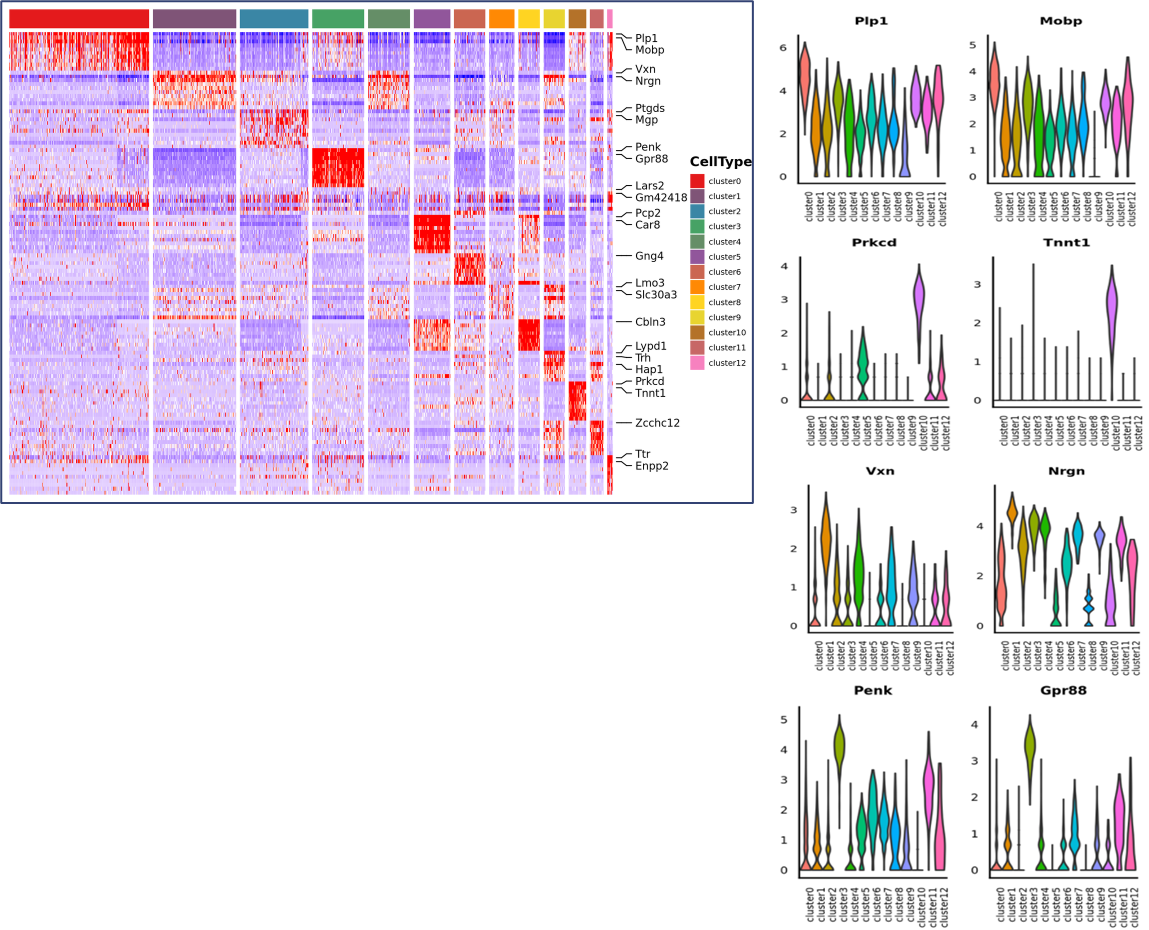
मार्कर जीन पहिचान र स्थानिय वितरण:
अन्तर-समूह विश्लेषण
दुबै समूह र पुन: क्लस्टरबाट डेटा संयोजन:
नयाँ क्लस्टरहरूको मार्कर जीन:
यस विशेष प्रकाशनमा BMKManu S1000 टेक्नोलोजीको साथ BMKGene को स्थानिय ट्रान्सक्रिप्टोमिक्स सेवाहरूद्वारा सहजीकरण गरिएका प्रगतिहरू अन्वेषण गर्नुहोस्:
गीत, X. et al। (2023) 'स्थानिक ट्रान्सक्रिप्टोमिक्सले टमाटर कलसमा गोलीको पुनरुत्थान प्रवर्द्धन गर्न संलग्न प्रकाश-प्रेरित क्लोरेन्काइमा कोशिकाहरू प्रकट गर्दछ',संयुक्त राज्य अमेरिका को नेशनल एकेडेमी अफ साइन्स को कार्यवाही, 120(38), p। e2310163120। doi: 10.1073/pnas.2310163120
You, Y. et al. (2023) 'अनुक्रम आधारित स्थानिय ट्रान्सक्रिप्टोमिक विधिहरूको व्यवस्थित तुलना',bioRxiv, p. २०२३.१२.०३.५६९७४४। doi: 10.1101/2023.12.03.569744।