hi-c ကိုအခြေခံ Chromatin အပြန်အလှန်ဆက်သွယ်မှု
ဝန်ဆောင်မှုအင်္ဂါရပ်များ
● Illumina Novasq ကို pe 150 နှင့်အတူ sequencing ။
● 0 န်ဆောင်မှုသည်သျှူးနမူနာများ, ပုံမှန်အားဖြင့်အနမ်းယူထားသောအက်ဆစ်များအစားပုံမှန် dina-protein နှင့်ဆက်နွယ်မှုကိုထိန်းသိမ်းရန်နှင့် DNA-protein interaction များကိုထိန်းသိမ်းရန်နှင့် DNA-protein interaction များကိုထိန်းသိမ်းရန်အတွက်တစ်သျှူးနမူနာများလိုအပ်သည်။
● Hi-C စမ်းသပ်မှုမှာ Biotin နှင့်အတူစေးကပ်၏ကန့်သတ်ခြင်းနှင့်အဆုံးပြုပြင်ခြင်းတို့ပါဝင်သည်။ ထို့နောက် DNA ကို cheptavidin ပုတီးနှင့်အတူဆွဲထုတ်ခြင်းနှင့်နောက်ဆက်တွဲစာကြည့်တိုက်ကြိုတင်ပြင်ဆင်မှုများအတွက်သန့်စင်။
ဝန်ဆောင်မှုအားသာချက်များ
●အကောင်းဆုံးကန့်သတ် inzyme ဒီဇိုင်း: 93% တရားဝင်အပြန်အလှန်ဆက်သွယ်မှုအားလုံးအတွက်မျိုးစိတ်များအပေါ်မြင့်မားသောမျိုးစိတ်များအပေါ်မြင့်မားသော hi-c ထိရောက်မှုကိုသေချာစေရန်။
●ကျယ်ပြန့်သောကျွမ်းကျင်မှုနှင့်ထုတ်ဝေမှတ်တမ်းများ:BMKGENENENE သည်မျိုးစိတ် 800 နှင့်မူပိုင်ခွင့်အမျိုးမျိုးမှ 2000 hi-c sequencing စီမံကိန်းများနှင့်အတူအတွေ့အကြုံများစွာရှိသည်။ 100 ကျော်သည် 900 ကျော်စုဆောင်းမှုသက်ရောက်မှုအချက်နှင့်အတူထုတ်ဝေမှုအမှု 100 ကျော်။
●ကျွမ်းကျင်စွာကျွမ်းကျင်သော Bioinformatics အသင်း -အိမ်တွင်းမူပိုင်ခွင့်များနှင့်ဆော့ဖ်ဝဲမူပိုင်ခွင့်များနှင့်အတူ Hi-C စမ်းသပ်မှုများနှင့်ဒေတာခွဲခြမ်းစိတ်ဖြာခြင်းနှင့်ကိုယ်ပိုင်ဖွံ့ဖြိုးပြီး visualization data software အတွက်မူပိုင်ခွင့်ရှိသည်။
●အရောင်းအ 0 င် Post ပံ့ပိုးမှု -ကျွန်ုပ်တို့၏ကတိက 0 တ်သည်စီမံကိန်းပြီးစီးခြင်းထက် 3 လအကြာရောင်း 0 ယ်သည့် 0 န်ဆောင်မှုကာလနှင့်အတူစီမံကိန်းပြီးဆုံးသည်။ ဤအချိန်အတောအတွင်းရလဒ်များနှင့်သက်ဆိုင်သောမေးမြန်းချက်များကိုဖြေရှင်းရန်စီမံကိန်းနောက်ဆက်တွဲ, မေးခွန်းများနှင့်မေးခွန်းများအတွက်စီမံကိန်းများအတွက်စီမံကိန်းများအတွက်စီမံကိန်းများပြုလုပ်ရန်,
●ဘက်စုံမှတ်ချက်များဖြေ - မျိုးရိုးဗီဇကိုခွဲခြားသတ်မှတ်ထားတဲ့ကွဲပြားခြားနားမှုတွေနဲ့ပတ်သက်ပြီးမျိုးရိုးဗီဇကိုဖော်ထုတ်နိုင်ဖို့အတွက်ဒေတာဘေ့စ်မျိုးစုံကိုသုံးတယ်။
ဝန်ဆောင်မှုသတ်မှတ်ချက်များ
| စာကြည့်တိုက် | မဟာဗျူဟာအစီအစဉ် | အကြံပြုဒေတာ output ကို | hi-c signal resolution |
| Hi-C စာကြည့်တိုက် | illumina pe150 | Chromatin Loop: 150x Tad: 50x | Chromatin Loop: 10kb Tad: 40KB |
ဝန်ဆောင်မှုလိုအပ်ချက်များကို
| နမူနာအမျိုးအစား | လိုအပ်သောပမာဏ |
| တိရိစ္ဆာန်တစ်သျှူး | ≥2g |
| အသွေးလုံး | ≥2ml |
| မှိုမှ | ≥1g |
| အပင် - လူငယ်တစ်သျှူး | 1G / Aliquot, 2-4 Aliquotes အကြံပြုခဲ့သည် |
| ယဉ်ကျေးမှုဆဲလ်များ | ≥1x107 |
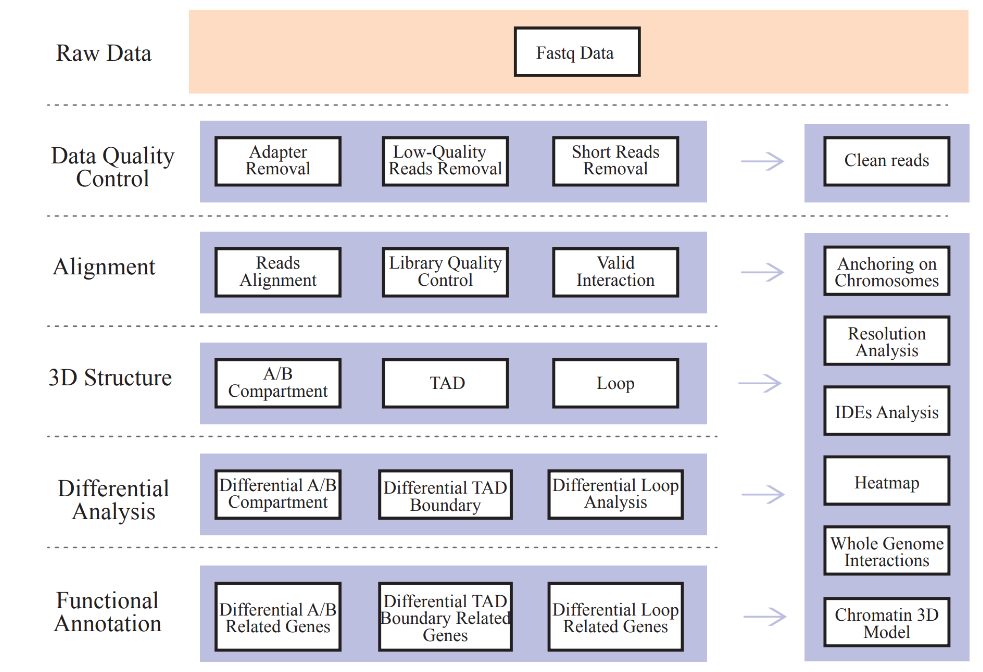
အောက်ပါသုံးသပ်ချက်ပါဝင်သည်:
●ဒေတာ qC ကိုကုန်ကြမ်း,
●မြေပုံနှင့် hi-c စာကြည့်တိုက် QC: ခိုင်လုံသောအပြန်အလှန်ဆက်သွယ်မှုအတွဲများနှင့်အပြန်အလှန်အကျိုးသက်ရောက်မှုများ (IDES),
●မျိုးရိုးဗီဇ - ကျယ်ပြန့်အပြန်အလှန်ဆက်သွယ်မှု - CIS / trans ခွဲခြမ်းစိတ်ဖြာခြင်းနှင့် hi-c interaction မြေပုံ;
● A / B Compartment ဖြန့်ဖြူးမှုကိုခွဲခြမ်းစိတ်ဖြာခြင်း,
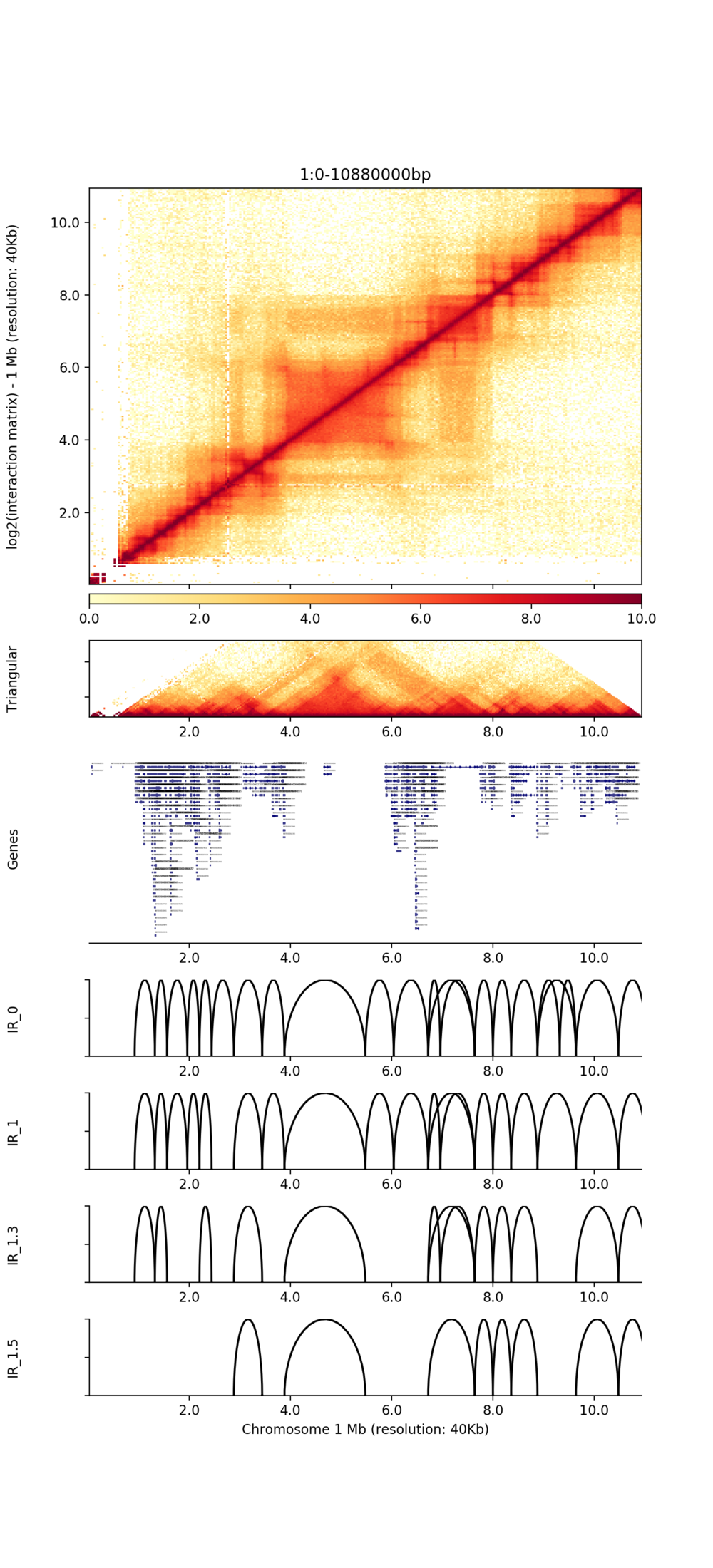
● TADs နှင့် Chromatin ကွင်းကိုခွဲခြားသတ်မှတ်ခြင်း,
● 3D Chromatin ဖွဲ့စည်းပုံနှင့်ဆက်စပ်သောမျိုးရိုးဗီဇများနှင့်သက်ဆိုင်သောမျိုးရိုးဗီဇများအကြား 3D Chromatin ဖွဲ့စည်းပုံတွင်ကွဲပြားခြားနားသောခွဲခြမ်းစိတ်ဖြာခြင်း။
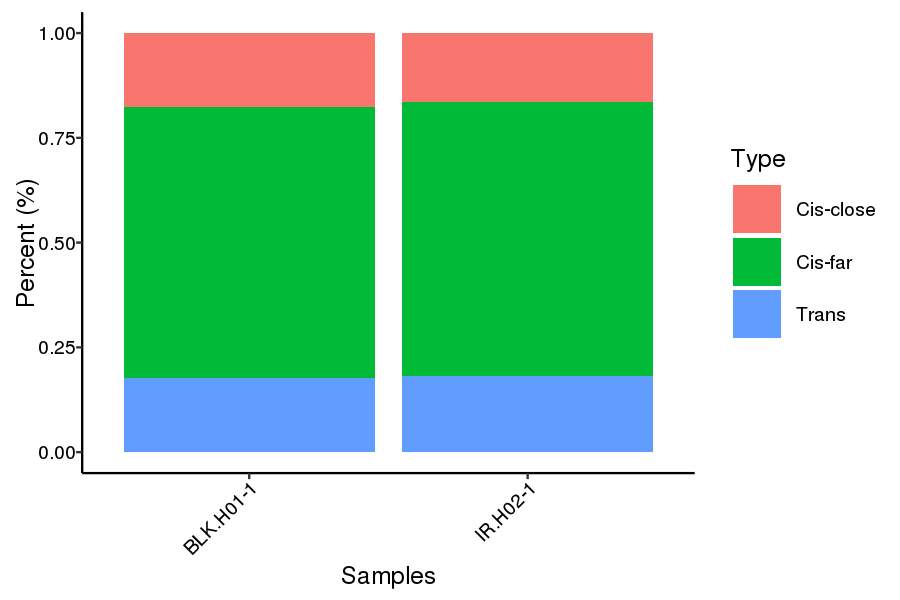
CIS နှင့် trans အချိုးအစားဖြန့်ဖြူး
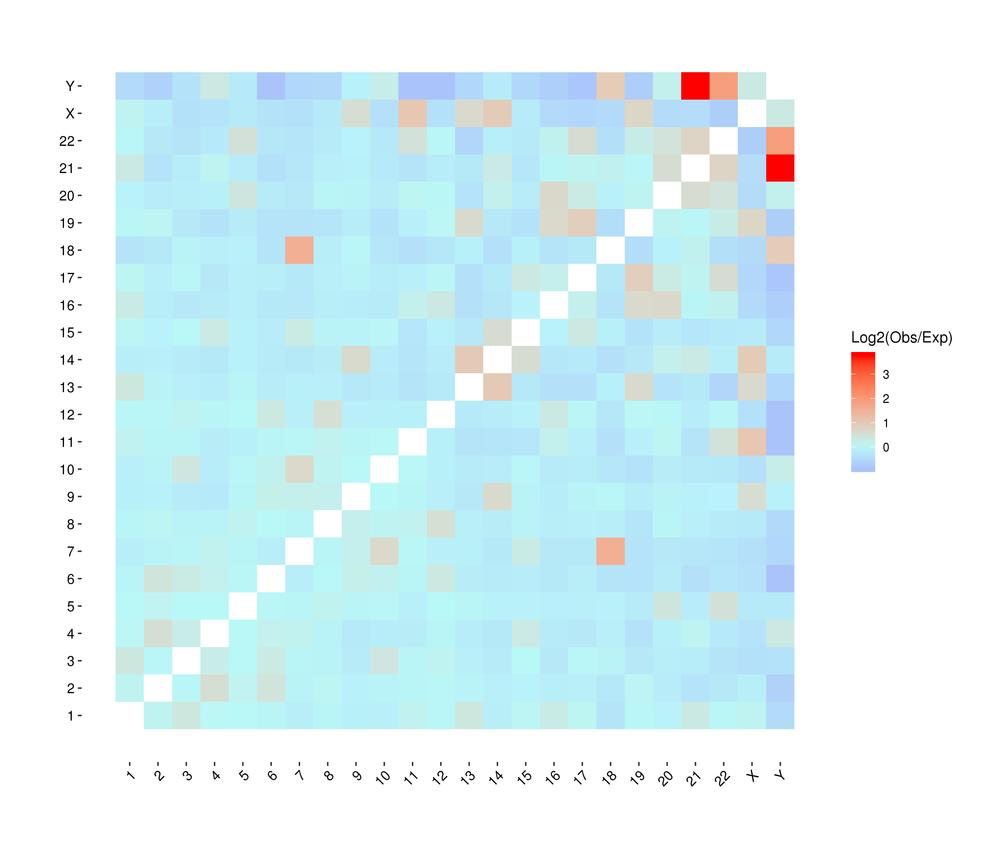
နမူနာအကြားခရိုမိုဆုန်း interactions ၏အပူပေး
A / B အခန်းများ၏မျိုးရိုးဗီဇ - ကျယ်ပြန့်ဖြန့်ဖြူး
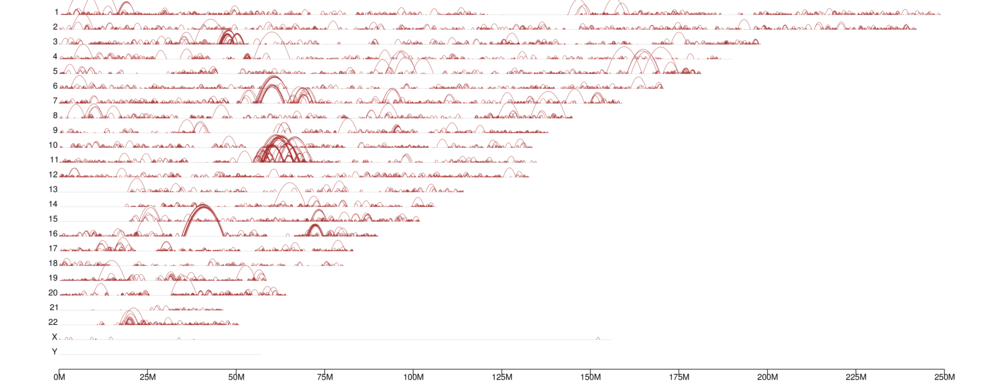
Chromatin ကွင်းဆက်၏မျိုးရိုးဗီဇ - ကျယ်ပြန့်ဖြန့်ဖြူး
TADs ၏ visualization
BMKGENE's Hi-C Sequencing 0 န်ဆောင်မှုများကိုကုသပေးသည့်စာပေများစုဆောင်းခြင်းဖြင့်ကူညီပံ့ပိုးပေးသောသုတေသနတိုးတက်မှုများကိုလေ့လာပါ။
Meng, T. et al ။ (2021) 'နှိုင်းယှဉ်ပေါင်းစပ်ထားသောပေါင်းစုံပေါင်းစုံခွဲခြမ်းစိတ်ဖြာမှုသည် Ca2 ကို Chordyma အတွက် 0 မ်းဖွဲ့ခြင်းအဖြစ်သတ်မှတ်သည်။neuro-oncology, 23 (10), PP ။ 1709-1722 ။ DOI: 10.1093 / neuonc / noab156 ။
Xu, L. et al ။ (2021) '' 32) '3D စနစ်တကျနှင့်မျိုးရိုးဗီဇကိုပြန်လည်ပြုပြင်ခြင်းသည်မိတ်ဆွေများကိုပုဂံ,Acta Pharacutica Sinica B11 (10), PP ။ 3150-3164 ။ Doi: 10.1016 / j.Apsb.apsb.apsb.apsb.2021.03.022 ။