BMKMANU S1000 Spatial စာသားမှတ်တမ်း
BMKMANU S1000 Spatial Transcriptome နည်းပညာပိုင်းဆိုင်ရာ အစီအစဉ်
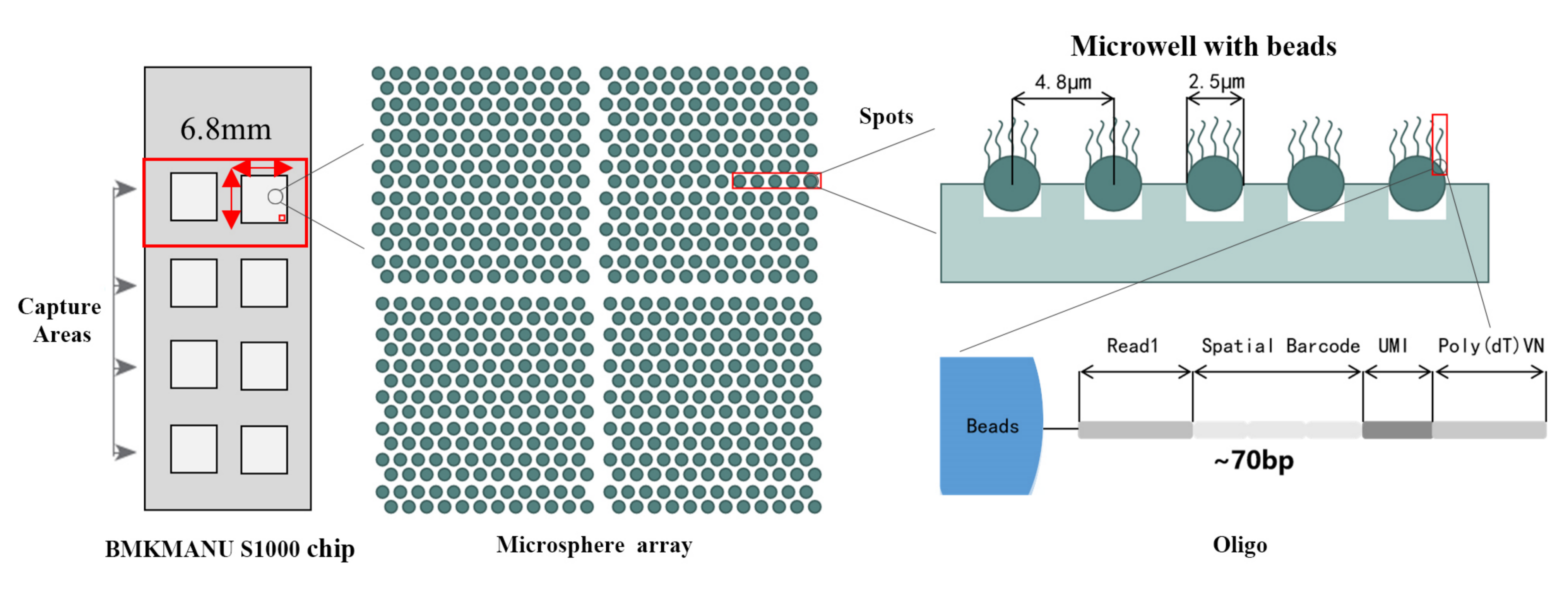
အင်္ဂါရပ်များ
● ကြည်လင်ပြတ်သားမှု- 5 µM
● Spot Diameter: 2.5 µM
● နေရာအရေအတွက်- ခန့်မှန်းခြေ 2 သန်း
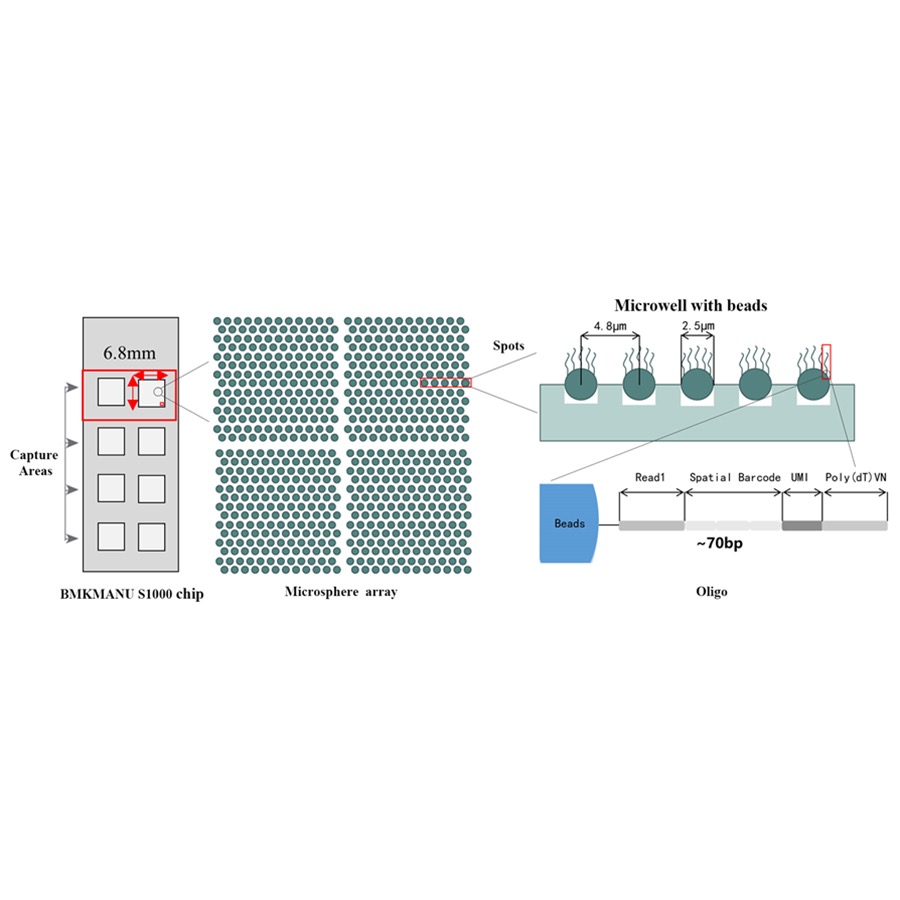
● ဖမ်းယူနိုင်သည့်ဧရိယာ ဖော်မတ် ၃ ခု- 6.8 မီလီမီတာ * 6.8 မီလီမီတာ၊ 11 မီလီမီတာ * 11 မီလီမီတာ သို့မဟုတ် 15 မီလီမီတာ * 20 မီလီမီတာ
● ဘားကုဒ်လုပ်ထားသော ပုတီးစေ့တစ်ခုစီတွင် အပိုင်း 4 ပိုင်းပါဝင်သည့် primers များပါရှိသည်-
mRNA priming နှင့် cDNA ပေါင်းစပ်မှုအတွက် poly(dT) အမြီး
ချဲ့ထွင်မှုဆိုင်ရာ ဘက်လိုက်မှုကို မှန်ကန်စေရန် သီးသန့် မော်လီကျူး အမှတ်အသား (UMI)
Spatial ဘားကုဒ်
တစ်စိတ်တစ်ပိုင်းဖတ်ရန် 1 sequencing primer ၏ စည်းနှောင်မှု အတွဲ
● H&E နှင့် အပိုင်းများ၏ ချောင်းဆိုးခြင်း
● အသုံးပြုရန် အလားအလာဆဲလ်ခွဲခြင်းနည်းပညာ: ဆဲလ်တစ်ခုစီ၏ နယ်နိမိတ်များကို ဆုံးဖြတ်ရန်နှင့် ဆဲလ်တစ်ခုစီသို့ ဗီဇဖော်ပြမှုကို မှန်ကန်စွာသတ်မှတ်ရန် H&E စွန်းထင်းမှု၊ ချောင်းဆိုးခြင်းနှင့် RNA စီစီခြင်းတို့ကို ပေါင်းစပ်ခြင်း။
BMKMANU S1000 ၏ အားသာချက်များ
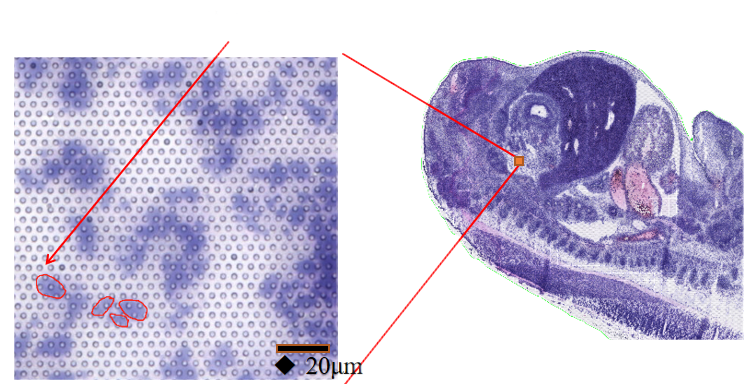
●Sub-cellular Resolution- ဖမ်းယူရရှိသည့်နေရာတစ်ခုစီတွင် အချင်း 2.5 µm နှင့် အချင်း 2.5 µm ရှိသော spatial barcoded Spots 2 သန်းကျော်ပါဝင်ပြီး အစက်အပြောက်စင်တာများကြား 5 µm အကွာအဝေးရှိကာ၊ ဆဲလ်ခွဲခွဲခွဲခြမ်းစိတ်ဖြာမှု (5 µm) ဖြင့် spatial transcriptome ခွဲခြမ်းစိတ်ဖြာမှုကို ပံ့ပိုးပေးပါသည်။
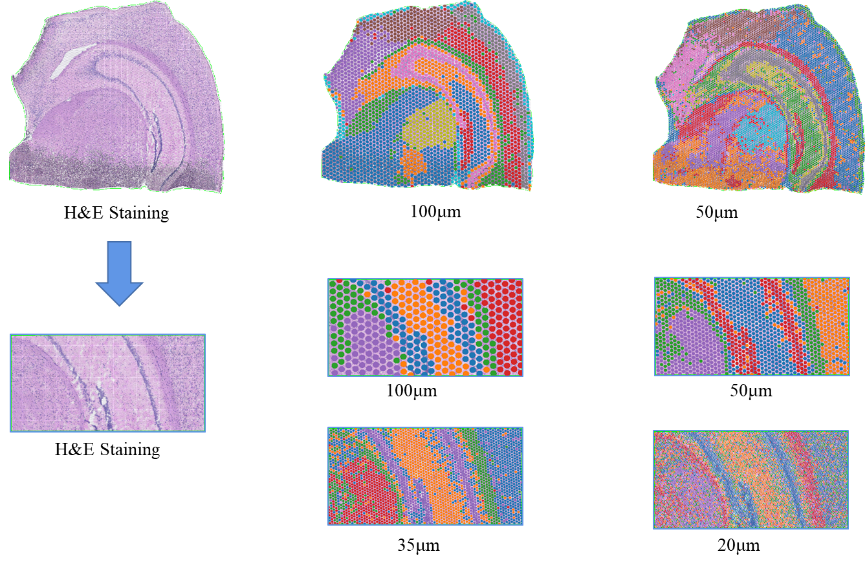
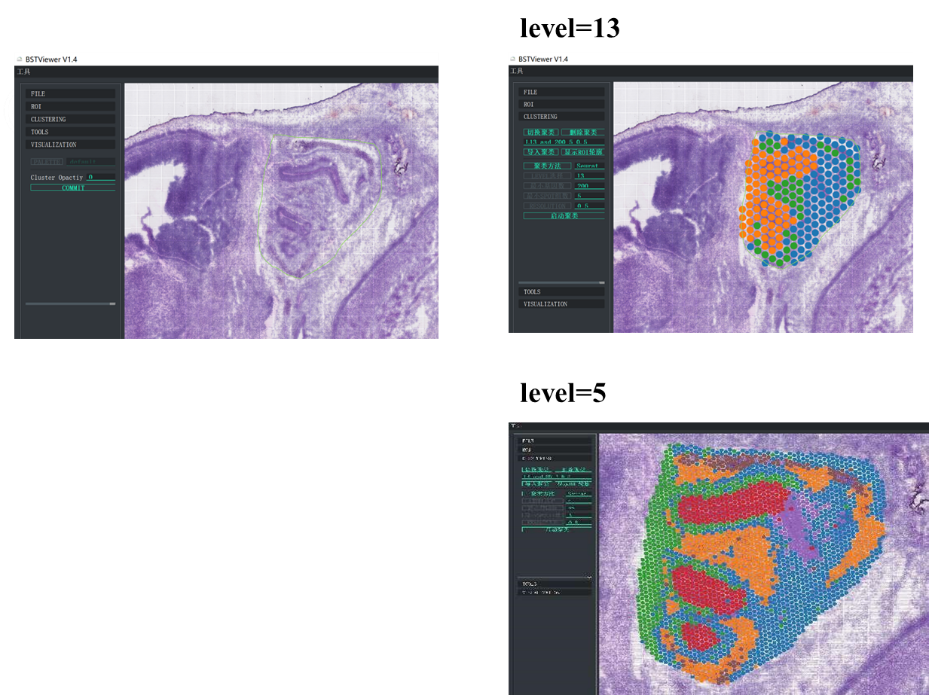
●Multi-level Resolution ပိုင်းခြားစိတ်ဖြာချက်-ကွဲပြားသောတစ်ရှူးအင်္ဂါရပ်များကို အကောင်းဆုံးဖြေရှင်းနိုင်စေရန် 100 μm မှ 5 μm အထိ လိုက်လျောညီထွေရှိသော အဆင့်ပေါင်းများစွာ ခွဲခြမ်းစိတ်ဖြာမှု။
● “ဆလိုက်သုံးခုတွင်” ဆဲလ်အပိုင်းပိုင်းနည်းပညာကို အသုံးပြုရန် ဖြစ်နိုင်ခြေ-ဆလိုက်တစ်ခုတည်းတွင် မီးချောင်းဆိုးခြင်း၊ H&E စွန်းထင်းခြင်းနှင့် RNA စီခြင်းတို့ကို ပေါင်းစပ်ထားသော ကျွန်ုပ်တို့၏ "သုံး-တစ်ပုံ" ခွဲခြမ်းစိတ်ဖြာမှုဆိုင်ရာ အယ်လဂိုရီသမ်သည် နောက်ဆက်တွဲ ဆဲလ်အခြေခံ စာသားပြောင်းခြင်းအတွက် ဆဲလ်နယ်နိမိတ်များကို ဖော်ထုတ်နိုင်စေပါသည်။
●Multiple Sequencing Platforms နှင့် လိုက်ဖက်သည်။NGS နှင့် long-read sequencing နှစ်မျိုးလုံး ရနိုင်ပါသည်။
●1-8 Active Capture Area ၏ ပြောင်းလွယ်ပြင်လွယ် ဒီဇိုင်း: ဖမ်းယူဧရိယာ၏အရွယ်အစားသည် ပြောင်းလွယ်ပြင်လွယ်ဖြစ်ပြီး ဖော်မတ် 3 ခု (6.8 မီလီမီတာ * 6.8 မီလီမီတာ၊ 11 မီလီမီတာ * 11 မီလီမီတာ နှင့် 15 မီလီမီတာ * 20 မီလီမီတာ) ကို အသုံးပြုနိုင်သည်။
●တစ်ခုတည်းသော ဝန်ဆောင်မှု: ၎င်းသည် cryo ခွဲမွေးခြင်း၊ စွန်းထင်းခြင်း၊ တစ်ရှူးများကို ပိုမိုကောင်းမွန်အောင်ပြုလုပ်ခြင်း၊ spatial barcoding၊ စာကြည့်တိုက်ပြင်ဆင်ခြင်း၊ စီစစ်ခြင်းနှင့် ဇီဝနည်းပညာဆိုင်ရာများ အပါအဝင် အတွေ့အကြုံနှင့် ကျွမ်းကျင်မှုအခြေခံအဆင့်များအားလုံးကို ပေါင်းစပ်ထားသည်။
●ပြီးပြည့်စုံသော Bioinformatics နှင့် User-friendly ရလဒ်များကို မြင်ယောင်ခြင်း-ပက်ကေ့ဂျ်တွင် ခွဲခြမ်းစိတ်ဖြာမှု ၂၉ ခုနှင့် အရည်အသွေးမြင့် ကိန်းဂဏန်း 100+ ပါ၀င်ပြီး ဆဲလ်ခွဲထွက်ခြင်းနှင့် အစက်အပြောက်အစုအဝေးများကို မြင်သာစေရန်နှင့် စိတ်ကြိုက်ပြုလုပ်ရန် အိမ်တွင်းတီထွင်ထားသောဆော့ဖ်ဝဲလ်ကို အသုံးပြုခြင်းဖြင့် ပေါင်းစပ်ထားသည်။
●စိတ်ကြိုက်ဒေတာခွဲခြမ်းစိတ်ဖြာခြင်းနှင့် စိတ်ကူးပုံဖော်ခြင်း: မတူညီသော သုတေသနတောင်းဆိုမှုများအတွက် ရနိုင်ပါသည်။
●ကျွမ်းကျင်သော နည်းပညာအဖွဲ့လူသား၊ ကြွက်၊ နို့တိုက်သတ္တဝါ၊ ငါးနှင့် အပင်များအပါအဝင် မျိုးစိတ်ပေါင်း 250 ကျော်နှင့် မျိုးစိတ်ပေါင်း 100+ တွင် အတွေ့အကြုံဖြင့်။
●ပရောဂျက်တစ်ခုလုံးရှိ အချိန်နှင့်တပြေးညီ အပ်ဒိတ်များ: စမ်းသပ်မှုတိုးတက်မှုကို အပြည့်အဝထိန်းချုပ်ထားသည်။
●Single-cell mRNA Sequencing ဖြင့် ရွေးချယ်နိုင်သော ပူးတွဲခွဲခြမ်းစိတ်ဖြာခြင်း။
ဝန်ဆောင်မှုသတ်မှတ်ချက်များ
|
နမူနာ လိုအပ်ချက်များ
| စာကြည့်တိုက် |
Sequence ဗျူဟာ
| ဒေတာအကြံပြုထားသည်။ | အရည်အသွေးထိန်းချုပ်ရေး |
| OCT-embedded cryo နမူနာများ၊ နမူနာတစ်ခုလျှင် 3 တုံး | S1000 cDNA စာကြည့်တိုက် | Illumina PE150 (အခြားပလပ်ဖောင်းများ ရနိုင်သည်) | 100 uM တွင် 100K PE ဖတ်သည်။ (60-150 Gb) | RIN>၇ |
နမူနာပြင်ဆင်မှုလမ်းညွှန်နှင့် ဝန်ဆောင်မှုလုပ်ငန်းအသွားအလာဆိုင်ရာ အသေးစိတ်အချက်အလက်များအတွက်၊ ကျေးဇူးပြု၍ တစ်ဦးနှင့်စကားပြောပါ။BMKGENE ကျွမ်းကျင်သူ
Service Work Flow
နမူနာပြင်ဆင်မှုအဆင့်တွင်၊ အရည်အသွေးမြင့် RNA ကိုရရှိနိုင်ကြောင်းသေချာစေရန် ကနဦးအမြောက်အများ RNA ထုတ်ယူစမ်းသပ်မှုကို လုပ်ဆောင်သည်။ တစ်ရှူးများကို ပိုမိုကောင်းမွန်အောင်ပြုလုပ်ခြင်းအဆင့်တွင် အပိုင်းများကို စွန်းထင်းပြီး မြင်သာထင်သာမြင်သာအောင်ပြုလုပ်ပြီး တစ်ရှူးမှ mRNA ထုတ်လွှတ်မှုအတွက် permeabilization အခြေအနေများကို အကောင်းဆုံးဖြစ်အောင်ပြုလုပ်ထားသည်။ ထို့နောက် စာကြည့်တိုက်တည်ဆောက်မှုအတွင်း ပိုမိုကောင်းမွန်အောင်ပြုလုပ်ထားသော ပရိုတိုကောကို ပေါင်းစပ်ခြင်းနှင့် ဒေတာခွဲခြမ်းစိတ်ဖြာခြင်းတို့ဖြင့် လုပ်ဆောင်သည်။
ပြီးပြည့်စုံသော ဝန်ဆောင်မှုလုပ်ငန်းအသွားအလာတွင် ပရောဂျက်ကို ချောမွေ့စွာလုပ်ဆောင်ခြင်းအား သေချာစေရန် တုံ့ပြန်မှုတုံ့ပြန်ချက်ကွင်းဆက်ကို ထိန်းသိမ်းထားရန် အချိန်နှင့်တပြေးညီ အပ်ဒိတ်များနှင့် ဖောက်သည်အတည်ပြုချက်များ ပါဝင်ပါသည်။
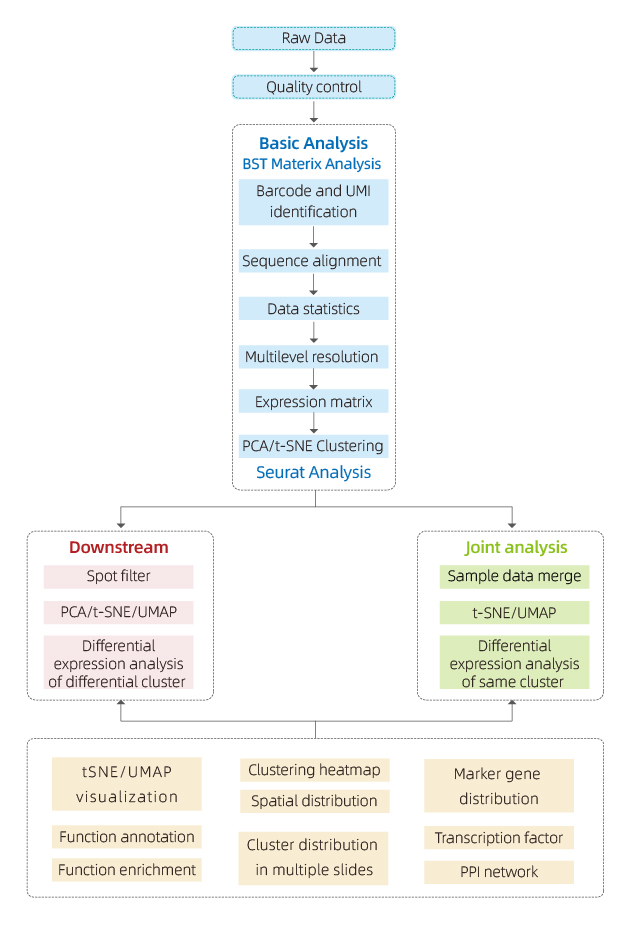
BMKMANU S1000 မှထုတ်ပေးသောဒေတာကို BMKGENE မှ သီးခြားဒီဇိုင်းထုတ်ထားသည့် ဆော့ဖ်ဝဲ “BSTMatrix” ကို အသုံးပြု၍ ခွဲခြမ်းစိပ်ဖြာကာ Gene Expression Matrix ကို ဖန်တီးပေးပါသည်။ ထိုမှနေ၍ ဒေတာအရည်အသွေးထိန်းချုပ်မှု၊ အတွင်းနမူနာခွဲခြမ်းစိတ်ဖြာမှုနှင့် အုပ်စုအချင်းချင်းခွဲခြမ်းစိတ်ဖြာမှုတို့ပါ၀င်သည့် စံအစီရင်ခံစာကို ထုတ်ပေးပါသည်။
● ဒေတာအရည်အသွေး ထိန်းချုပ်မှု-
ဒေတာထုတ်ပေးမှုနှင့် အရည်အသွေးရမှတ်များ ဖြန့်ဖြူးခြင်း။
နေရာအလိုက် မျိုးဗီဇ ထောက်လှမ်းခြင်း။
တစ်ရှူးများ လွှမ်းခြုံထားသည်။
● အတွင်း-နမူနာ ခွဲခြမ်းစိတ်ဖြာမှု-
မျိုးဗီဇကြွယ်ဝမှု
လျှော့ချထားသော အတိုင်းအတာ ခွဲခြမ်းစိတ်ဖြာမှု အပါအဝင် အစက်အပြောက် အစုလိုက် ပြုလုပ်ခြင်း။
အစုအဝေးများအကြား ကွဲပြားသောဖော်ပြချက်ခွဲခြမ်းစိတ်ဖြာမှု- အမှတ်အသားမျိုးဗီဇများကို ဖော်ထုတ်ခြင်း။
လုပ်ဆောင်နိုင်သော မှတ်ချက်များနှင့် အမှတ်အသားမျိုးဗီဇများ ကြွယ်ဝစေသည်။
● အုပ်စုအချင်းချင်း ခွဲခြမ်းစိတ်ဖြာခြင်း-
နမူနာနှစ်ခုလုံးမှ အစက်အပြောက်များကို ပြန်လည်ပေါင်းစပ်ခြင်း (ဥပမာ။ ရောဂါရှိသော၊ ထိန်းချုပ်မှု) နှင့် ပြန်လည်စုဖွဲ့ခြင်း။
အစုအဖွဲ့တစ်ခုစီအတွက် အမှတ်အသားမျိုးဗီဇများကို ဖော်ထုတ်ခြင်း။
လုပ်ဆောင်နိုင်သော မှတ်ချက်များနှင့် အမှတ်အသားမျိုးဗီဇများ ကြွယ်ဝစေသည်။
အုပ်စုများကြားတွင် တူညီသောအစုအဝေး၏ ကွဲပြားသောဖော်ပြချက်
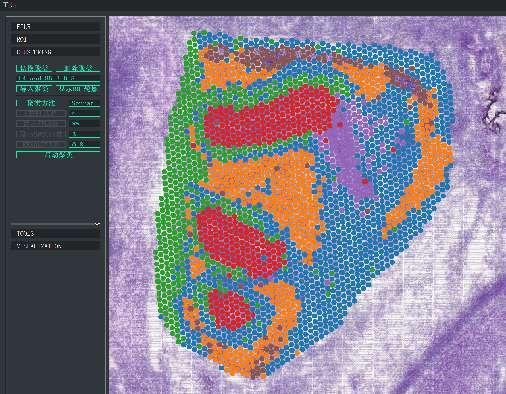
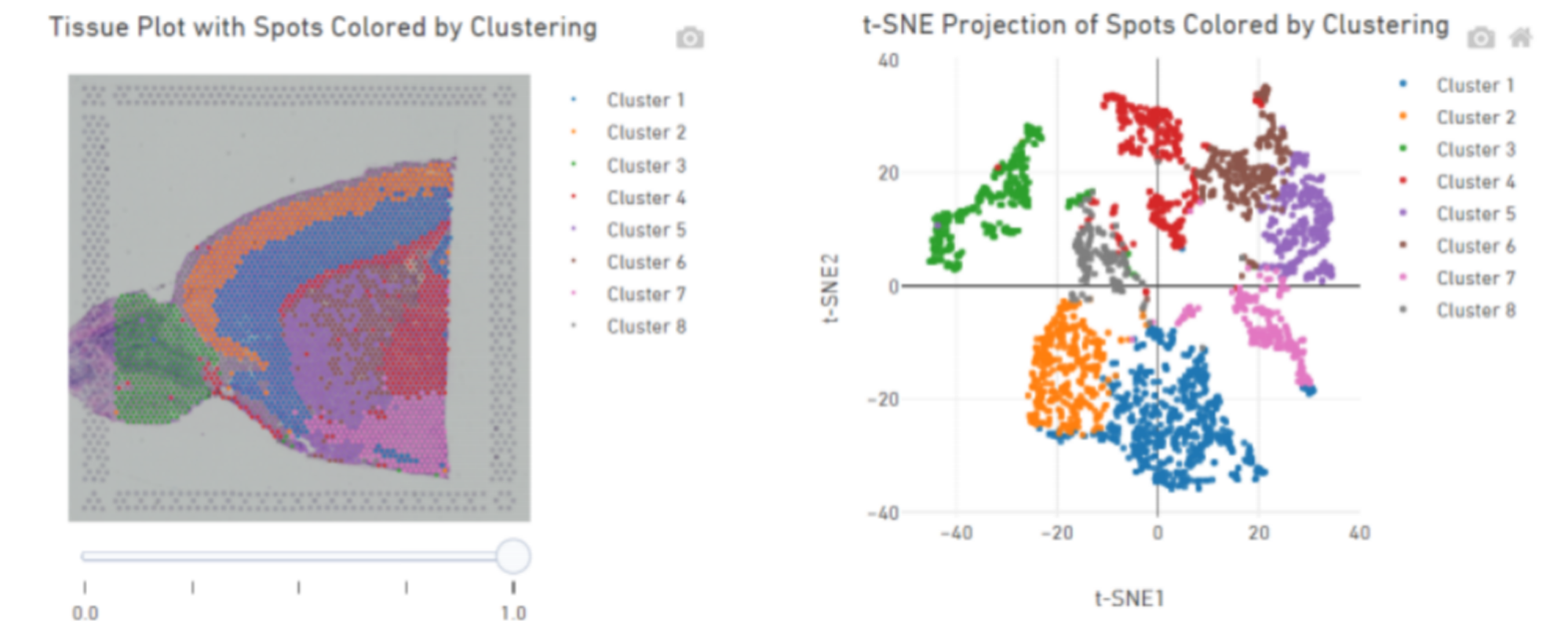
ထို့အပြင် BMKGENE မှတီထွင်ထားသည့် “BSTViewer” သည် အသုံးပြုသူအတွက် အဆင်ပြေစေမည့် ကိရိယာတစ်ခုဖြစ်ပြီး အသုံးပြုသူသည် မျိုးရိုးဗီဇဖော်ပြမှုကို မြင်သာစေရန်နှင့် မတူညီသော resolution များတွင် အစုလိုက်အပုံလိုက်ပြုလုပ်နိုင်စေပါသည်။
BMKGene သည် အသုံးပြုသူဖော်ရွေသော အမြင်အာရုံအတွက် ဆော့ဖ်ဝဲလ်ကို တီထွင်ခဲ့သည်။
BSTViewer သည် အဆင့်များစွာသော ကြည်လင်ပြတ်သားမှုတွင် အစုလိုက်အပြုံလိုက် အစက်အပြောက်လုပ်ခြင်း။
BSTcellViewer- အလိုအလျောက်နှင့် လူကိုယ်တိုင် ဆဲလ်ခွဲခြမ်းခြင်း။
အတွင်း-နမူနာ ခွဲခြမ်းစိတ်ဖြာခြင်း။
အစက်အပြောက်အစုအဝေး-
အမှတ်အသား မျိုးဗီဇ ခွဲခြားသတ်မှတ်ခြင်းနှင့် နေရာအနှံ့ ဖြန့်ဖြူးခြင်း-
အုပ်စုအချင်းချင်း ခွဲခြမ်းစိတ်ဖြာခြင်း။
အုပ်စုနှစ်ခုလုံးမှ ဒေတာပေါင်းစပ်ပြီး ပြန်လည်အစုအဝေး-
အစုအဝေးအသစ်များ၏ အမှတ်အသား ဗီဇ-
ဤအသားပေးထုတ်ဝေမှုတွင် BMKGene ၏ spatial transcriptomics ဝန်ဆောင်မှုများက BMKManu S1000 နည်းပညာဖြင့် ပံ့ပိုးပေးထားသည့် တိုးတက်မှုများကို စူးစမ်းလေ့လာပါ-
သီချင်း၊ X. et al. (2023) 'Spatial transcriptomics သည် ခရမ်းချဉ်သီးတွင် အညွန့်များ ပေါက်ထွက်ခြင်းကို မြှင့်တင်ရာတွင် ပါဝင်သည့် အလင်း-သွေးဆောင် ကလိုရန်ခမာဆဲလ်များကို ဖော်ထုတ်ပြသသည်'၊အမေရိကန်ပြည်ထောင်စု၏ အမျိုးသားသိပ္ပံအကယ်ဒမီ၏ လုပ်ငန်းစဉ်များ, 120(38), p. e2310163120။ doi: 10.1073/pnas.2310163120
မင်းက Y. et al. (2023) ' sequencing-based spatial transcriptomic methods ၏စနစ်တကျ နှိုင်းယှဉ်ခြင်း '၊bioRxiv, p ။ 2023.12.03.569744။ doi: 10.1101/2023.12.03.569744။