10x Genomics Visium Spatial စာသားမှတ်တမ်း
နည်းပညာပိုင်းဆိုင်ရာအစီအစဉ်

အင်္ဂါရပ်များ
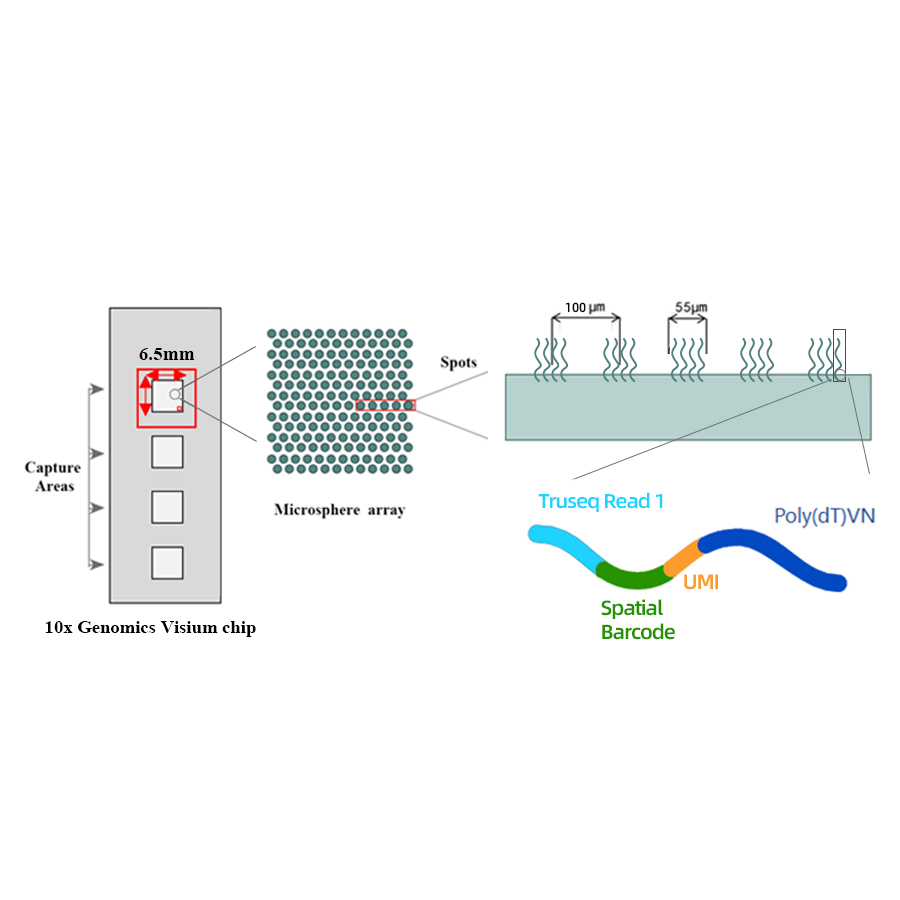
● ကြည်လင်ပြတ်သားမှု- 100 µM
● Spot Diameter: 55 µM
● အစက်အပြောက်အရေအတွက်- 4992
● ဖမ်းယူဧရိယာ- 6.5 x 6.5 မီလီမီတာ
● ဘားကုဒ်လုပ်ထားသော နေရာတစ်ခုစီတွင် အပိုင်း 4 ပိုင်းပါဝင်သည့် primers များပါရှိသည်-
- mRNA priming နှင့် cDNA ပေါင်းစပ်မှုအတွက် poly(dT) အမြီး
- ချဲ့ထွင်မှုဆိုင်ရာ ဘက်လိုက်မှုကို ပြုပြင်ရန် သီးသန့် Molecular Identifier (UMI)
- Spatial ဘားကုဒ်
- တစ်စိတ်တစ်ပိုင်းဖတ်ရန် 1 sequencing primer ၏ စည်းနှောင်မှု အစီအစဥ်
● H&E အပိုင်းများကို စွန်းထင်းခြင်း။
အားသာချက်များ
●တစ်ခုတည်းသော ဝန်ဆောင်မှု: cryo ခွဲမွေးခြင်း၊ စွန်းထင်းခြင်း၊ တစ်ရှူးများ ပိုမိုကောင်းမွန်အောင်ပြုလုပ်ခြင်း၊ spatial barcoding၊ စာကြည့်တိုက်ပြင်ဆင်မှု၊ စီစစ်ခြင်းနှင့် ဇီဝနည်းပညာဆိုင်ရာများ အပါအဝင် အတွေ့အကြုံနှင့် ကျွမ်းကျင်မှုအခြေခံအဆင့်များအားလုံးကို ပေါင်းစပ်ထားသည်။
● ကျွမ်းကျင်သော နည်းပညာအဖွဲ့လူသား၊ ကြွက်၊ နို့တိုက်သတ္တဝါ၊ ငါးနှင့် အပင်များအပါအဝင် မျိုးစိတ်ပေါင်း 250 ကျော်နှင့် မျိုးစိတ်ပေါင်း 100+ တွင် အတွေ့အကြုံဖြင့်။
●ပရောဂျက်တစ်ခုလုံးတွင် အချိန်နှင့်တပြေးညီ အပ်ဒိတ်: စမ်းသပ်မှုတိုးတက်မှုကို အပြည့်အဝထိန်းချုပ်ထားသည်။
●ပြီးပြည့်စုံသော စံဇီဝသတင်းအချက်အလက်များ-အထုပ်တွင် ခွဲခြမ်းစိတ်ဖြာမှု ၂၉ ခုနှင့် အရည်အသွေးမြင့် ကိန်းဂဏန်း 100+ ပါဝင်သည်။
●စိတ်ကြိုက်ဒေတာခွဲခြမ်းစိတ်ဖြာခြင်းနှင့် စိတ်ကူးပုံဖော်ခြင်း: မတူညီသော သုတေသနတောင်းဆိုမှုများအတွက် ရနိုင်ပါသည်။
●Single-cell mRNA Sequencing ဖြင့် ရွေးချယ်နိုင်သော ပူးတွဲခွဲခြမ်းစိတ်ဖြာခြင်း။
သတ်မှတ်ချက်များ
| နမူနာလိုအပ်ချက်များ | စာကြည့်တိုက် | Sequence ဗျူဟာ | ဒေတာအကြံပြုထားသည်။ | အရည်အသွေးထိန်းချုပ်မှု |
| OCT-embedded cryo နမူနာများ (အကောင်းဆုံး အချင်း- 6x6x6 mm³ ခန့်) နမူနာတစ်ခုလျှင် 2 တုံး | 10X Visium cDNA စာကြည့်တိုက် | Illumina PE150 | တစ်ကွက်လျှင် 50K PE ဖတ်သည်။ (60Gb) | RIN > ၇ |
နမူနာပြင်ဆင်မှုလမ်းညွှန်နှင့် ဝန်ဆောင်မှုလုပ်ငန်းအသွားအလာဆိုင်ရာ အသေးစိတ်အချက်အလက်များအတွက် ကျေးဇူးပြု၍ တစ်ဦးနှင့်စကားပြောပါ။BMKGENE ကျွမ်းကျင်သူ
ဝန်ဆောင်မှုလုပ်ငန်းအသွားအလာ
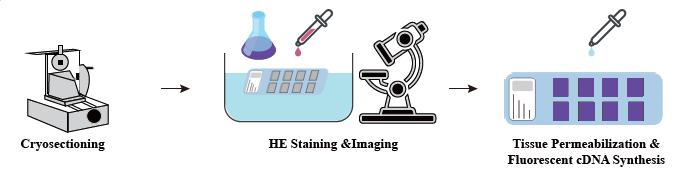
နမူနာပြင်ဆင်မှုအဆင့်တွင်၊ အရည်အသွေးမြင့် RNA ကိုရရှိနိုင်ကြောင်းသေချာစေရန် ကနဦးအမြောက်အများ RNA ထုတ်ယူစမ်းသပ်မှုကို လုပ်ဆောင်သည်။ တစ်ရှူးများကို ပိုမိုကောင်းမွန်အောင်ပြုလုပ်ခြင်းအဆင့်တွင် အပိုင်းများကို စွန်းထင်းပြီး မြင်သာထင်သာမြင်သာအောင်ပြုလုပ်ပြီး တစ်ရှူးမှ mRNA ထုတ်လွှတ်မှုအတွက် permeabilization အခြေအနေများကို အကောင်းဆုံးဖြစ်အောင်ပြုလုပ်ထားသည်။ ထို့နောက် စာကြည့်တိုက်တည်ဆောက်မှုအတွင်း ပိုမိုကောင်းမွန်အောင်ပြုလုပ်ထားသော ပရိုတိုကောကို ပေါင်းစပ်ခြင်းနှင့် ဒေတာခွဲခြမ်းစိတ်ဖြာခြင်းတို့ဖြင့် လုပ်ဆောင်သည်။
ပြီးပြည့်စုံသော ဝန်ဆောင်မှုလုပ်ငန်းအသွားအလာတွင် ပရောဂျက်ကို ချောမွေ့စွာလုပ်ဆောင်ခြင်းအား သေချာစေရန် တုံ့ပြန်မှုတုံ့ပြန်ချက်ကွင်းဆက်ကို ထိန်းသိမ်းထားရန် အချိန်နှင့်တပြေးညီ အပ်ဒိတ်များနှင့် ဖောက်သည်အတည်ပြုချက်များ ပါဝင်ပါသည်။

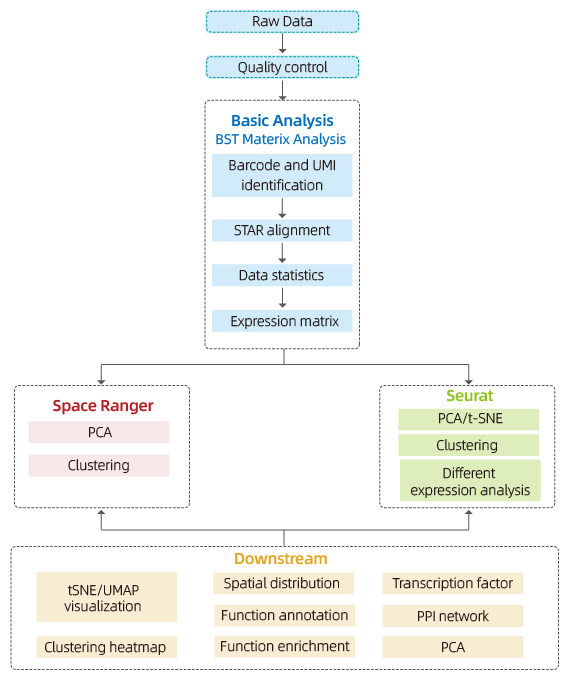
အောက်ပါ ခွဲခြမ်းစိတ်ဖြာမှု ပါဝင်သည်-
ဒေတာအရည်အသွေး ထိန်းချုပ်ရေး-
o ဒေတာထုတ်ပေးမှုနှင့် အရည်အသွေးရမှတ်များ ဖြန့်ဖြူးခြင်း။
o အစက်အပြောက်အလိုက် မျိုးဗီဇ ထောက်လှမ်းခြင်း။
o တစ်ရှူးကို ဖုံးအုပ်ထားပါ။
အတွင်း-နမူနာ ခွဲခြမ်းစိတ်ဖြာခြင်း-
o မျိုးဗီဇကြွယ်ဝမှု
o အစက်အပြောက် အစုလိုက် ခွဲခြမ်းစိတ်ဖြာခြင်း အပါအဝင်၊
o အစုအဝေးများအကြား ကွဲပြားသောဖော်ပြချက်ခွဲခြမ်းစိတ်ဖြာမှု- အမှတ်အသားမျိုးဗီဇများကို ဖော်ထုတ်ခြင်း။
o လုပ်ဆောင်ချက်ဆိုင်ရာ မှတ်ချက်များနှင့် အမှတ်အသားမျိုးဗီဇများ ကြွယ်ဝစေခြင်း။
အုပ်စုအချင်းချင်း ခွဲခြမ်းစိတ်ဖြာခြင်း။
o နမူနာနှစ်ခုလုံးမှ အစက်အပြောက်များကို ပြန်လည်ပေါင်းစပ်ခြင်း (ဥပမာ- ရောဂါဖြစ်ပွားပြီး ထိန်းချုပ်ခြင်း) နှင့် ပြန်လည်စုဖွဲ့ခြင်း
o အစုအဝေးတစ်ခုစီအတွက် အမှတ်အသားမျိုးဗီဇများကို ဖော်ထုတ်ခြင်း။
o လုပ်ဆောင်ချက်ဆိုင်ရာ မှတ်ချက်များနှင့် အမှတ်အသားမျိုးဗီဇများ ကြွယ်ဝစေခြင်း။
o အုပ်စုများကြားတွင် တူညီသောအစုအဝေး၏ ကွဲပြားသောဖော်ပြချက်
အတွင်း-နမူနာ ခွဲခြမ်းစိတ်ဖြာခြင်း။
အစက်အပြောက်အစုအဝေး

အမှတ်အသားမျိုးဗီဇများ ခွဲခြားသတ်မှတ်ခြင်းနှင့် နေရာအနှံ့ ဖြန့်ဖြူးခြင်း။


အုပ်စုအချင်းချင်း ခွဲခြမ်းစိတ်ဖြာခြင်း။
အုပ်စုနှစ်ခုလုံးမှ ဒေတာပေါင်းစပ်ပြီး ပြန်လည်အစုအဝေး
အစုအဖွဲ့အသစ်များ၏ အမှတ်အသား ဗီဇများ
10X Visium မှ BMKGene ၏ spatial transcriptomics ဝန်ဆောင်မှုမှ ပံ့ပိုးပေးထားသည့် တိုးတက်မှုများကို စူးစမ်းလေ့လာပါ-
Chen, D. et al. (2023) 'mthl1၊ နို့တိုက်သတ္တဝါများ၏ ကပ်တွယ်မှု GPCRs များ၏ အလားအလာရှိသော Drosophila တူညီသောတူတူတူဖြစ်သော 'mthl1၊ သည် ယင်ကောင်များတွင် ထိုးသွင်းထားသော oncogenic ဆဲလ်များကို ကင်ဆာဖြစ်စေသော တုံ့ပြန်မှုများတွင် ပါဝင်သည်'၊အမေရိကန်ပြည်ထောင်စု၏ အမျိုးသားသိပ္ပံအကယ်ဒမီ၏ လုပ်ငန်းစဉ်များ, 120(30), p. e2303462120။ doi: /10.1073/pnas.2303462120
Chen, Y. et al. (2023) 'STEEL သည် high-resolution delineation of spatiotemporal transscriptomic data'၊Bioinformatics တွင် ရှင်းလင်းချက်များ၊ ၂၄(၂)၊စ၊ ၁–၁၀။ doi- 10.1093/BIB/BBAD068။
Liu, C. et al. (2022) 'သစ်ခွပန်းများ ဖွံ့ဖြိုးတိုးတက်မှုအတွက် spatiotemporal atlas of organogenesis'၊Nucleic Acids သုတေသန, 50(17), စစ. 9724–9737။ doi- 10.1093/NAR/GKAC773။
Wang, J. et al. (2023) 'Spatial Transcriptomics နှင့် Single-nucleus RNA Sequencing ပေါင်းစပ်ခြင်းသည် သားအိမ် Leiomyoma အတွက် ဖြစ်နိုင်ချေရှိသော ကုထုံးနည်းဗျူဟာများကို ဖော်ပြသည်'၊နိုင်ငံတကာ ဇီဝသိပ္ပံဂျာနယ်၊ ၁၉(၈)၊စ။ ၂၅၁၅–၂၅၃၀။ doi: 10.7150/IJBS.83510။