മുഴുനീള mRNA സീക്വൻസിങ്-നാനോപോർ
ഫീച്ചറുകൾ
● സിഡിഎൻഎ സിന്തസിസ്, ലൈബ്രറി തയ്യാറാക്കൽ എന്നിവയ്ക്ക് ശേഷം പോളി-എ എംആർഎൻഎയുടെ ക്യാപ്ചർ
● പൂർണ്ണ ദൈർഘ്യമുള്ള ട്രാൻസ്ക്രിപ്റ്റുകളുടെ ക്രമം
● ഒരു റഫറൻസ് ജീനോമിലേക്കുള്ള വിന്യാസത്തെ അടിസ്ഥാനമാക്കിയുള്ള ബയോ ഇൻഫോർമാറ്റിക് വിശകലനം
● ബയോഇൻഫോർമാറ്റിക് വിശകലനത്തിൽ ജീൻ, ഐസോഫോം തലത്തിലുള്ള പ്രകടനങ്ങൾ മാത്രമല്ല, lncRNA, ജീൻ ഫ്യൂഷനുകൾ, പോളി-അഡെനൈലേഷൻ, ജീൻ ഘടന എന്നിവയുടെ വിശകലനവും ഉൾപ്പെടുന്നു.
സേവന നേട്ടങ്ങൾ
●ഐസോഫോം തലത്തിൽ ആവിഷ്കാരത്തിൻ്റെ അളവ്: വിശദവും കൃത്യവുമായ എക്സ്പ്രഷൻ വിശകലനം പ്രാപ്തമാക്കുന്നു, മുഴുവൻ ജീൻ എക്സ്പ്രഷനും വിശകലനം ചെയ്യുമ്പോൾ മറഞ്ഞിരിക്കാവുന്ന മാറ്റം അനാവരണം ചെയ്യുന്നു
●കുറഞ്ഞ ഡാറ്റ ആവശ്യകതകൾ:നെക്സ്റ്റ്-ജനറേഷൻ സീക്വൻസിംഗുമായി (NGS) താരതമ്യപ്പെടുത്തുമ്പോൾ, നാനോപോർ സീക്വൻസിംഗ് കുറഞ്ഞ ഡാറ്റ ആവശ്യകതകൾ പ്രകടിപ്പിക്കുന്നു, ഇത് ചെറിയ ഡാറ്റയ്ക്കൊപ്പം തുല്യമായ ജീൻ എക്സ്പ്രഷൻ ക്വാണ്ടിഫിക്കേഷൻ സാച്ചുറേഷൻ അനുവദിക്കുന്നു.
●എക്സ്പ്രഷൻ ക്വാണ്ടിഫിക്കേഷൻ്റെ ഉയർന്ന കൃത്യത: ജീൻ തലത്തിലും ഐസോഫോം തലത്തിലും
●അധിക ട്രാൻസ്ക്രിപ്റ്റോമിക് വിവരങ്ങളുടെ തിരിച്ചറിയൽ: ഇതര പോളിഡെനൈലേഷൻ, ഫ്യൂഷൻ ജീനുകളും എൽസിഎൻആർഎൻഎയും അവയുടെ ലക്ഷ്യ ജീനുകളും
●വിപുലമായ വൈദഗ്ധ്യം: 850 നാനോപോർ മുഴുനീള ട്രാൻസ്ക്രിപ്റ്റോം പ്രോജക്റ്റുകൾ പൂർത്തിയാക്കി 8,000-ലധികം സാമ്പിളുകൾ പ്രോസസ്സ് ചെയ്ത ഞങ്ങളുടെ ടീം ഓരോ പ്രോജക്റ്റിനും അനുഭവ സമ്പത്ത് നൽകുന്നു.
●വിൽപ്പനാനന്തര പിന്തുണ: ഞങ്ങളുടെ പ്രതിബദ്ധത 3 മാസത്തെ വിൽപ്പനാനന്തര സേവന കാലയളവിനൊപ്പം പ്രോജക്റ്റ് പൂർത്തീകരണത്തിനപ്പുറമാണ്. ഈ സമയത്ത്, ഞങ്ങൾ പ്രോജക്റ്റ് ഫോളോ-അപ്പ്, ട്രബിൾഷൂട്ടിംഗ് സഹായം, ഫലങ്ങളുമായി ബന്ധപ്പെട്ട ഏത് ചോദ്യങ്ങളും പരിഹരിക്കുന്നതിന് ചോദ്യോത്തര സെഷനുകൾ എന്നിവ വാഗ്ദാനം ചെയ്യുന്നു.
സാമ്പിൾ ആവശ്യകതകളും ഡെലിവറിയും
| ലൈബ്രറി | ക്രമപ്പെടുത്തൽ തന്ത്രം | ഡാറ്റ ശുപാർശ ചെയ്യുന്നു | ഗുണനിലവാര നിയന്ത്രണം |
| പോളി എ സമ്പുഷ്ടമാക്കി | ഇല്ലുമിന PE150 | 6/12 ജിബി | ശരാശരി ഗുണനിലവാര സ്കോർ: Q10 |
സാമ്പിൾ ആവശ്യകതകൾ:
ന്യൂക്ലിയോടൈഡുകൾ:
| Conc.(ng/μl) | തുക (μg) | ശുദ്ധി | സമഗ്രത |
| ≥ 100 | ≥ 1.0 | OD260/280=1.7-2.5 OD260/230=0.5-2.5 ജെല്ലിൽ കാണിച്ചിരിക്കുന്ന പ്രോട്ടീനോ DNA മലിനീകരണമോ പരിമിതമോ ഇല്ലയോ. | ചെടികൾക്ക്: RIN≥7.0; മൃഗങ്ങൾക്ക്: RIN≥7.5; 5.0≥28S/18S≥1.0; പരിമിതമായ അല്ലെങ്കിൽ അടിസ്ഥാനപരമായ ഉയരം ഇല്ല |
● സസ്യങ്ങൾ:
റൂട്ട്, തണ്ട് അല്ലെങ്കിൽ ഇതളുകൾ: 450 മില്ലിഗ്രാം
ഇല അല്ലെങ്കിൽ വിത്ത്: 300 മില്ലിഗ്രാം
ഫലം: 1.2 ഗ്രാം
● മൃഗം:
ഹൃദയം അല്ലെങ്കിൽ കുടൽ: 300 മില്ലിഗ്രാം
വിസെറ അല്ലെങ്കിൽ മസ്തിഷ്കം: 240 മില്ലിഗ്രാം
പേശി: 450 മില്ലിഗ്രാം
അസ്ഥികൾ, മുടി അല്ലെങ്കിൽ ചർമ്മം: 1 ഗ്രാം
● ആർത്രോപോഡുകൾ:
പ്രാണികൾ: 6 ഗ്രാം
ക്രസ്റ്റേഷ്യ: 300 മില്ലിഗ്രാം
● മുഴുവൻ രക്തം: 1 ട്യൂബ്
● സെല്ലുകൾ: 106 കോശങ്ങൾ
ശുപാർശ ചെയ്യുന്ന സാമ്പിൾ ഡെലിവറി
കണ്ടെയ്നർ: 2 മില്ലി സെൻട്രിഫ്യൂജ് ട്യൂബ് (ടിൻ ഫോയിൽ ശുപാർശ ചെയ്യുന്നില്ല)
സാമ്പിൾ ലേബലിംഗ്: ഗ്രൂപ്പ്+റെപ്ലിക്കേറ്റ് ഉദാ A1, A2, A3; B1, B2, B3.
കയറ്റുമതി:
1. ഡ്രൈ-ഐസ്: സാമ്പിളുകൾ ബാഗുകളിൽ പായ്ക്ക് ചെയ്യുകയും ഡ്രൈ-ഐസിൽ കുഴിച്ചിടുകയും വേണം.
2. ആർഎൻഎ-സ്റ്റബിൾ ട്യൂബുകൾ: ആർഎൻഎ സാമ്പിളുകൾ ആർഎൻഎ സ്റ്റബിലൈസേഷൻ ട്യൂബിൽ ഉണക്കി മുറിയിലെ ഊഷ്മാവിൽ അയയ്ക്കാം.
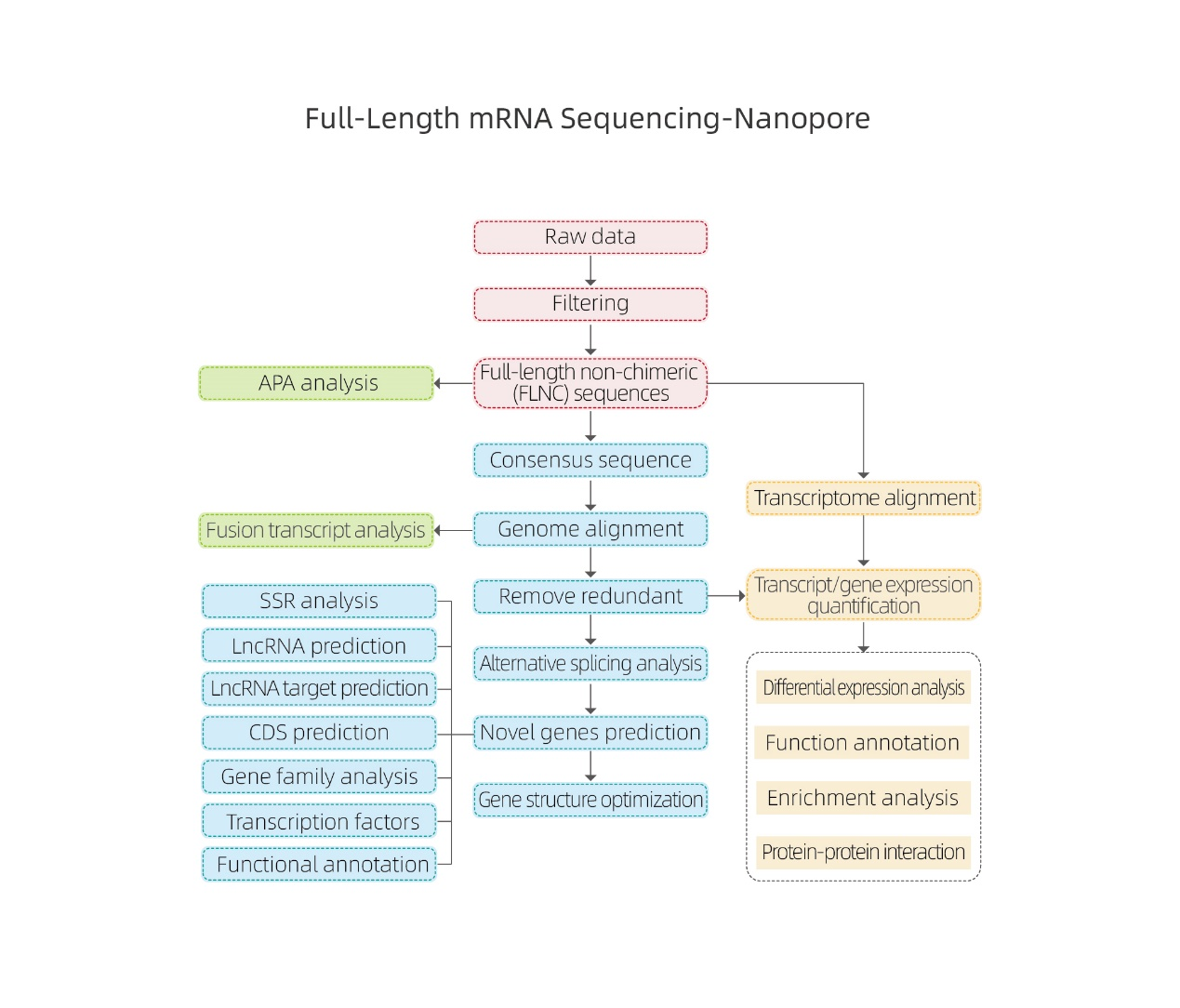
സർവീസ് വർക്ക് ഫ്ലോ
ന്യൂക്ലിയോടൈഡുകൾ:

സാമ്പിൾ ഡെലിവറി

ലൈബ്രറി നിർമ്മാണം

സീക്വൻസിങ്

ഡാറ്റ വിശകലനം

വിൽപ്പനാനന്തര സേവനങ്ങൾ
സർവീസ് വർക്ക് ഫ്ലോ
ടിഷ്യു:

പരീക്ഷണ രൂപകൽപ്പന

സാമ്പിൾ ഡെലിവറി

ആർഎൻഎ വേർതിരിച്ചെടുക്കൽ

ലൈബ്രറി നിർമ്മാണം

സീക്വൻസിങ്

ഡാറ്റ വിശകലനം

വിൽപ്പനാനന്തര സേവനങ്ങൾ
● റോ ഡാറ്റ പ്രോസസ്സിംഗ്
● ട്രാൻസ്ക്രിപ്റ്റ് തിരിച്ചറിയൽ
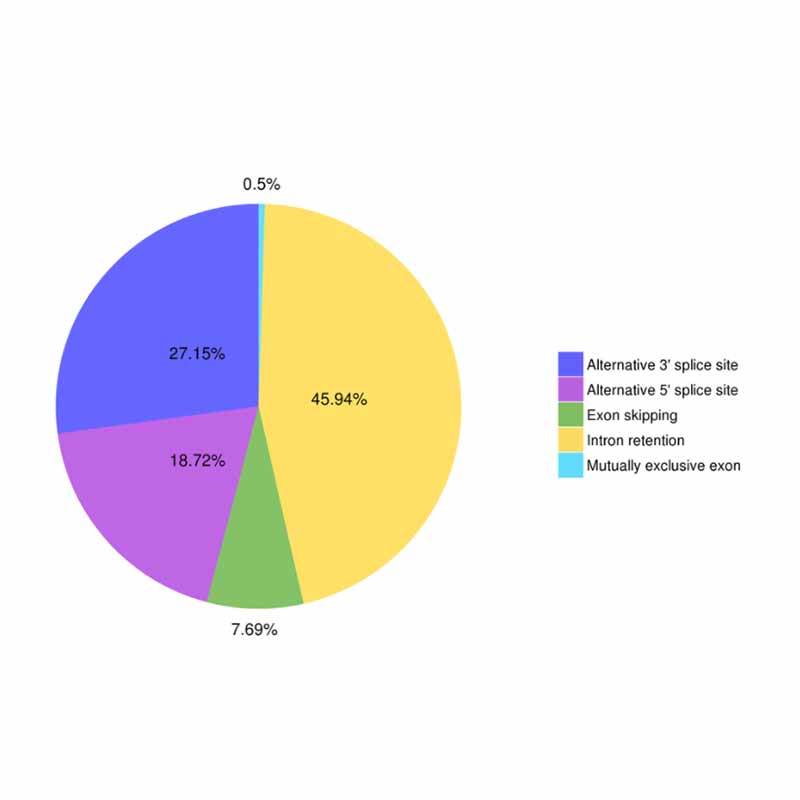
● ഇതര വിഭജനം
● ജീൻ ലെവലിലും ഐസോഫോം ലെവലിലും എക്സ്പ്രഷൻ ക്വാണ്ടിഫിക്കേഷൻ
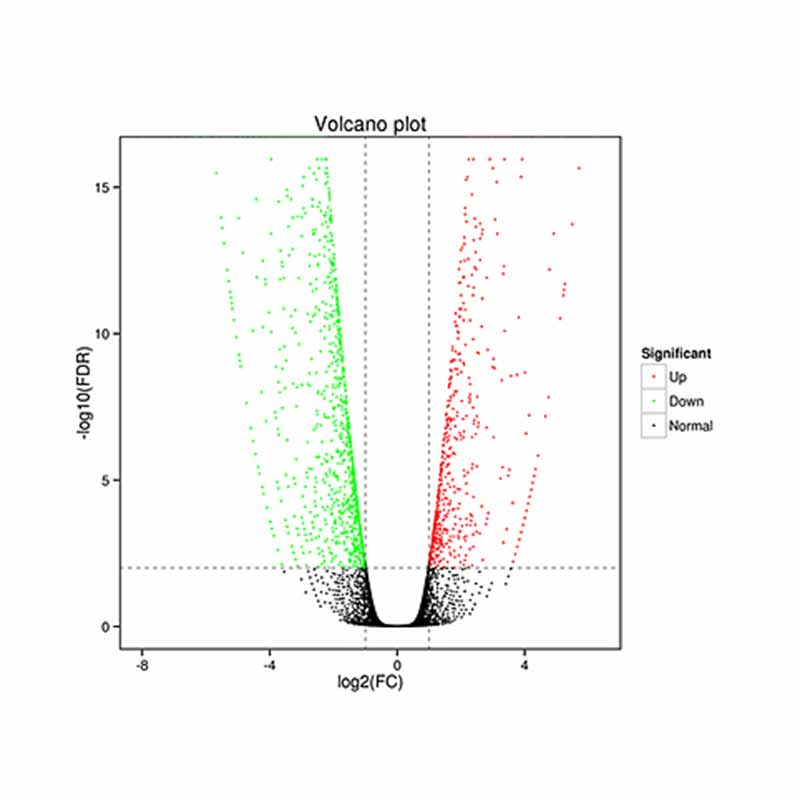
● ഡിഫറൻഷ്യൽ എക്സ്പ്രഷൻ വിശകലനം
● പ്രവർത്തന വ്യാഖ്യാനവും സമ്പുഷ്ടീകരണവും (DEG-കളും DET-കളും)
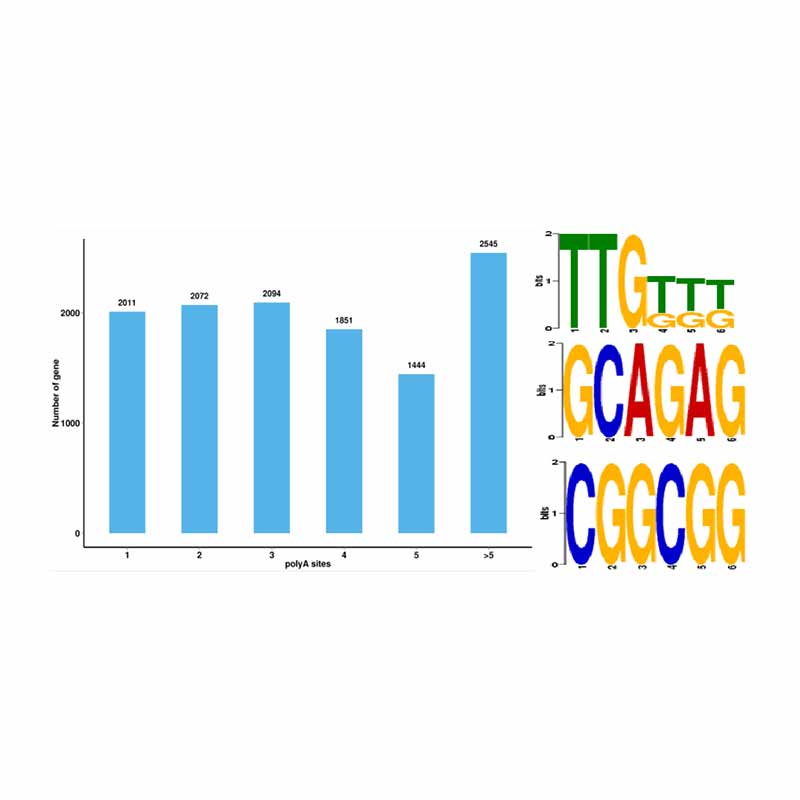
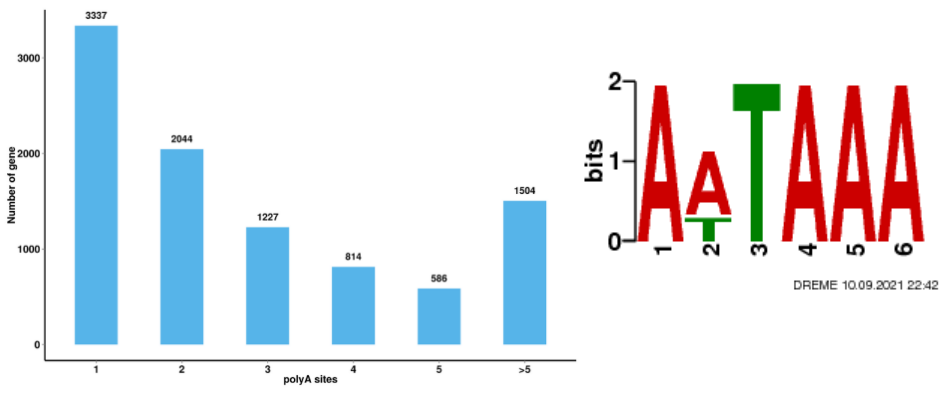
ഇതര വിഭജന വിശകലനം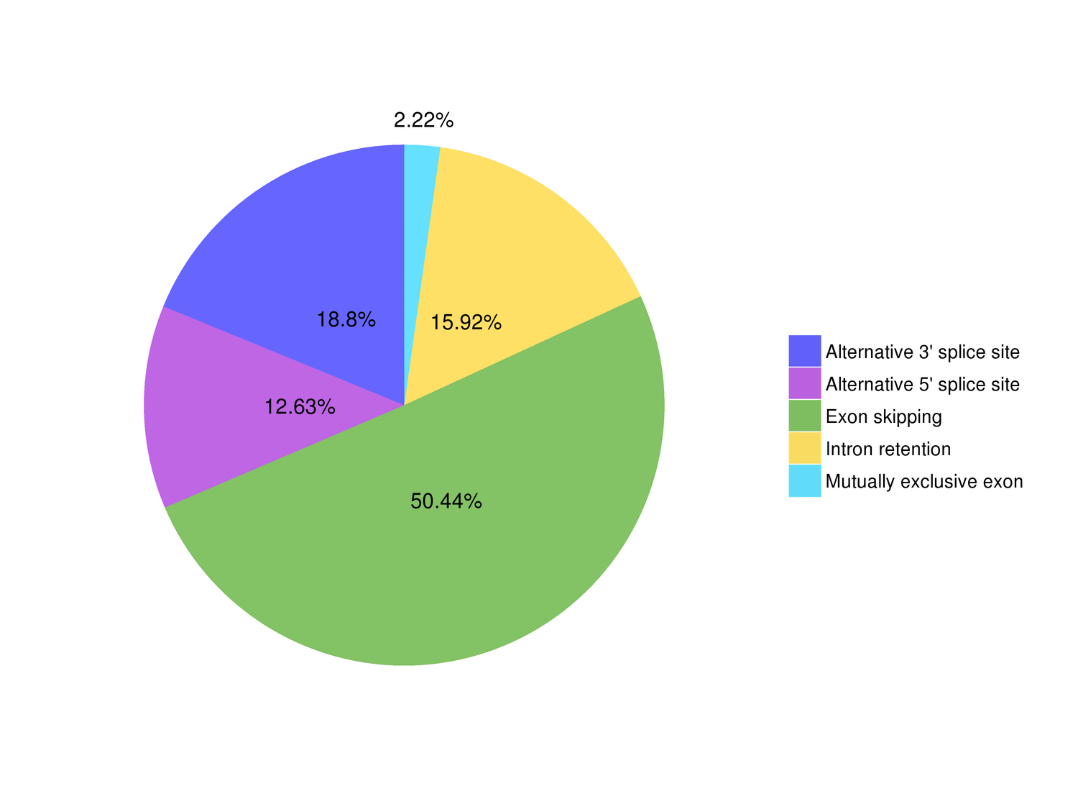 ആൾട്ടർനേറ്റീവ് പോളിഡെനൈലേഷൻ അനാലിസിസ് (APA)
ആൾട്ടർനേറ്റീവ് പോളിഡെനൈലേഷൻ അനാലിസിസ് (APA)
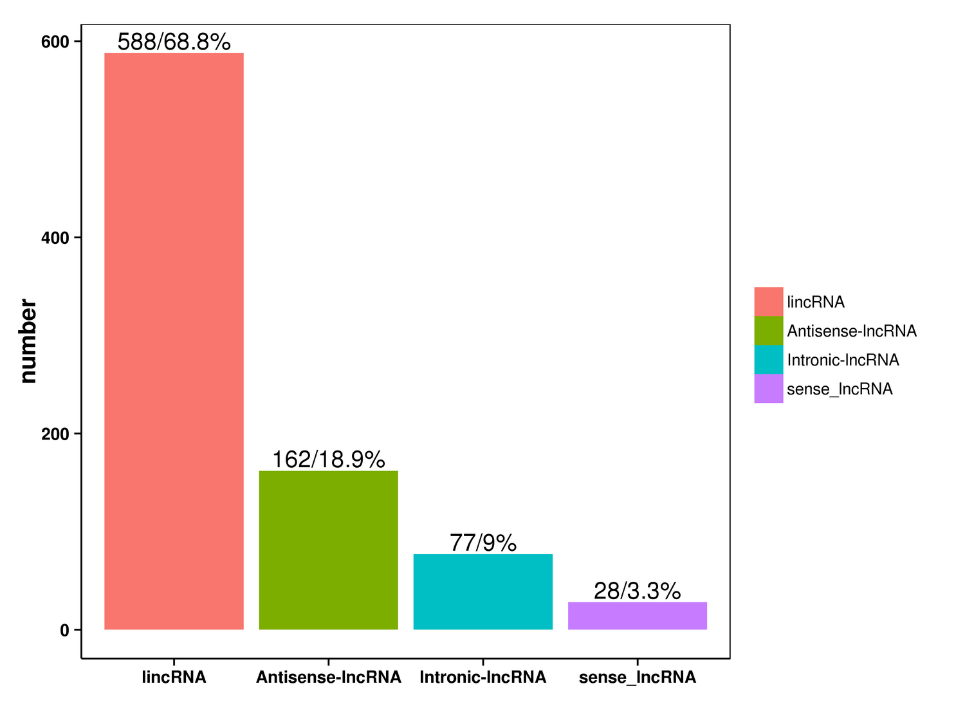
lncRNA പ്രവചനം
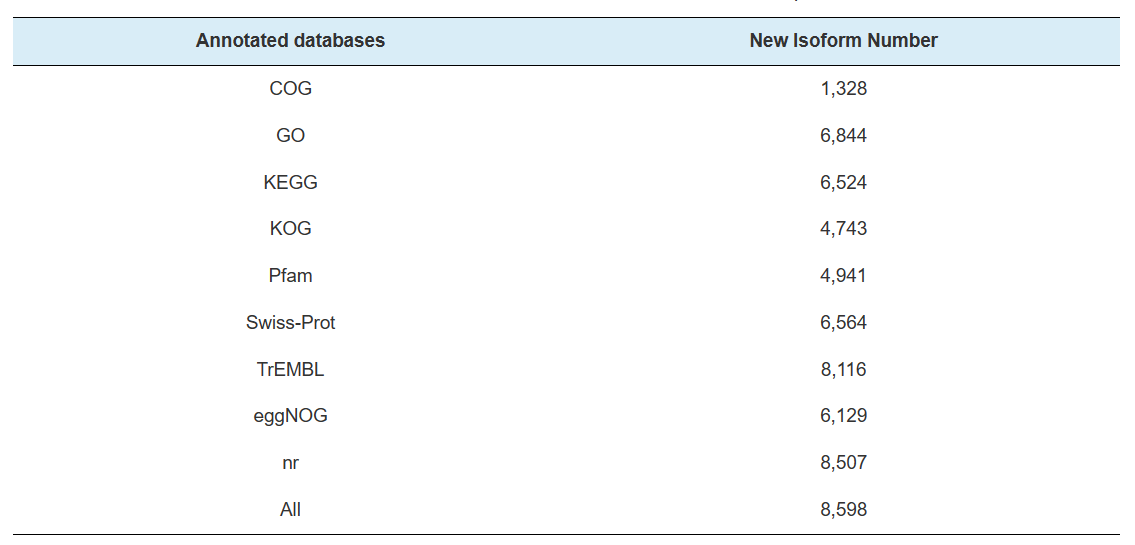
നോവൽ ജീനുകളുടെ വ്യാഖ്യാനം
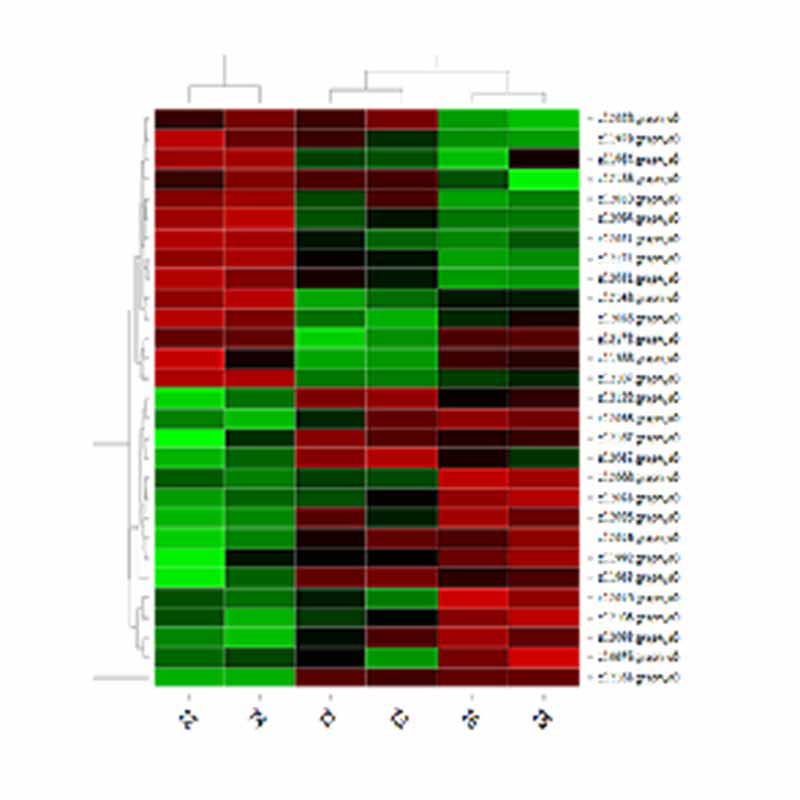
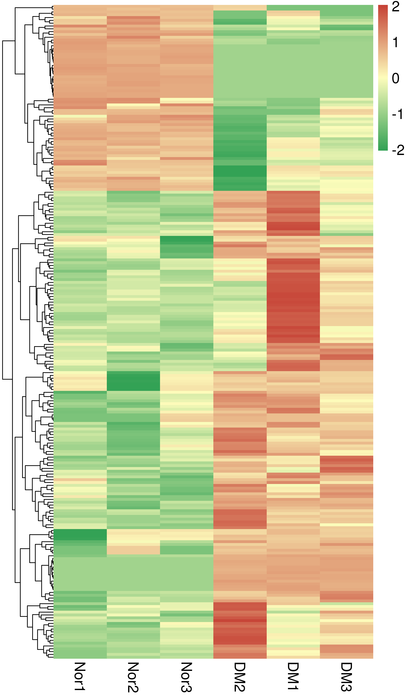
ഡിഇടികളുടെ ക്ലസ്റ്ററിംഗ്
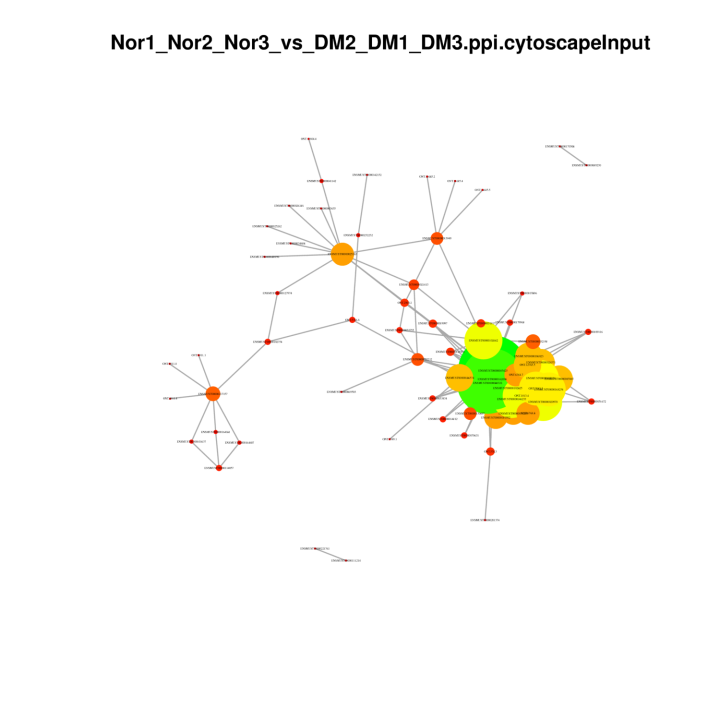
DEG-കളിലെ പ്രോട്ടീൻ-പ്രോട്ടീൻ നെറ്റ്വർക്കുകൾ
പ്രസിദ്ധീകരണങ്ങളുടെ ക്യൂറേറ്റ് ചെയ്ത ശേഖരത്തിലൂടെ BMKGene-ൻ്റെ നാനോപോർ മുഴുനീള mRNA സീക്വൻസിങ് സേവനങ്ങൾ വഴി സുഗമമാക്കിയ പുരോഗതികൾ പര്യവേക്ഷണം ചെയ്യുക.
ഗോങ്, ബി. തുടങ്ങിയവർ. (2023) 'ഗ്ലിയോമയിലെ ഓങ്കോജീനായി FAM20C സ്രവിക്കുന്ന കൈനസിൻ്റെ എപ്പിജെനെറ്റിക് ആൻഡ് ട്രാൻസ്ക്രിപ്ഷണൽ ആക്റ്റിവേഷൻ', ജേണൽ ഓഫ് ജനറ്റിക്സ് ആൻഡ് ജെനോമിക്സ്, 50(6), പേജ്. 422–433. doi: 10.1016/J.JGG.2023.01.008.
അവൻ, Z. et al. (2023) 'ലിംഫോസൈറ്റുകളുടെ ഫുൾ-ലെങ്ത് ട്രാൻസ്ക്രിപ്റ്റോം സീക്വൻസിങ് IFN-γ ലേക്ക് പ്രതികരിക്കുന്നത് Th1-skewed immune response in flounder (Paralichthys olivaceus)', Fish & Shellfish Immunology, 134, p. 108636. doi: 10.1016/J.FSI.2023.108636.
Ma, Y. et al. (2023) 'Nemopilema Nomurai വിഷ ഐഡൻ്റിഫിക്കേഷനായുള്ള PacBio, ONT RNA സീക്വൻസിങ് രീതികളുടെ താരതമ്യ വിശകലനം', ജീനോമിക്സ്, 115(6), പേ. 110709. doi: 10.1016/J.YGENO.2023.110709.
യു, ഡി. തുടങ്ങിയവർ. (2023) 'HUMSC-ൽ നിന്ന് ഉരുത്തിരിഞ്ഞ എക്സോസോമുകളും മൈക്രോവെസിക്കിളുകളും തമ്മിലുള്ള വ്യത്യസ്ത പ്രവർത്തന പ്രവണതയെ നാനോ-സെക് വിശകലനം വെളിപ്പെടുത്തുന്നു', സ്റ്റെം സെൽ റിസർച്ച് ആൻഡ് തെറാപ്പി, 14(1), പേജ്. 1–13. doi: 10.1186/S13287-023-03491-5/TABLES/6.