Visa transkripta sekvencēšana — Illumina
Funkcijas
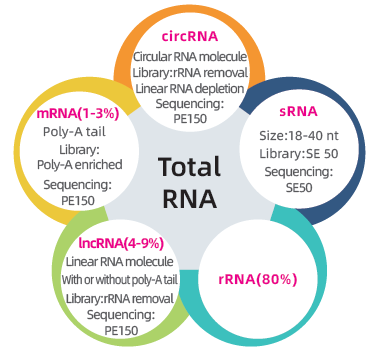
● Dubultā bibliotēka, lai secinātu visu transkriptu: rRNS izsīkšana, kam seko PE150 bibliotēkas sagatavošana un lieluma izvēle, kam seko SE50 bibliotēkas sagatavošana
● Pilnīga mRNS, lncRNS, cirRNS un miRNS bioinformātikas analīze atsevišķos bioinformātikas pārskatos
● Kopīga visas RNS ekspresijas analīze apvienotā ziņojumā, tostarp ceRNS tīklu analīze.
Pakalpojuma priekšrocības
●Regulēšanas tīklu padziļināta analīze: ceRNS tīkla analīzi nodrošina kopīga mRNS, lncRNS, cirRNS un miRNS sekvencēšana un izsmeļoša bioinformātiskā darbplūsma.
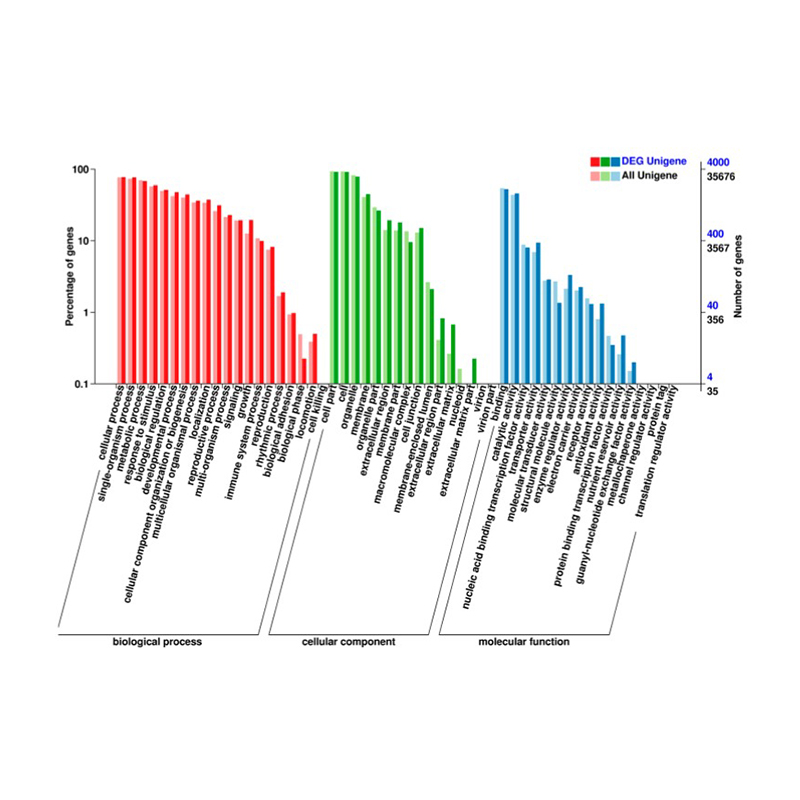
●Visaptveroša anotācija: Mēs izmantojam vairākas datu bāzes, lai funkcionāli anotētu diferenciāli ekspresētos gēnus (DEG) un veiktu atbilstošo bagātināšanas analīzi, sniedzot ieskatu šūnu un molekulārajos procesos, kas ir transkripta reakcijas pamatā.
●Plaša ekspertīze: Mūsu komanda ir veiksmīgi noslēgusi vairāk nekā 2100 visu transkripta projektus dažādās pētniecības jomās, tāpēc mūsu komanda sniedz bagātīgu pieredzi katrā projektā.
●Stingra kvalitātes kontrole: Mēs ieviešam galvenos kontroles punktus visos posmos, sākot no paraugu un bibliotēkas sagatavošanas līdz sekvencēšanai un bioinformātikai. Šī rūpīgā uzraudzība nodrošina nemainīgi augstas kvalitātes rezultātu piegādi.
●Pēcpārdošanas atbalsts: Mūsu saistības sniedzas tālāk par projekta pabeigšanu ar 3 mēnešu pēcpārdošanas servisa periodu. Šajā laikā mēs piedāvājam projekta pārraudzību, problēmu novēršanas palīdzību un jautājumu un atbilžu sesijas, lai atbildētu uz visiem jautājumiem, kas saistīti ar rezultātiem.
Prasību paraugs un piegāde
| Bibliotēka | Sekvences stratēģija | Ieteicamie dati | Kvalitātes kontrole |
| rRNS ir izsmelta | Illumina PE150 | 16 Gb | Q30≥85% |
| Izmērs izvēlēts | Illumina SE50 | 10-20 miljoni nolasījumu |
Prasību paraugs:
Nukleotīdi:
| Konc. (ng/μl) | Daudzums (μg) | Tīrība | Integritāte |
| ≥ 80 | ≥ 1,6 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Uz gēla parādīts ierobežots proteīnu vai DNS piesārņojums vai tā nav. | RIN≥6,0 5,0≥28S/18S≥1,0; ierobežots vai vispār nav bāzes līmeņa pacēluma |
Ieteicamā paraugu piegāde
Tvertne: 2 ml centrifūgas caurule (skārda folija nav ieteicama)
Marķējuma paraugs: grupa + atkārtojums, piemēram, A1, A2, A3; B1, B2, B3.
Sūtījums:
1. Sausais ledus: paraugi jāiepako maisos un jāierok sausajā ledū.
2. RNAstable caurules: RNS paraugus var žāvēt RNS stabilizācijas mēģenē (piemēram, RNAstable®) un nosūtīt istabas temperatūrā.
Pakalpojuma darba plūsma

Eksperimenta dizains

Paraugu piegāde

RNS ekstrakcija

Bibliotēkas celtniecība

Secība

Datu analīze

Pēcpārdošanas pakalpojumi
Bioinformātika
RNS ekspresijas pārskats
Atšķirīgi izteikti gēni
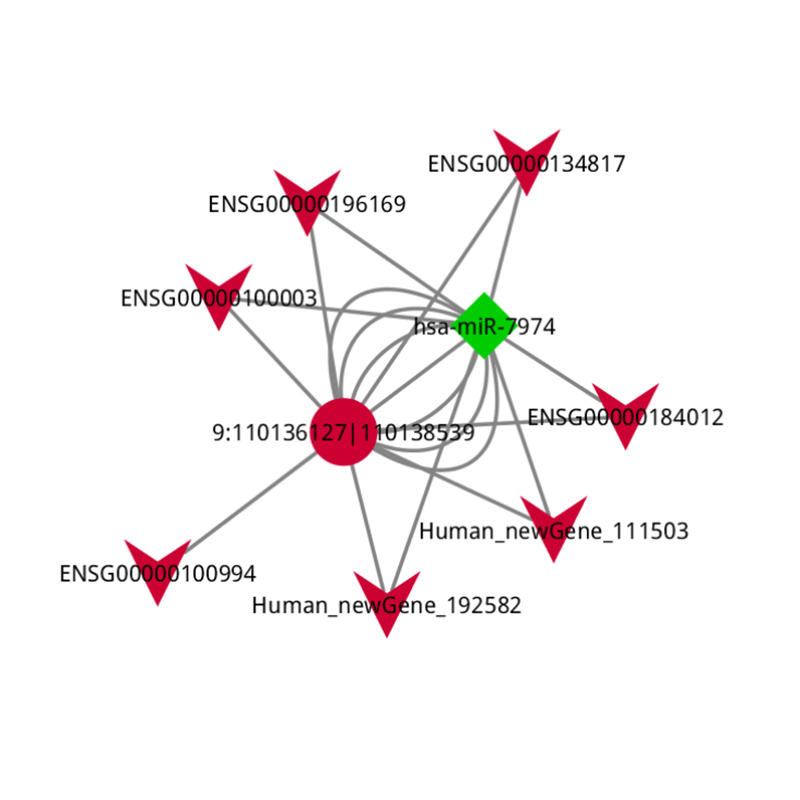
ceRNS analīze
Izpētiet pētniecības sasniegumus, ko veicina visi BMKGene transkriptu sekvencēšanas pakalpojumi, izmantojot apkopotu publikāciju kolekciju.
Dai, Y. et al. (2022) “Visaptveroši mRNS, lncRNS un miRNS ekspresijas profili Kašina-Beka slimībā, kas identificēta ar RNS sekvencēšanu”, Molecular Omics, 18(2), 154.–166. lpp. doi: 10.1039/D1MO00370D.
Liu, N. nan et al. (2022) “Apis cerana aukstumizturības pilna garuma transkriptu analīze Čangbai kalnā ziemošanas periodā.”, Gene, 830, 146503.–146503. lpp. doi: 10.1016/J.GENE.2022.146503.
Wang, XJ et al. (2022) “Uz vairāku omiku integrāciju balstīta prioritāšu noteikšana konkurējošiem endogēnās RNS regulēšanas tīkliem sīkšūnu plaušu vēža gadījumā: molekulārās īpašības un zāļu kandidāti”, Frontiers in Oncology, 12. lpp. 904865. doi: 10.3389/FONC.2022.904865/BIBTEX.
Xu, P. et al. (2022) "Integrētā lncRNS/cirRNS-miRNS-mRNS ekspresijas profilu analīze atklāj jaunus ieskatus potenciālajos mehānismos, reaģējot uz sakņu nematodēm zemesriekstos", BMC Genomics, 23(1), 1.–12. lpp. doi: 10.1186/S12864-022-08470-3/FIGURES/7.
Yan, Z. et al. (2022) “Visa transkripta RNS sekvencēšana izceļ molekulāros mehānismus, kas saistīti ar brokoļu kvalitātes uzturēšanu pēc ražas novākšanas, izmantojot sarkano LED apstarošanu”, Postharvest Biology and Technology, 188. lpp. 111878. doi: 10.1016/J.POSTHARVBIO.2022.111878.