Maza RNS sekvencēšanas-illumina
Funkcijas
● Bibliotēkas sagatavošana ietver lieluma izvēles soli
● Bioinformātiskā analīze, kas vērsta uz miRNA prognozēšanu un to mērķiem
Pakalpojumu priekšrocības
●Visaptveroša bioinformātikas analīze:Ļaujot identificēt gan zināmas, gan jaunas miRNS, miRNS mērķu identificēšanu un atbilstošu funkcionālo anotāciju un bagātināšanu ar vairākām datu bāzēm (Kegg, Go)
●Stingra kvalitātes kontrole: Mēs ieviešam galvenos vadības punktus visos posmos, sākot no parauga un bibliotēkas sagatavošanas līdz secībai un bioinformātikai. Šī rūpīgā uzraudzība nodrošina pastāvīgi augstas kvalitātes rezultātu piegādi.
●Pēcpārdošanas atbalsts: Mūsu apņemšanās pārsniedz projekta pabeigšanu ar 3 mēnešu pēcpārdošanas dienesta periodu. Šajā laikā mēs piedāvājam projektu turpinājumu, palīdzības novēršanu un Q&A sesijas, lai risinātu visus rezultātus saistītos jautājumus.
●Plaša kompetence: Veicot veiksmīgu vairāku SRNA projektu slēgšanu, kas aptver vairāk nekā 300 sugu dažādās pētniecības jomās, mūsu komanda katram projektam sniedz daudz pieredzes.
Parauga prasības un piegāde
| Bibliotēka | Platforma | Ieteicamie dati | Data QC |
| Atlasītais izmērs | Illumina SE50 | 10 m-20m lasījumi | Q30≥85% |
Parauga prasības:
Nukleotīdi:
| Konc. (Ng/μl) | Daudzums (μg) | Tīrība | Godīgums |
| ≥ 80 | ≥ 0,8 | OD260/280 = 1,7–2,5 OD260/230 = 0,5–2,5 Ierobežots vai bez olbaltumvielu vai DNS piesārņojuma, kas parādīts uz gēla. | Rin≥6,0; 5,0 ≥28s/18S≥1,0; ierobežots vai nav sākotnējais augstums |
● Augi:
Sakne, kāts vai ziedlapa: 450 mg
Lapa vai sēkla: 300 mg
Augļi: 1,2 g
● Dzīvnieks:
Sirds vai zarnas: 450 mg
Iekšējo orgeru vai smadzenes: 240 mg
Muskulis: 600 mg
Kauli, mati vai āda: 1,5 g
● Piesaista:
Kukaiņi: 9g
Crustacea: 450 mg
● pilnas asinis: 2 caurules
● šūnas: 106 šūnās
● serums un plazma:6 ml
Ieteicamā parauga piegāde
Konteiners: 2 ml centrifūgas caurule (nav ieteicams alvas folija)
Parauga marķēšana: grupa+replicate, piemēram, A1, A2, A3; B1, B2, B3.
Sūtījums:
1. Sausais ledus: Paraugi jāiesaiņo maisos un jāapglabā sausā ledus.
2. RNASTABLE CEĻAS: RNS paraugus var žāvēt RNS stabilizācijas caurulē (piemēram, RNastable®) un nosūtīt istabas temperatūrā.
Servisa darba plūsma

Eksperimenta dizains

Paraugu piegāde

RNS ekstrakcija

Bibliotēkas celtniecība

Sekvencēšana

Datu analīze

Pēcpārdošanas pakalpojumi
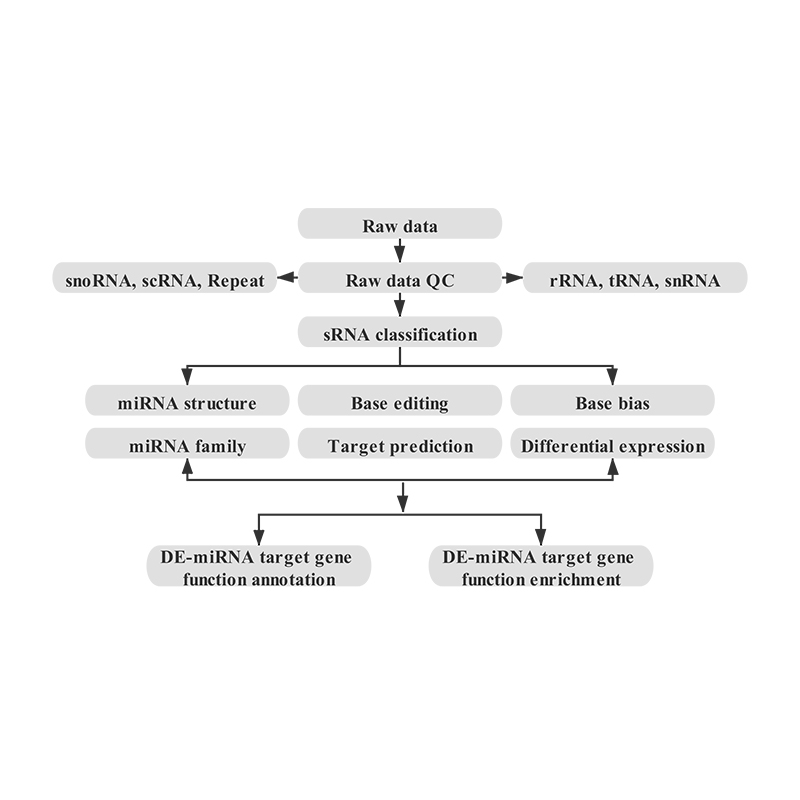
Bioinformātika
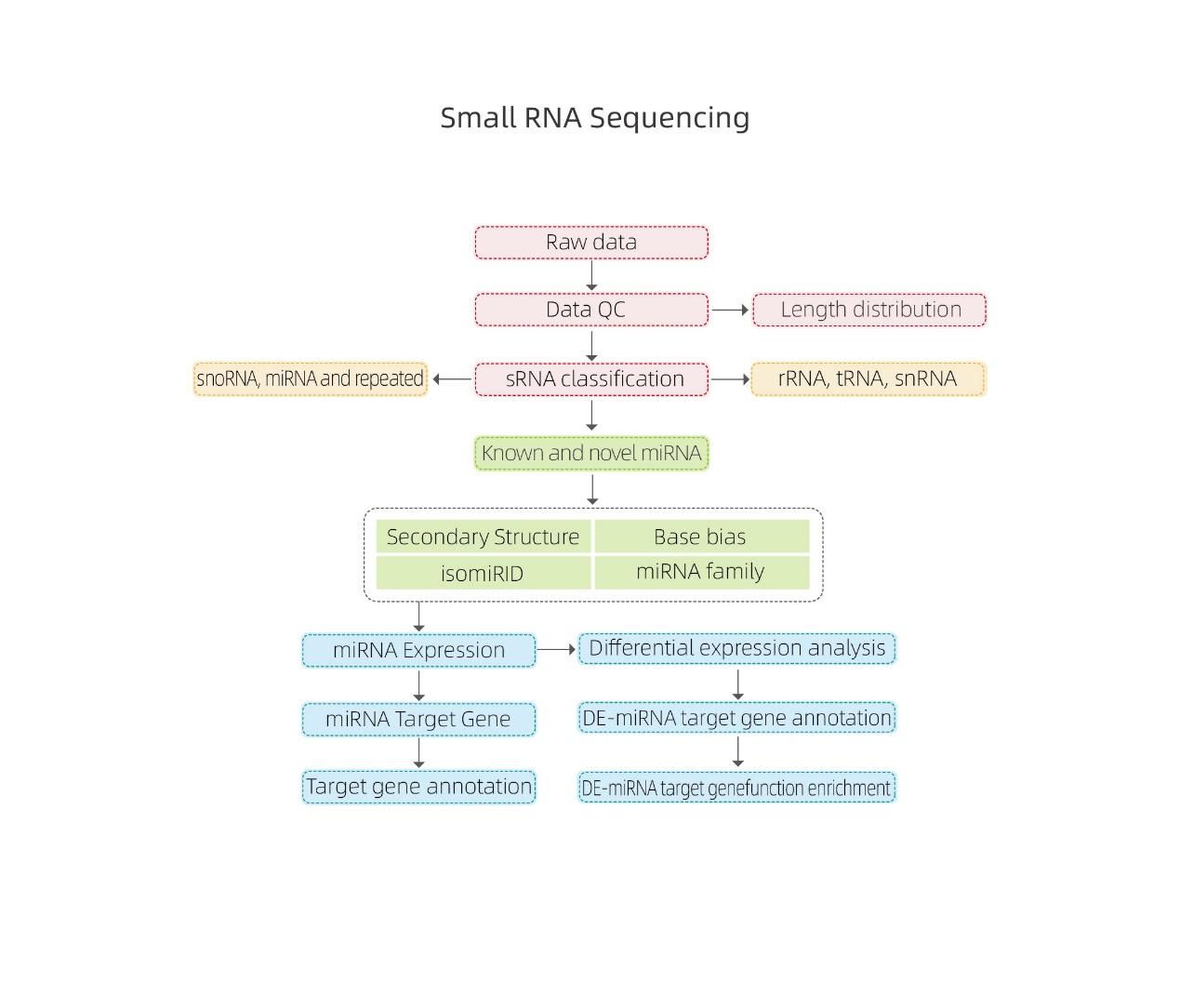 ● Neapstrādātu datu kvalitātes kontrole
● Neapstrādātu datu kvalitātes kontrole
● SRNA klasifikācija
● Izlīdzināšana ar atsauces genomu
● Zināmas un jaunas miRNA identificēšana
● Diferenciālā miRNA ekspresijas analīze
● MiRNA mērķu funkcionālā anotācija
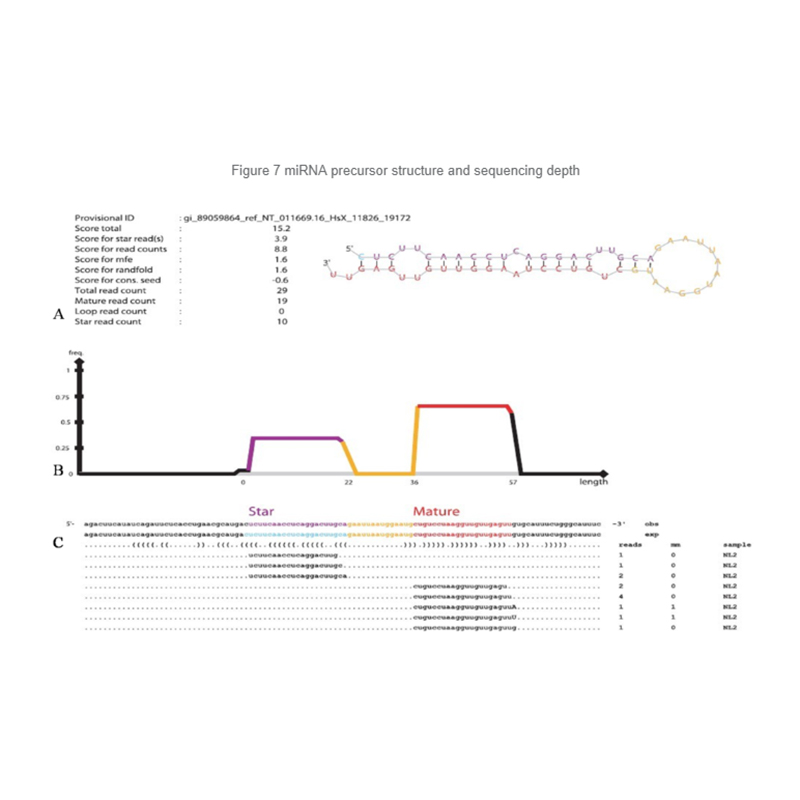
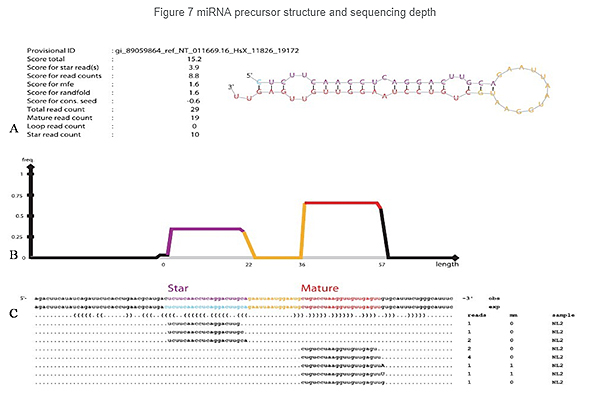
MiRNS identifikācija: struktūra un dziļums
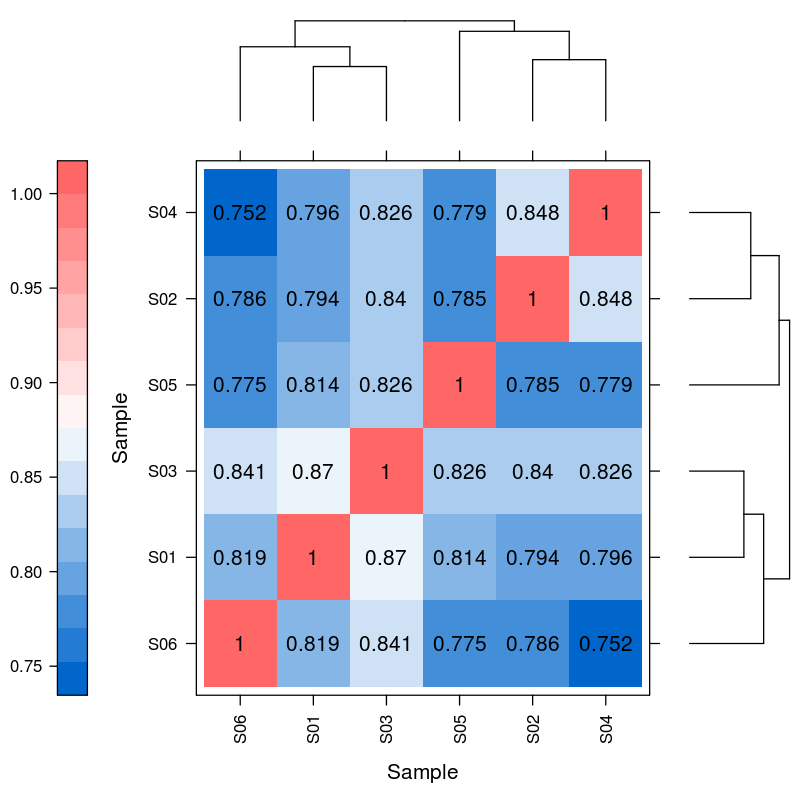
MiRNS diferenciālā ekspresija - hiearchical klasterizācija
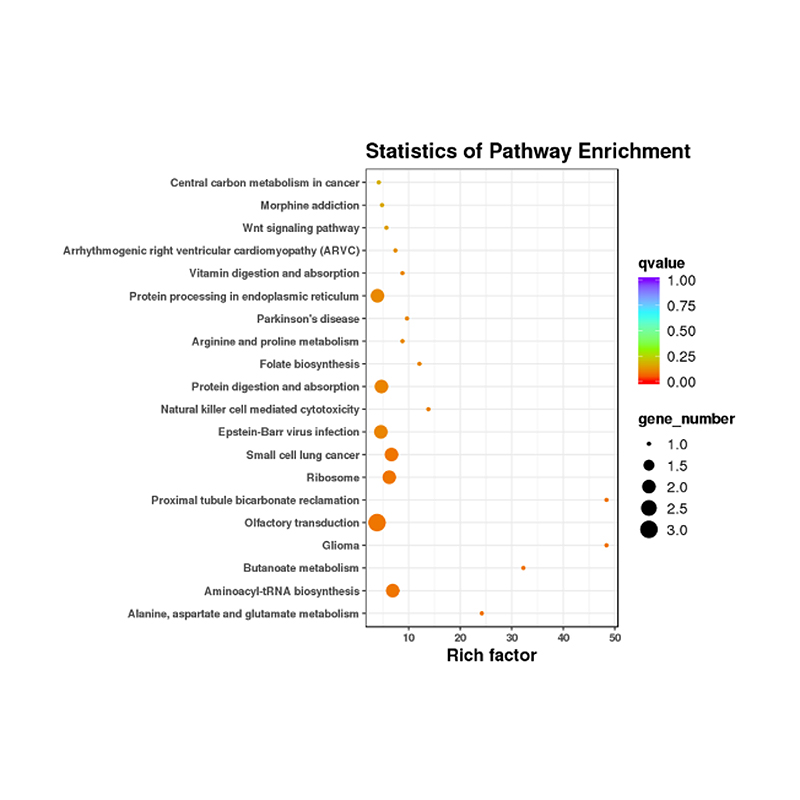
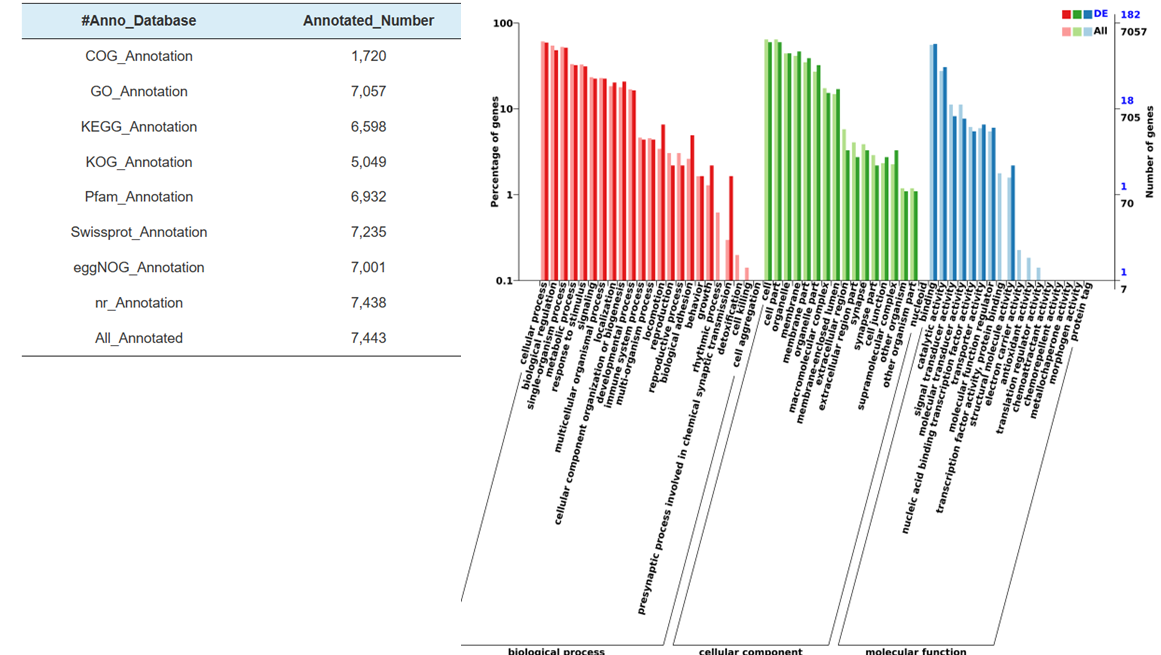
Diferenciāli izteiktu miRNS mērķa funkcionālā anotācija
Izpētiet pētījumu sasniegumus, ko veicina BMKGENE SRNA sekvencēšanas pakalpojumi, izmantojot kuratoru publikāciju kolekciju.
Chen, H. et al. (2023) “Vīrusu infekcijas kavē saponīna biosintēzi un fotosintēzi Panax Notoginseng”, augu fizioloģijā un bioķīmijā, 203, lpp. 108038. Doi: 10.1016/j.plaphy.2023.108038.
Li, H. et al. (2023) “Augu FYVE domēnu saturošais olbaltumvielu brīvais1 asociējas ar mikroprocesora komponentiem, lai apspiestu miRNA bioģenēzi”, EMBO ziņojumi, 24 (1). doi: 10.15252/emb.202255037/Suppl_file/Emmr202255037-Sup-0004-sdatafig4.tif.
Yu, J. et al. (2023) “MikroRNA AME-BANTAM-3P kontrolē kāpuru skolēna attīstību, mērķējot uz vairākiem epidermas augšanas faktoriem līdzīgajiem domēniem 8 gēns (MEGF8) medus bišu, Apis Mellifera”, Starptautiskais molekulāro zinātņu žurnāls, 24 (6), P Apvidū 5726. DOI: 10.3390/IJMS24065726/S1.
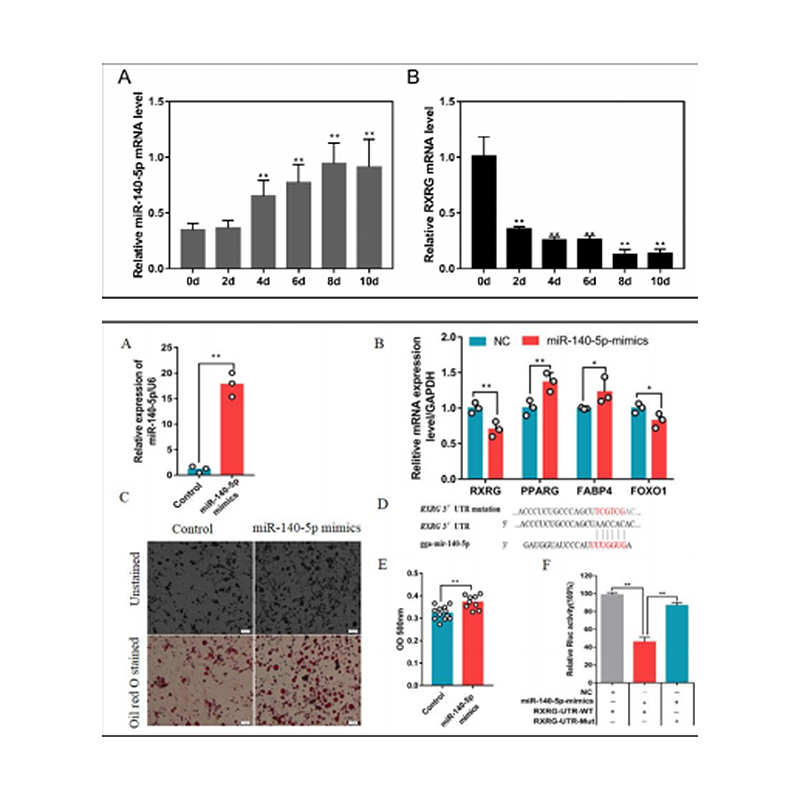
Džan, M. et al. (2018) “MiRNS un gēnu integrētā analīze, kas saistīta ar gaļas kvalitāti, atklāj, ka GGA-MIR-140-5P ietekmē intramuskulāro tauku nogulsnēšanos cāļu, šūnu fizioloģijā un bioķīmijā, 46 (6), 2421.-2433. Lpp. doi: 10.1159/000489649.