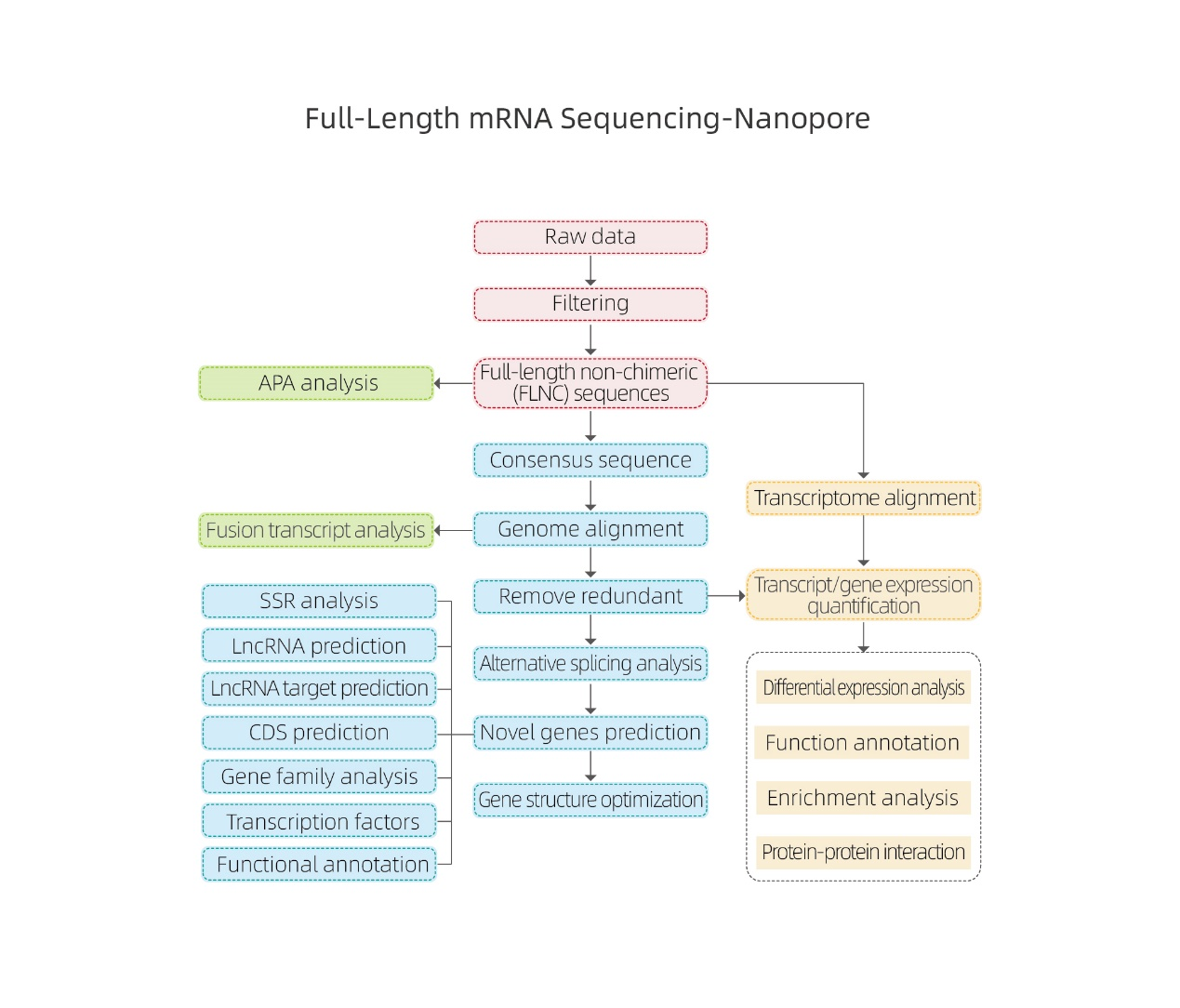
Pilna garuma mRNS sekvencēšana-nanopore
Funkcijas
● Poli-A mRNS uztveršana, kam seko cDNS sintēze un bibliotēkas sagatavošana
● Pilna garuma atšifrējumu secība
● Bioinformātiskā analīze, kuras pamatā ir saskaņošana ar atsauces genomu
● Bioinformātiskā analīze ietver ne tikai ekspresiju gēnu un izoformu līmenī, bet arī lncRNS, gēnu saplūšanas, poliadenilācijas un gēnu struktūras analīzi.
Pakalpojuma priekšrocības
●Izteiksmes kvantitatīva noteikšana izoformas līmenī: ļauj veikt detalizētu un precīzu ekspresijas analīzi, atklājot izmaiņas, kas var tikt maskētas, analizējot visu gēna ekspresiju
●Samazinātas datu prasības:Salīdzinot ar nākamās paaudzes sekvencēšanu (NGS), Nanopore sekvencēšanai ir zemākas datu prasības, kas ļauj nodrošināt līdzvērtīgus gēnu ekspresijas kvantitatīvās piesātinājuma līmeņus ar mazākiem datiem.
●Augstāka izteiksmes kvantitatīvās noteikšanas precizitāte: gan gēnu, gan izoformu līmenī
●Papildu transkriptomiskās informācijas identificēšana: alternatīva poliadenilācija, saplūšanas gēni un lcnRNS un to mērķa gēni
●Plaša ekspertīze: Mūsu komanda sniedz bagātīgu pieredzi katrā projektā, pabeidzot vairāk nekā 850 Nanopore pilna garuma transkripta projektus un apstrādājot vairāk nekā 8000 paraugu.
●Pēcpārdošanas atbalsts: mūsu saistības sniedzas tālāk par projekta pabeigšanu ar 3 mēnešu pēcpārdošanas servisa periodu. Šajā laikā mēs piedāvājam projekta pārraudzību, problēmu novēršanas palīdzību un jautājumu un atbilžu sesijas, lai risinātu visus ar rezultātiem saistītus vaicājumus.
Prasību paraugs un piegāde
| Bibliotēka | Sekvences stratēģija | Ieteicamie dati | Kvalitātes kontrole |
| Poly A bagātināts | Illumina PE150 | 6/12 Gb | Vidējais kvalitātes rādītājs: Q10 |
Prasību paraugs:
Nukleotīdi:
| Konc. (ng/μl) | Daudzums (μg) | Tīrība | Integritāte |
| ≥ 100 | ≥ 1,0 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Uz gēla parādīts ierobežots proteīnu vai DNS piesārņojums vai tā nav. | Augiem: RIN≥7,0; Dzīvniekiem: RIN≥7,5; 5,0≥28S/18S≥1,0; ierobežots vai vispār nav bāzes līmeņa pacēluma |
● Augi:
Sakne, stublājs vai ziedlapa: 450 mg
Lapa vai sēklas: 300 mg
Augļi: 1,2 g
● Dzīvnieks:
Sirds vai zarnas: 300 mg
Iekšējie orgāni vai smadzenes: 240 mg
Muskuļi: 450 mg
Kauli, mati vai āda: 1g
● Posmkāji:
Kukaiņi: 6g
Vēžveidīgie: 300 mg
● Pilnas asinis: 1 caurule
● Šūnas: 106 šūnas
Ieteicamā paraugu piegāde
Tvertne: 2 ml centrifūgas caurule (skārda folija nav ieteicama)
Marķējuma paraugs: grupa + atkārtojums, piemēram, A1, A2, A3; B1, B2, B3.
Sūtījums:
1. Sausais ledus: paraugi jāiepako maisos un jāierok sausajā ledū.
2. RNAstable caurules: RNS paraugus var žāvēt RNS stabilizācijas mēģenē (piemēram, RNAstable®) un nosūtīt istabas temperatūrā.
Pakalpojuma darba plūsma
Nukleotīdi:

Paraugu piegāde

Bibliotēkas celtniecība

Secība

Datu analīze

Pēcpārdošanas pakalpojumi
Pakalpojuma darba plūsma
Audi:

Eksperimenta dizains

Paraugu piegāde

RNS ekstrakcija

Bibliotēkas celtniecība

Secība

Datu analīze

Pēcpārdošanas pakalpojumi
● Neapstrādātu datu apstrāde
● Atšifrējuma identifikācija
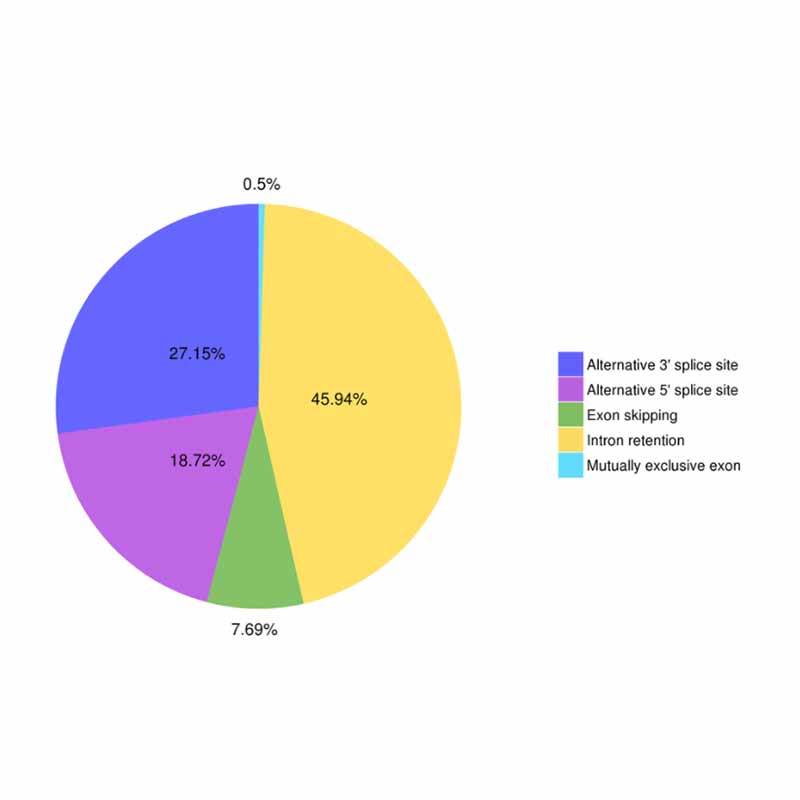
● Alternatīva savienošana
● Ekspresijas kvantitatīva noteikšana gēnu līmenī un izoformu līmenī
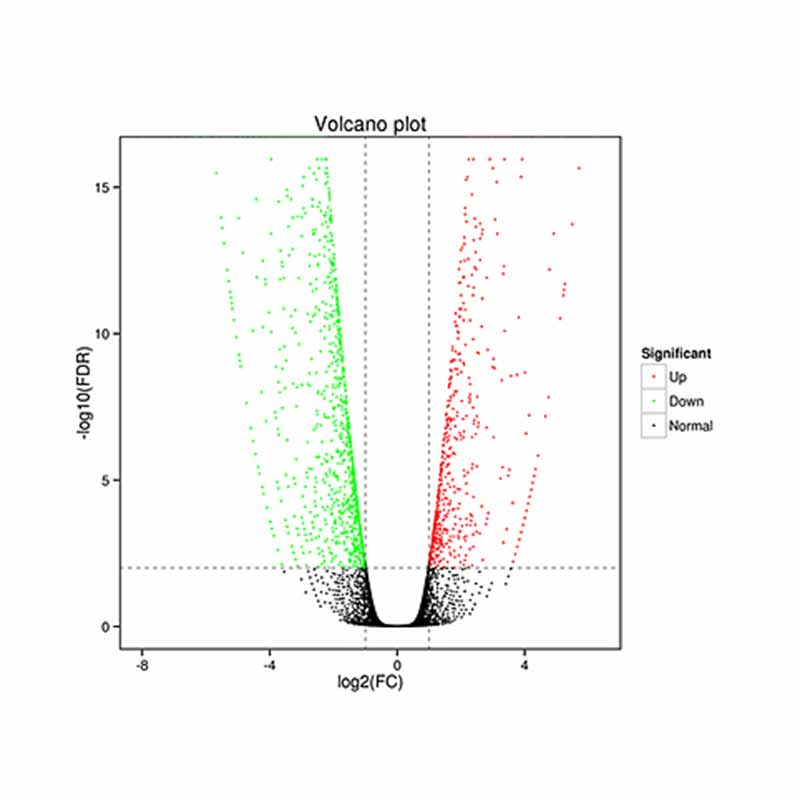
● Diferenciālās izteiksmes analīze
● Funkciju anotācija un bagātināšana (DEG un DET)
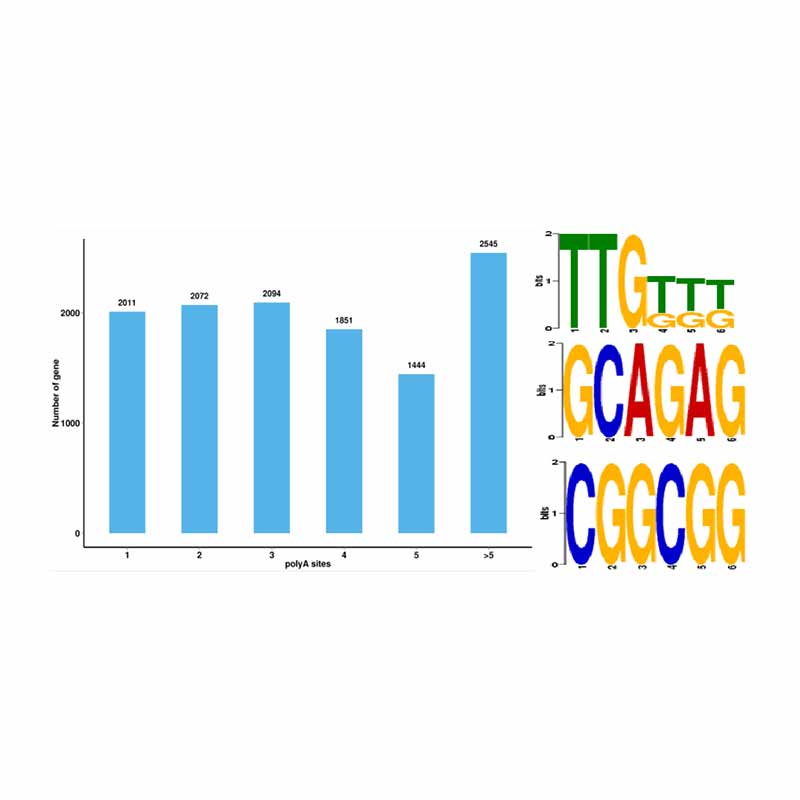
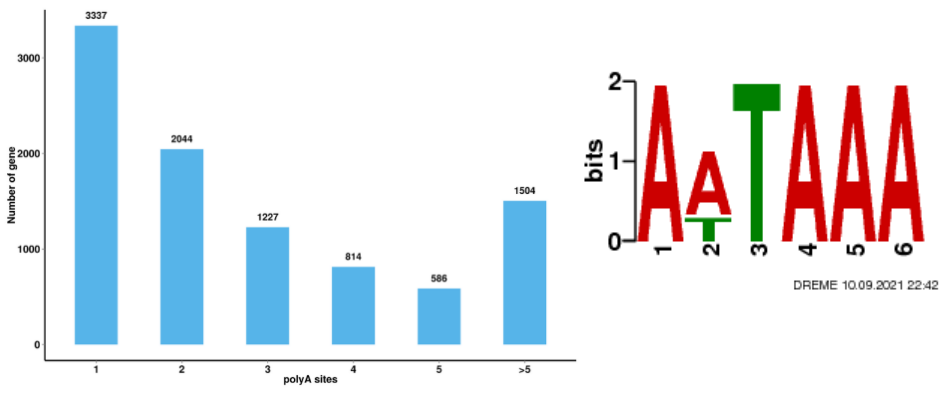
Alternatīva savienojuma analīze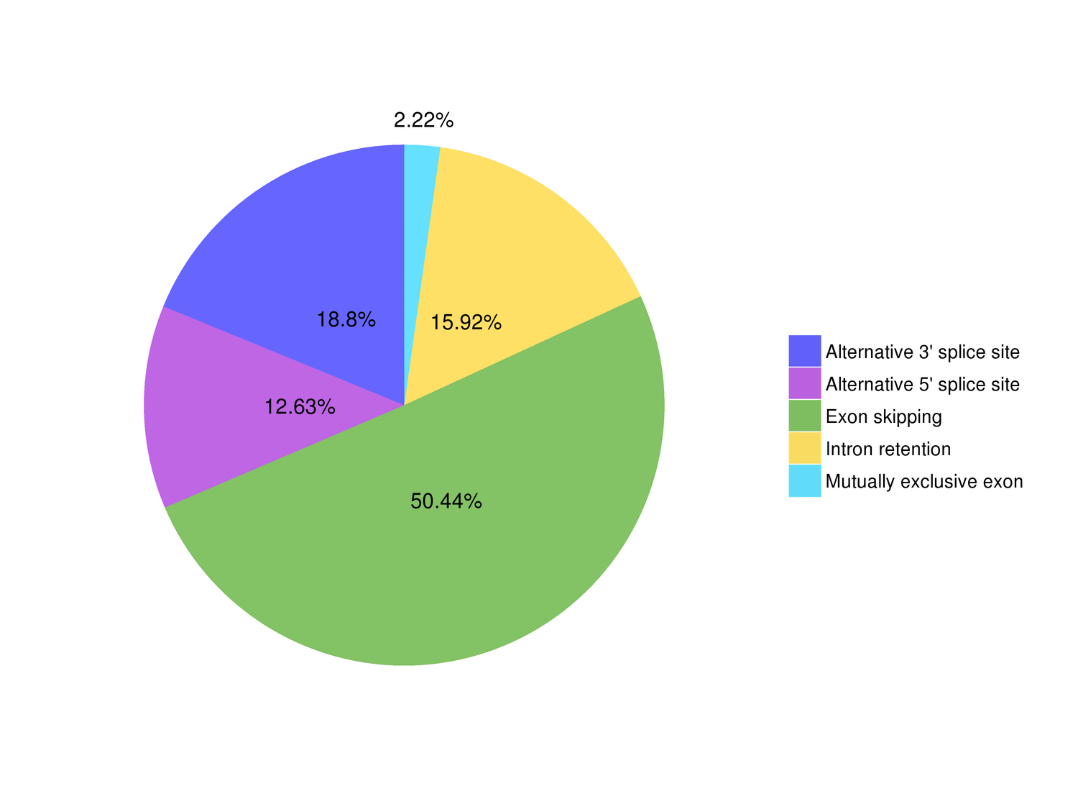 Alternatīvā poliadenilācijas analīze (APA)
Alternatīvā poliadenilācijas analīze (APA)
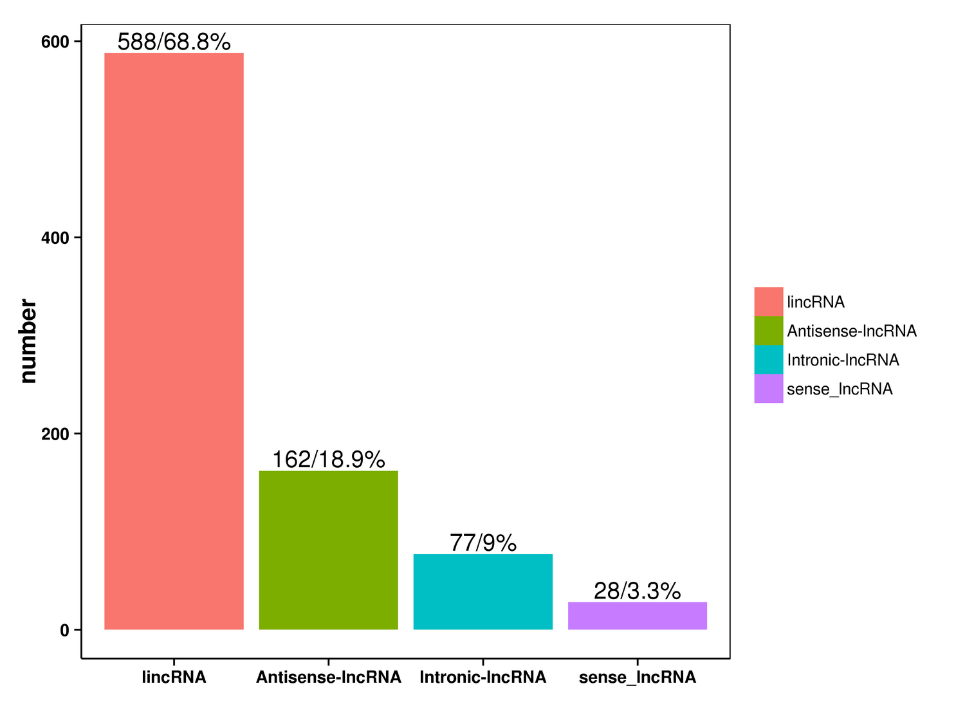
lncRNS prognozēšana
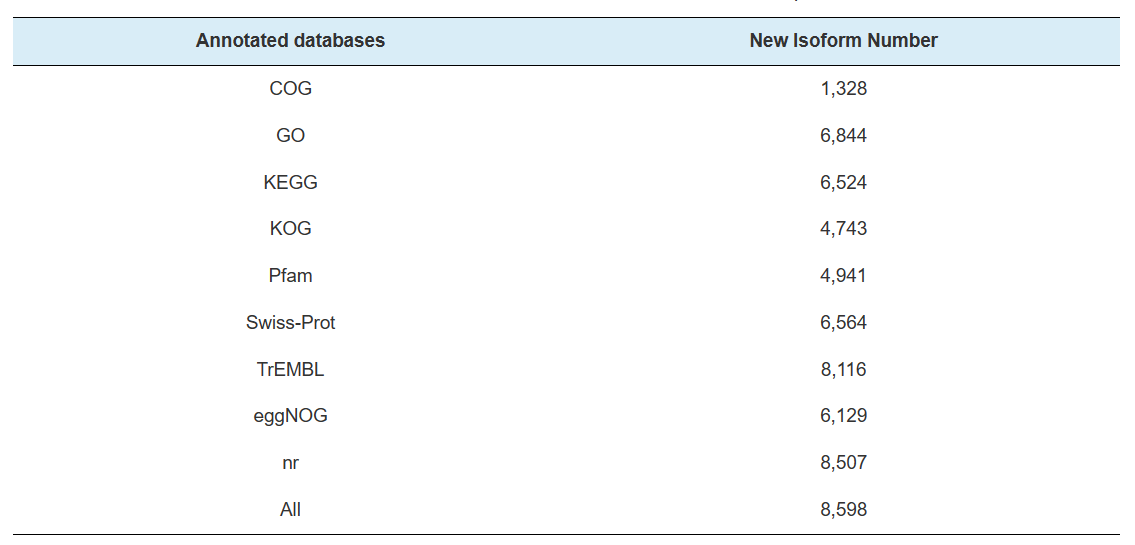
Jaunu gēnu anotācija
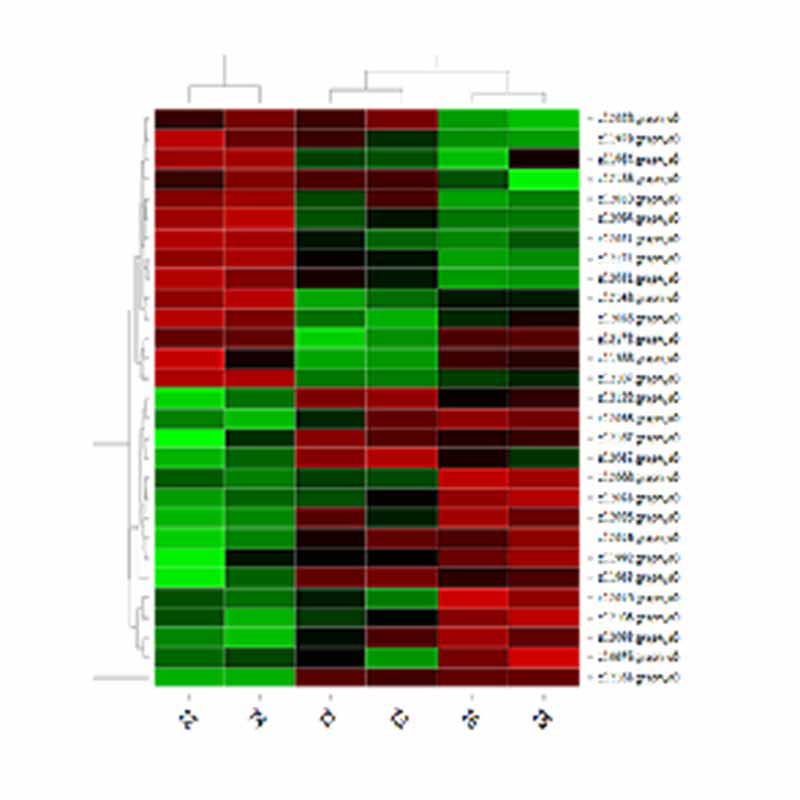
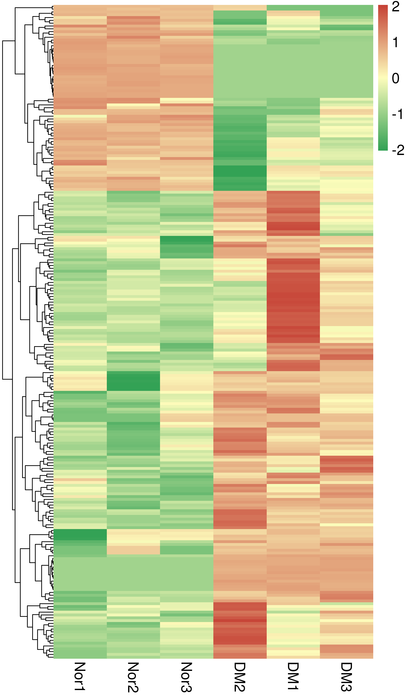
DET klasterizācija
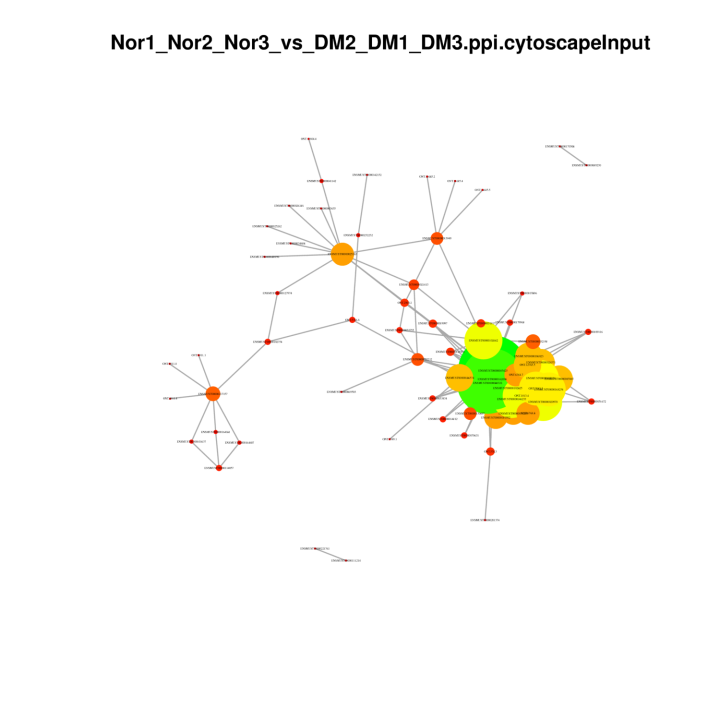
Olbaltumvielu un olbaltumvielu tīkli DEG
Izpētiet sasniegumus, ko veicina BMKGene Nanopore pilna garuma mRNS sekvencēšanas pakalpojumi, izmantojot apkopotu publikāciju kolekciju.
Gong, B. et al. (2023) “Sekrēcijas kināzes FAM20C kā onkogēna epiģenētiskā un transkripcijas aktivizēšana gliomas gadījumā”, Journal of Genetics and Genomics, 50 (6), 422.–433. lpp. doi: 10.1016/J.JGG.2023.01.008.
Viņš, Z. et al. (2023) “Pilna garuma limfocītu transkripta sekvencēšana, kas reaģē uz IFN-γ, atklāj Th1 novirzītu imūnreakciju plekstei (Paralichthys olivaceus)”, Fish & Shellfish Immunology, 134. lpp. 108636. doi: 10.1016/J.FSI.2023.108636.
Ma, Y. et al. (2023) “PacBio un ONT RNS sekvencēšanas metožu salīdzinošā analīze Nemopilema Nomurai indes identifikācijai”, Genomics, 115(6), lpp. 110709. doi: 10.1016/J.YGENO.2023.110709.
Yu, D. et al. (2023) “Nano-seq analīze atklāj dažādas funkcionālās tendences starp eksosomām un mikrovezikulām, kas iegūtas no hUMSC”, Stem Cell Research and Therapy, 14(1), 1.–13. lpp. doi: 10.1186/S13287-023-03491-5/TABLES/6.