Liela segreganta analīze
Pakalpojumu priekšrocības

Takagi et al., The Plant Journal, 2013
● Precīza lokalizācija: sajaukšana ar bugām ar 30+30 līdz 200+200 indivīdiem, lai samazinātu fona troksni; Neinonīms mutatantiem balstīts kandidātu reģiona prognoze.
● Visaptveroša analīze: padziļināta kandidāta gēnu funkcijas anotācija, ieskaitot NR, Swissprot, Go, Kegg, Cog, Kog utt.
● Ātrāks apgrozījuma laiks: ātra gēnu lokalizācija 45 darba dienās.
● Plaša pieredze: BMK ir veicinājis tūkstošiem pazīmju lokalizāciju, aptverot dažādas sugas, piemēram, kultūras, ūdens izstrādājumus, mežu, ziedus, augļus utt.
Pakalpojumu specifikācijas
Iedzīvotāji:
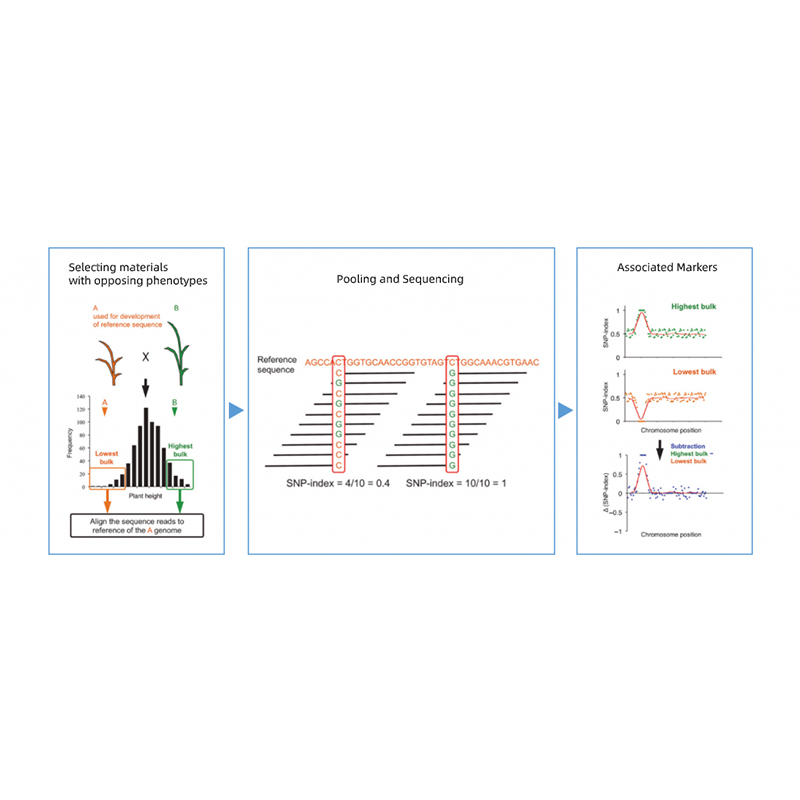
Vecāku pēcnācēju nodalīšana ar pretējiem fenotipiem.
Piemēram
Sajaukšanas baseins
Kvalitatīvām iezīmēm: no 30 līdz 50 indivīdiem (vismaz 20)/lielapjoma
Kvantitatīvajam tratim: 5% līdz 10% cilvēku ar vai nu ekstrēmiem fenotipiem visā populācijā (vismaz 30+30).
Ieteicamais secības dziļums
Vismaz 20x/vecāku un 1x/pēcnācēju indivīds (piemēram, par pēcnācēju sajaukšanas fondu, kurā ir 30+30 indivīds, sekvencēšanas dziļums būs 30x par lielapjomu)
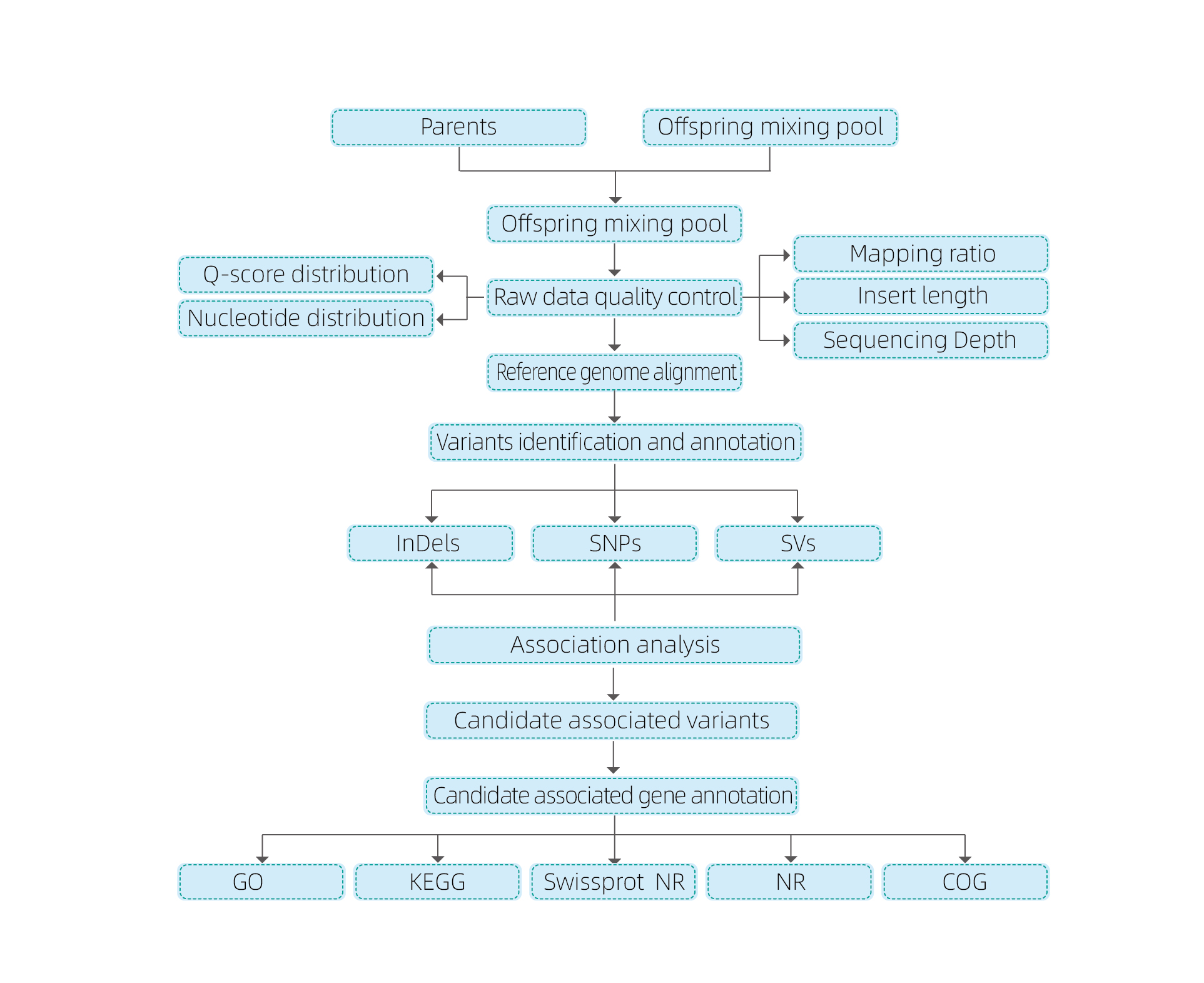
Bioinformātikas analīze
● vesela genoma atjaunošana
● Datu apstrāde
● SNP/indel zvana
● Kandidāta reģiona skrīnings
● Kandidāta gēnu funkcijas anotācija

Parauga prasības un piegāde
Parauga prasības:
Nukleotīdi:
| gDNS paraugs | Audu paraugs |
| Koncentrācija: ≥30 ng/μl | Augi: 1-2 g |
| Daudzums: ≥2 μg (Volumn ≥15 μl) | Dzīvnieki: 0,5-1 g |
| Tīrība: OD260/280 = 1,6–2,5 | Pilnas asinis: 1,5 ml |
Servisa darba plūsma

Eksperimenta dizains

Paraugu piegāde

RNS ekstrakcija

Bibliotēkas celtniecība

Sekvencēšana

Datu analīze

Pēcpārdošanas pakalpojumi
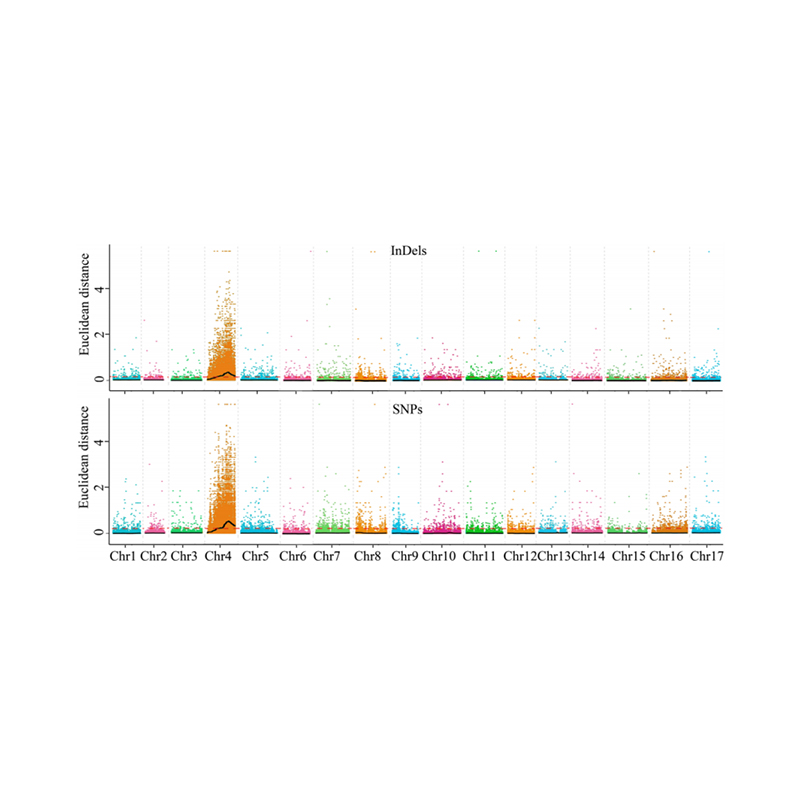
1. Asociācijas analīzes bāze uz Eiklīda attālumu (ed), lai identificētu kandidātu reģionu. Šajā attēlā
X ass: hromosomu numurs; Katrs punkts apzīmē SNP ed vērtību. Melnā līnija atbilst uzstādītajai ED vērtībai. Augstāka ED vērtība norāda uz nozīmīgāku saistību starp vietni un fenotipu. Red Dash Line apzīmē nozīmīgas asociācijas slieksni.

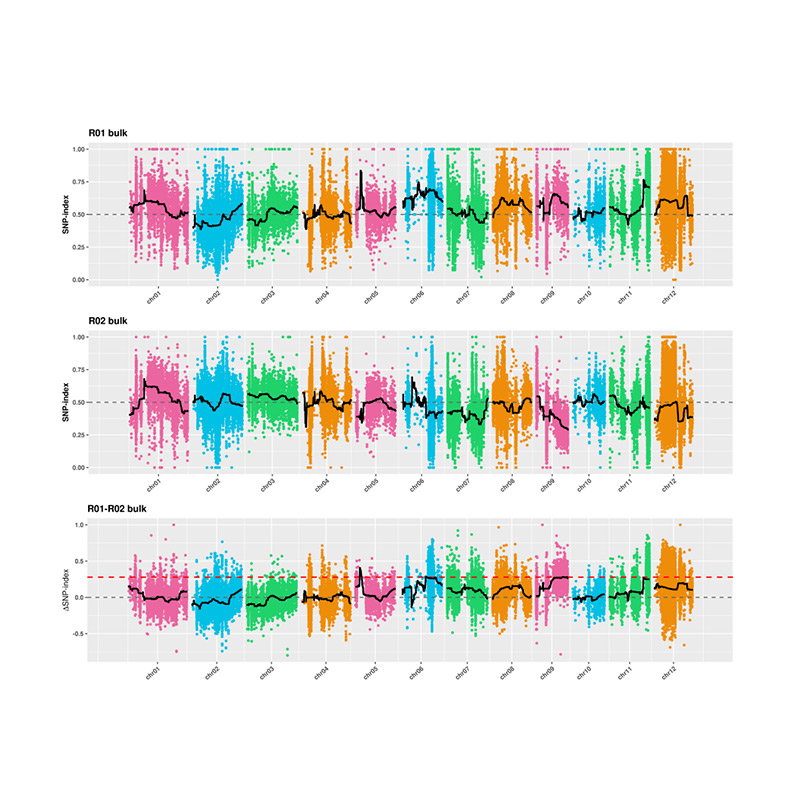
2.Asociācijas analīze, kuras pamatā nav SNP indekss
X ass: hromosomu numurs; Katrs punkts apzīmē SNP-indeksa vērtību. Melnā līnija apzīmē uzstādīto SNP-indeksa vērtību. Jo lielāka ir vērtība, jo nozīmīgāka ir asociācija.

BMK korpuss
Galvenā efekta kvantitatīvās pazīmes lokuss fnl7.1 kodē vēlu embrioģenēzi bagātīgs proteīns, kas saistīts ar augļu kakla garumu gurķī
Publicēts: Augu biotehnoloģijas žurnāls, 2020. gads
Secības stratēģija:
Vecāki (Jin5-508, YN): vesela genoma atkārtota veiktspēja 34 × un 20 ×.
DNS baseini (50 garu kaklu un 50 īsu kaklu): 61 × un 52 × atkārtota vienādošana
Galvenie rezultāti
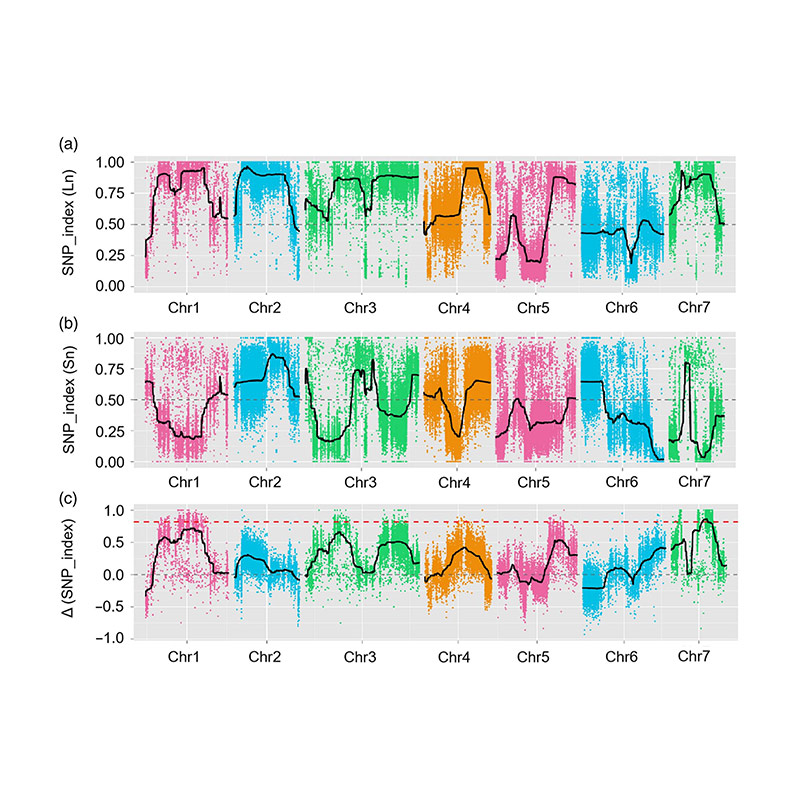
Šajā pētījumā tika izveidota populācija (F2 un F2: 3), šķērsojot garu kakla gurķu līniju Jin5-508 un īsu kaklu YN. Divus DNS baseinus uzbūvēja 50 ekstrēmi garu kaklu un 50 ekstrēmi īsu kaklu indivīdi. Lielāko efektu QTL tika identificēts ar CHR07 ar BSA analīzi un tradicionālo QTL kartēšanu. Kandidāta reģions tika vēl vairāk sašaurināts ar smalku kartēšanu, gēnu ekspresijas kvantitatīvo noteikšanu un transgēniem eksperimentiem, kas atklāja galveno gēnu, kontrolējot kakla garumu, csfnl7.1. Turklāt tika konstatēts, ka polimorfisms CSFNL7.1 promotora reģionā ir saistīts ar atbilstošo ekspresiju. Turpmākā filoģenētiskā analīze liecināja, ka FNL7.1 lokuss, visticamāk, ir cēlies no Indijas.
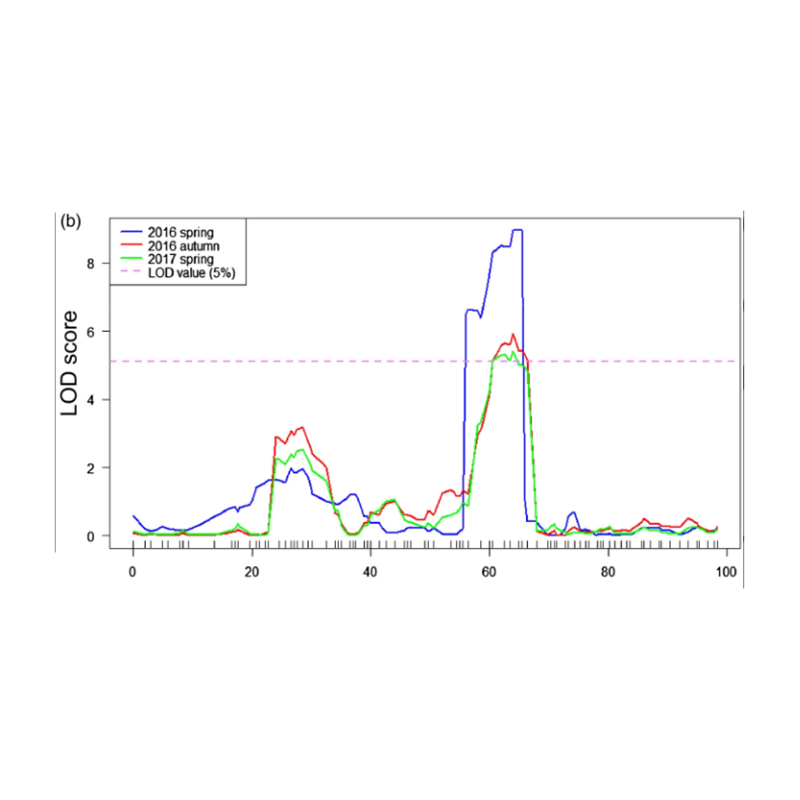
 QTL kartēšana BSA analīzē, lai identificētu kandidātu reģionu, kas saistīts ar gurķu kakla garumu |  LOD profili no gurķu kakla garuma QTL, kas identificēts uz CHR07 |
Xu, X., et al. "Galvenā efekta kvantitatīvā iezīme Locus fnl7.1 kodē vēlu embrioģenēzi bagātīgu olbaltumvielu, kas saistīta ar augļu kakla garumu gurķī." Augu biotehnoloģijas žurnāls 18.7 (2020).