Visas transkripto sekos nustatymas - „Illumina“
Savybės
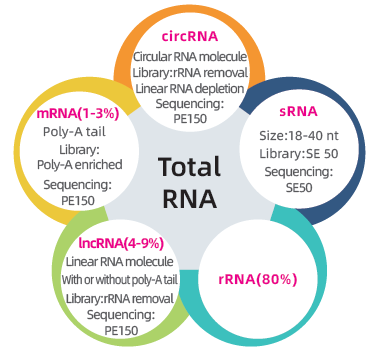
● Dviguba biblioteka, skirta sekti visą transkriptą: rRNR išeikvojimas, po kurio seka PE150 bibliotekos paruošimas ir dydžio pasirinkimas, po kurio seka SE50 bibliotekos paruošimas
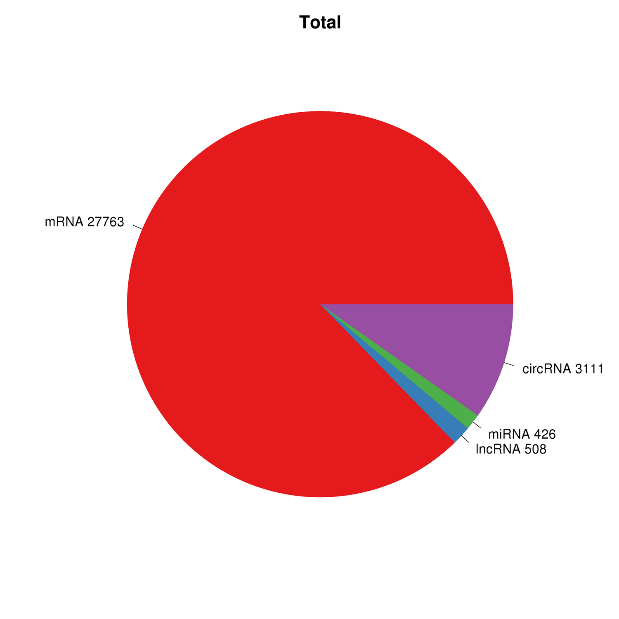
● Visiška mRNR, lncRNR, circRNR ir miRNR bioinformatikos analizė atskirose bioinformatikos ataskaitose pateiktose bioinformatikos ataskaitose
● Bendra visos RNR ekspresijos analizė kombinuotoje ataskaitoje, įskaitant CERNA tinklų analizę.
Aptarnavimo pranašumai
●Išsami reguliavimo tinklų analizė: CERNA tinklo analizę įgalina sąnario mRNR, lncRNR, circRNR ir miRNR sekos nustatymas, o išsami bioinformatinė darbo eiga.
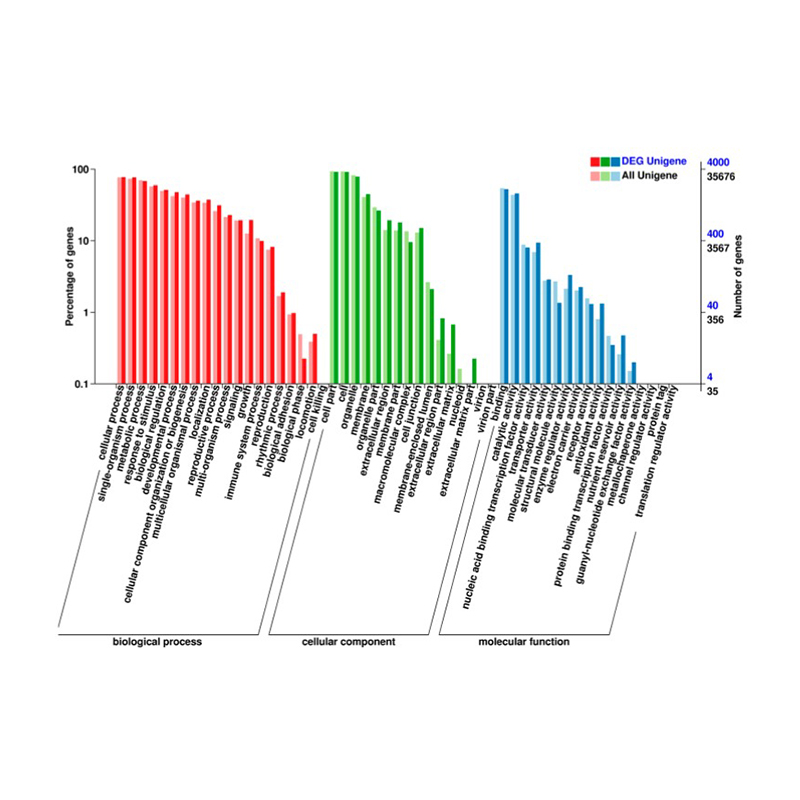
●Išsami anotacija: Mes naudojame kelias duomenų bazes, kad funkciškai komentuojame skirtingai išreikštus genus (DEG) ir atliekame atitinkamą praturtinimo analizę, suteikdami įžvalgų apie ląstelių ir molekulinius procesus, kuriais grindžiamas transkripto atsakas.
●Didelė patirtis: Turėdama sėkmingą daugiau nei 2100 transkriptų projektų uždarymą įvairiose tyrimų srityje, mūsų komanda suteikia daug patirties kiekvienam projektui.
●Griežta kokybės kontrolė: Mes įgyvendiname pagrindinius kontrolės taškus visuose etapuose, pradedant nuo mėginių ir bibliotekos paruošimo iki sekos nustatymo ir bioinformatikos. Šis kruopštus stebėjimas užtikrina nuolat aukštos kokybės rezultatus.
●Palaikymas po pardavimo: Mūsų įsipareigojimas apima ne tik projekto pabaigą su 3 mėnesių aptarnavimo laikotarpiu po pardavimo. Per tą laiką mes siūlome projekto tolesnius veiksmus, pagalbą triktis ir klausimų ir atsakymų sesijas
Imties reikalavimai ir pristatymas
| Biblioteka | Sekos strategija | Rekomenduojami duomenys | Kokybės kontrolė |
| RRNR išeikvojo | „Illumina PE150“ | 16 GB | Q30≥85% |
| Pasirinktas dydis | „Illumina SE50“ | 10-20m skaito |
Imties reikalavimai:
Nukleotidai:
| Konc. (Ng/μl) | Kiekis (μg) | Grynumas | Vientisumas |
| ≥ 80 | ≥ 1,6 | OD260/280 = 1,7–2,5 OD260/230 = 0,5–2,5 Ribotas baltymų ar DNR užterštumas, parodytas ant gelio. | RIN≥6,0 5,0≥28S/18S ≥1,0; Ribotas arba jo nėra pradiniame aukštyje |
Rekomenduojamas pavyzdžių pristatymas
Konteineris: 2 ml centrifugos vamzdis (alavo folija nerekomenduojama)
Imties ženklinimas: grupė+pakartoja EG A1, A2, A3; B1, B2, B3.
Siunta:
1. Sausas ledas: mėginius reikia supakuoti į maišus ir palaidoti sauso ledo.
2. RNASTATS vamzdeliai: RNR mėginius galima džiovinti RNR stabilizavimo vamzdyje (pvz., RNASTABLE®) ir išsiųsti kambario temperatūroje.
Aptarnavimo darbo srautas

Eksperimento dizainas

Mėginio pristatymas

RNR ekstrahavimas

Bibliotekos statyba

Sekos nustatymas

Duomenų analizė

Paslaugos po pardavimo
Bioinformatika
RNR išraiškos apžvalga
Diferencijuotai išreikšti genai
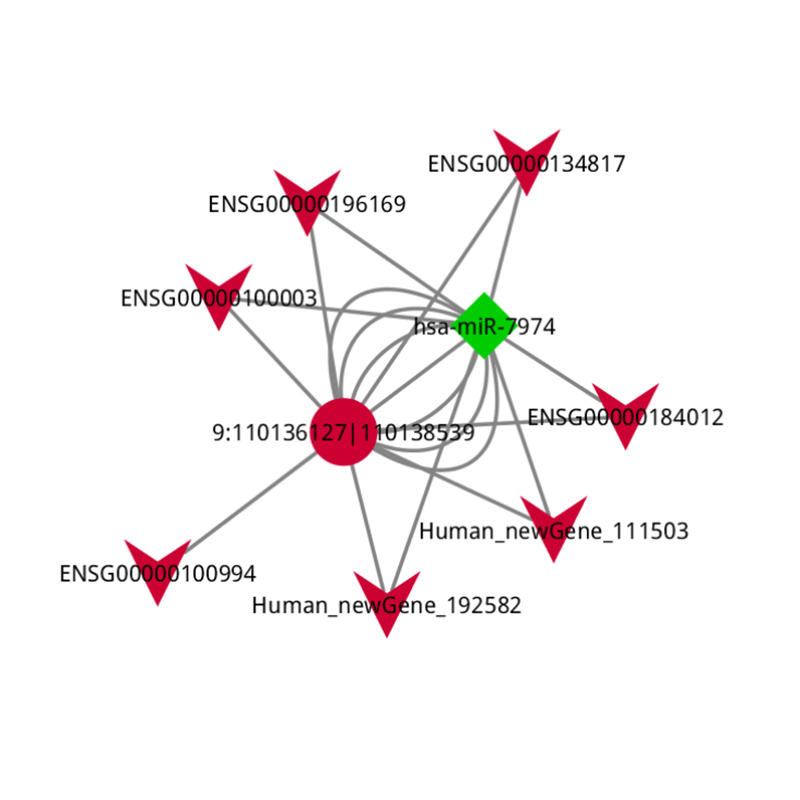
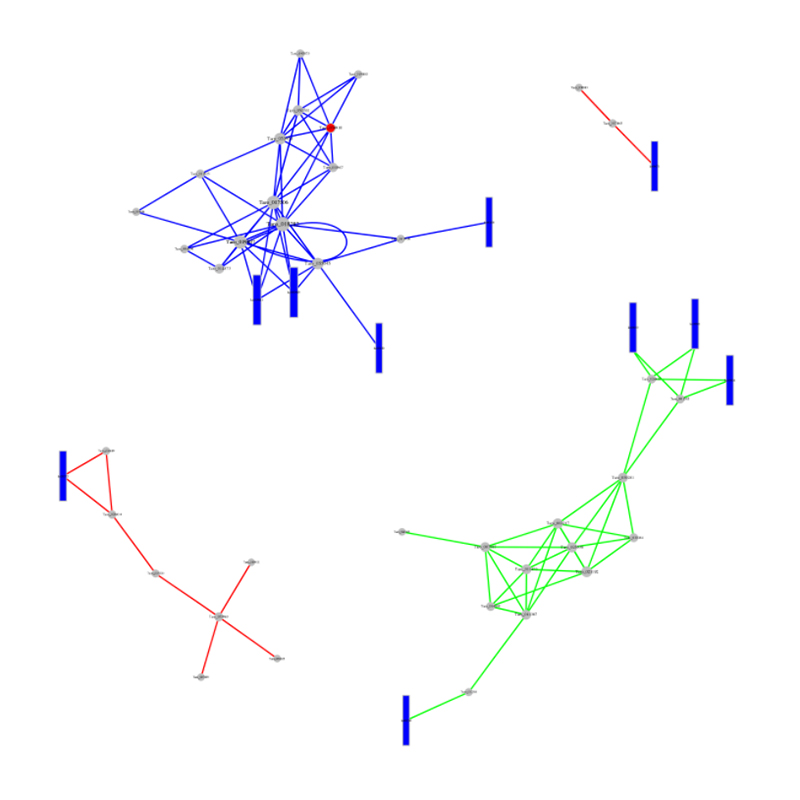
CERNA analizė
Ištirkite tyrimų pažangą, palengvintą visos „Bmkgene“ transkriptų sekos nustatymo paslaugų per kuruojamą leidinių kolekciją.
Dai, Y. et al. (2022) „Išsamios mRNR, LNCRNR ir miRNR raiškos profiliai, sergant Kashin-Beck liga, identifikuojama RNR sekos nustatymu“, Molecular Omics, 18 (2), p. 154–166. doi: 10.1039/d1mo00370d.
Liu, N. Nan ir kt. (2022 m.) „Viso ilgio transkriptų, susijusių su API CERANA atsparumu šaltai, Changbai kalne analizė žiemojančiu laikotarpiu.“, Gene, 830, p. 146503–146503. doi: 10.1016/j.gene.2022.146503.
Wang, XJ ir kt. (2022 m.) „Konkuruojančių endogeninių RNR reguliavimo tinklų, esančių mažų ląstelių plaučių vėžyje, daugialypės integracijai pagrįstas prioritetų nustatymas: molekulinės charakteristikos ir kandidatai į narkotikus“, „Frontiers in onkologijoje“, 12, p. 904865. Doi: 10.3389/fonc.2022.904865/bibtex.
Xu, P. et al. (2022) „Integruota lncRNR/circRNR-mirNR-mRNR ekspresijos profilių analizė atskleidžia naujas įžvalgas apie galimus mechanizmus, reaguojant į šaknies mazgų nematodus žemės riešute“, BMC genomika, 23 (1), p. 1–12. doi: 10.1186/s12864-022-08470-3/paveikslai/7.
Yan, Z. et al. (2022) „Viso transkripto RNR sekos nustatymas pabrėžia molekulinius mechanizmus, susijusius su brokolių povilio kokybės palaikymu raudoname LED švitinime“, „Postharvest“ biologija ir technologija, 188, p. 111878. Doi: 10.1016/j.postharvbio.2022.111878.