Maža RNR seka-iliusnacija
Savybės
● Bibliotekos paruošimas apima dydžio pasirinkimo žingsnį
● Bioinformatinė analizė, nukreipta į miRNR prognozę ir jų taikinius
Aptarnavimo pranašumai
●Išsami bioinformatikos analizė:Įgalinant tiek žinomų, tiek naujų miRNR identifikavimą, miRNR taikinių identifikavimą ir atitinkamą funkcinę anotaciją ir praturtėjimą naudojant kelias duomenų bazes (KEGG, GO)
●Griežta kokybės kontrolė: Mes įgyvendiname pagrindinius kontrolės taškus visuose etapuose, pradedant nuo mėginių ir bibliotekos paruošimo iki sekos nustatymo ir bioinformatikos. Šis kruopštus stebėjimas užtikrina nuolat aukštos kokybės rezultatus.
●Palaikymas po pardavimo: Mūsų įsipareigojimas apima ne tik projekto pabaigą su 3 mėnesių aptarnavimo laikotarpiu po pardavimo. Per tą laiką mes siūlome projekto tolesnius veiksmus, pagalbą trikčių šalinimui ir klausimų ir atsakymų sesijoms, kad išspręstume bet kokius su rezultatus susijusius klausimus.
●Didelė patirtis: Turėdami sėkmingai užbaigti kelis SRNR projektus, apimančius daugiau nei 300 rūšių įvairiose tyrimų srityse, mūsų komanda suteikia daug patirties kiekvienam projektui.
Imties reikalavimai ir pristatymas
| Biblioteka | Platforma | Rekomenduojami duomenys | Duomenys QC |
| Pasirinktas dydis | „Illumina SE50“ | 10m-20m skaito | Q30≥85% |
Imties reikalavimai:
Nukleotidai:
| Konc. (Ng/μl) | Kiekis (μg) | Grynumas | Vientisumas |
| ≥ 80 | ≥ 0,8 | OD260/280 = 1,7–2,5 OD260/230 = 0,5–2,5 Ribotas baltymų ar DNR užterštumas, parodytas ant gelio. | RIN≥6,0; 5,0≥28S/18S ≥1,0; Ribotas arba jo nėra pradiniame aukštyje |
● Augalai:
Šaknis, kamienas ar žiedlapis: 450 mg
Lapai ar sėkla: 300 mg
Vaisiai: 1,2 g
● Gyvūnas:
Širdis ar žarnynas: 450 mg
Vėžė arba smegenys: 240 mg
Raumenys: 600 mg
Kaulai, plaukai ar oda: 1,5 g
● Narofodai:
Vabzdžiai: 9g
Crustacea: 450 mg
● Visas kraujas: 2 vamzdžiai
● Ląstelės: 106 ląstelės
● Serumas ir plazma:6 ml
Rekomenduojamas pavyzdžių pristatymas
Konteineris: 2 ml centrifugos vamzdis (alavo folija nerekomenduojama)
Imties ženklinimas: grupė+pakartoja EG A1, A2, A3; B1, B2, B3.
Siunta:
1. Sausas ledas: mėginius reikia supakuoti į maišus ir palaidoti sauso ledo.
2. RNASTABLE vamzdeliai: RNR mėginius galima džiovinti RNR stabilizavimo vamzdyje (pvz., RNASTABLE®) ir išsiunčiami kambario temperatūroje.
Aptarnavimo darbo srautas

Eksperimento dizainas

Mėginio pristatymas

RNR ekstrahavimas

Bibliotekos statyba

Sekos nustatymas

Duomenų analizė

Paslaugos po pardavimo
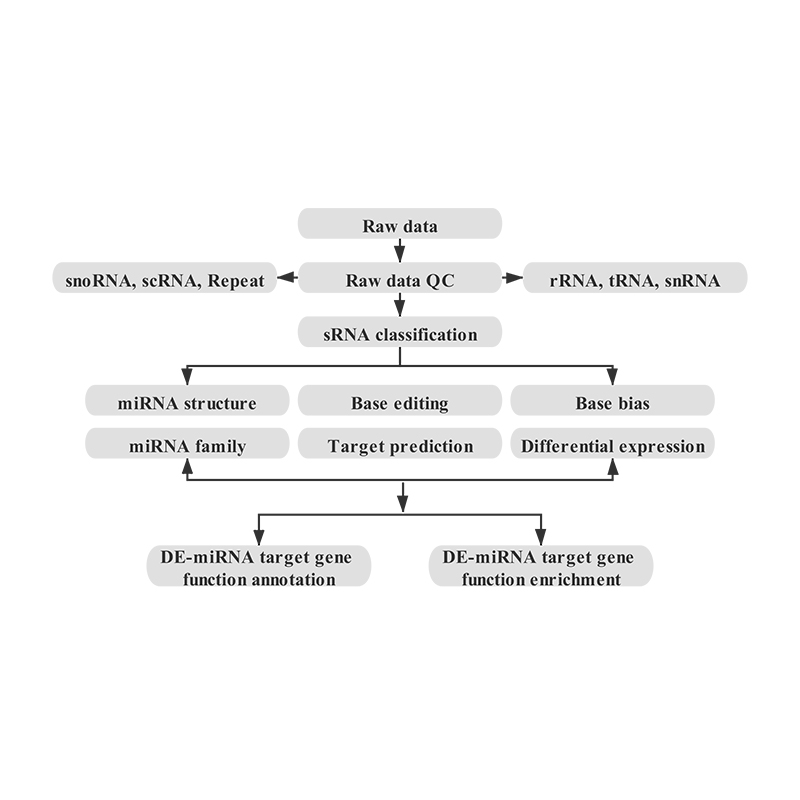
Bioinformatika
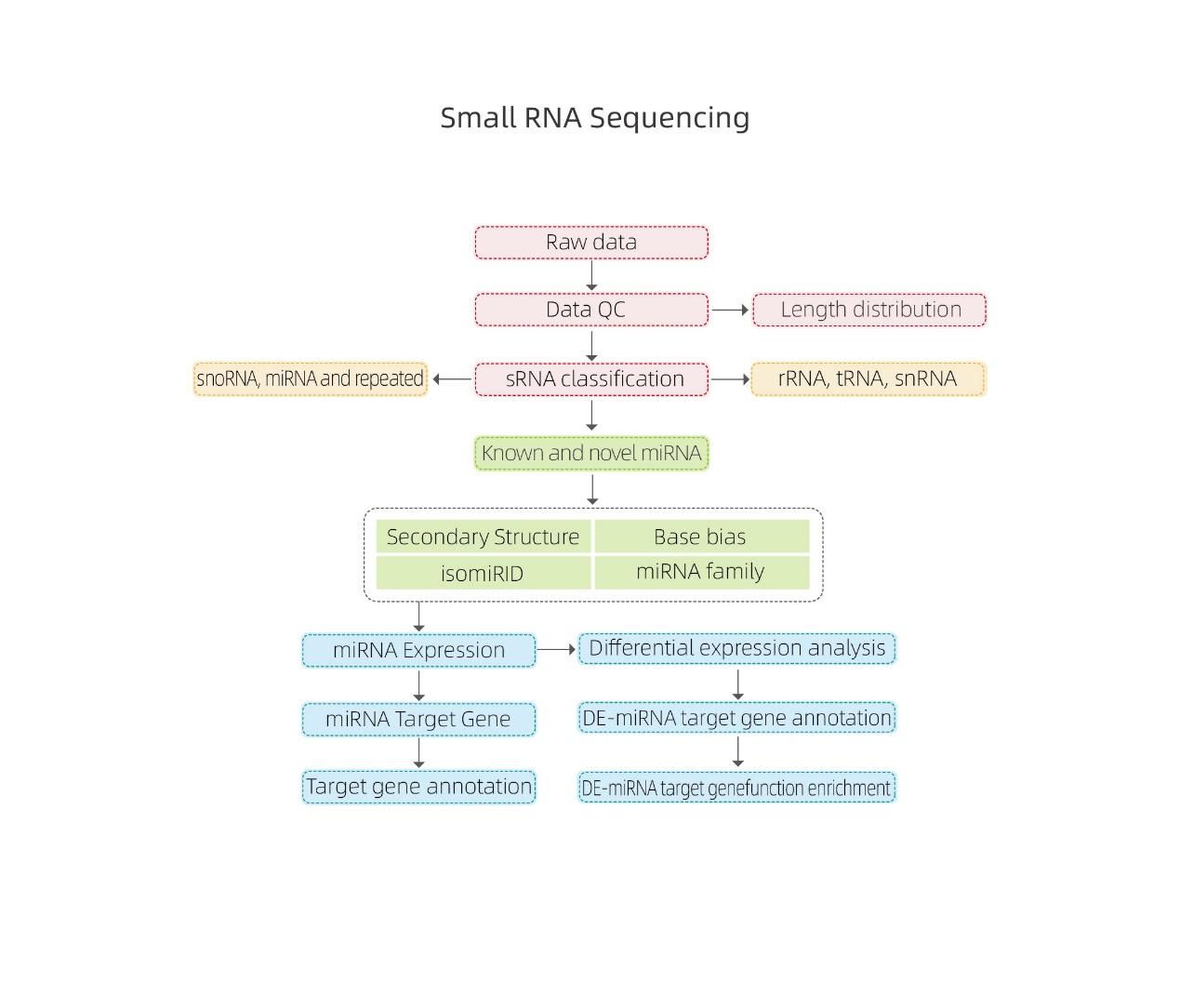 ● Neapdoroto duomenų kokybės kontrolė
● Neapdoroto duomenų kokybės kontrolė
● SRNR klasifikacija
● Derinimas su etaloniniu genomu
● Žinomos ir naujos miRNR identifikavimas
● Diferencialinė miRNR išraiškos analizė
● Funkcinis miRNR taikinių anotacija
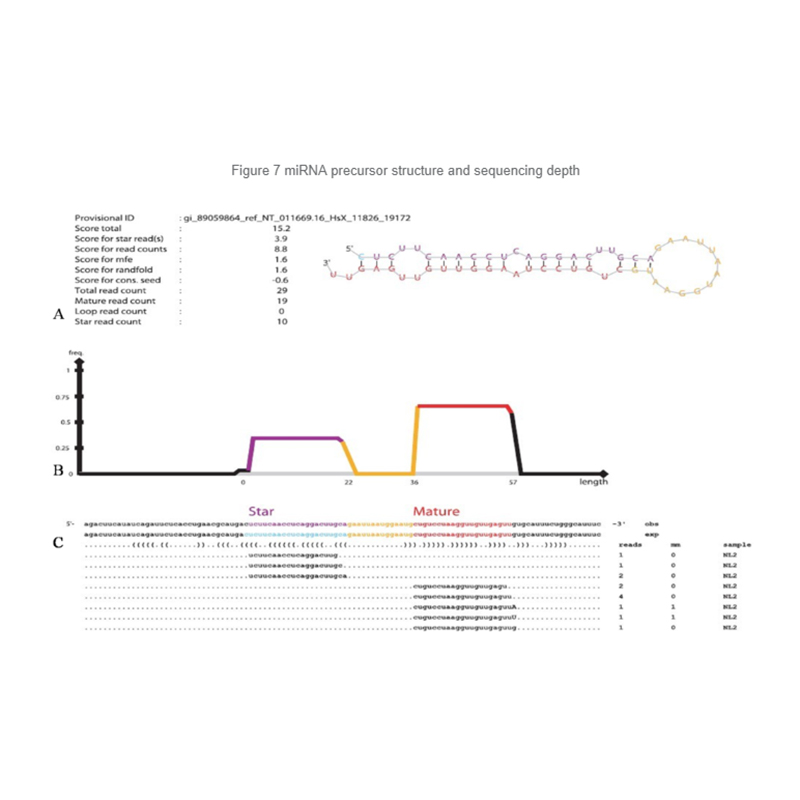
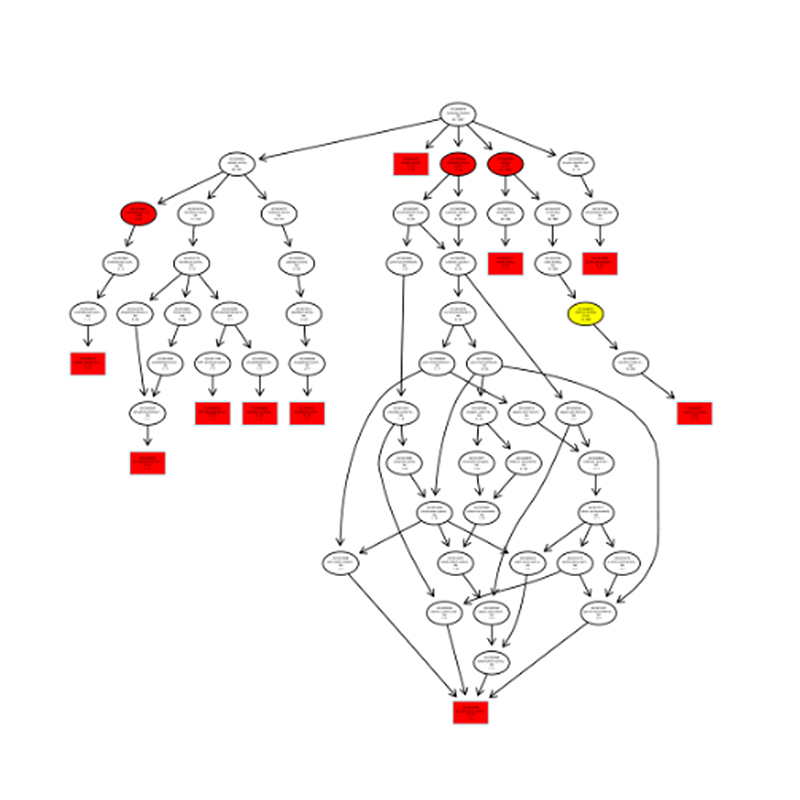
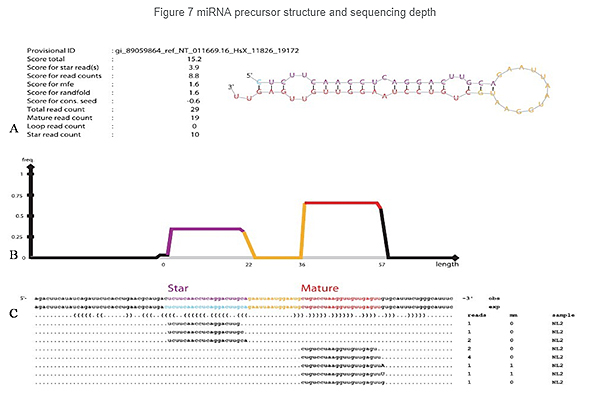
MiRNR identifikavimas: struktūra ir gylis
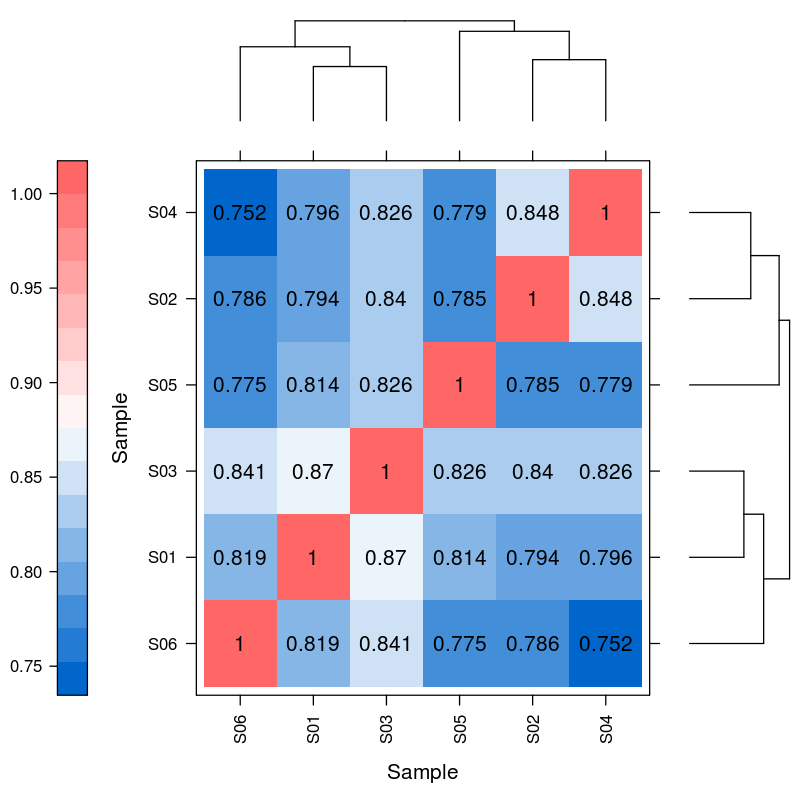
Diferencinė miRNR išraiška - hiearchical Clustering
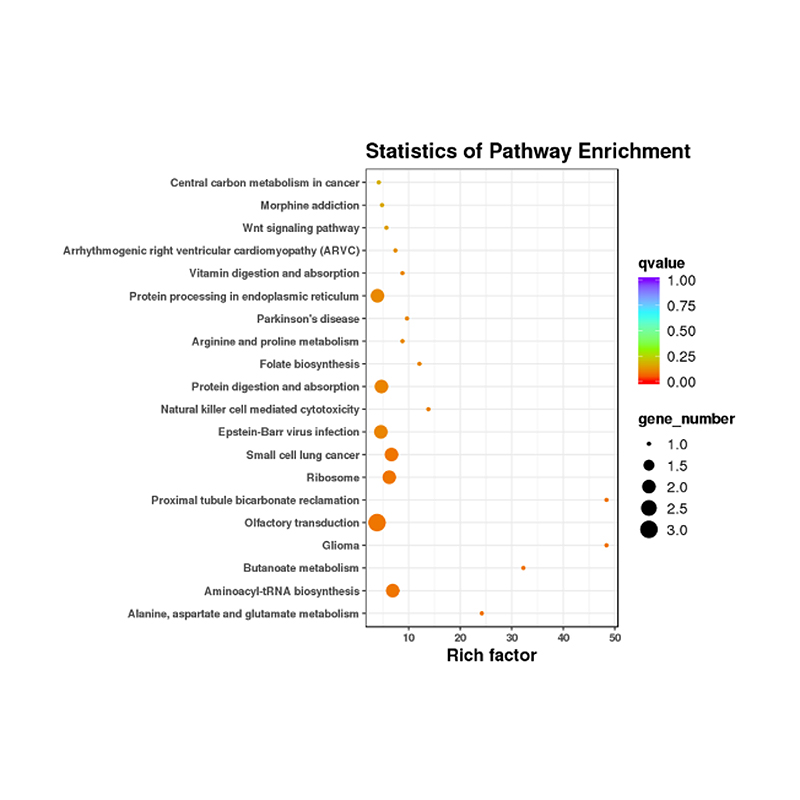
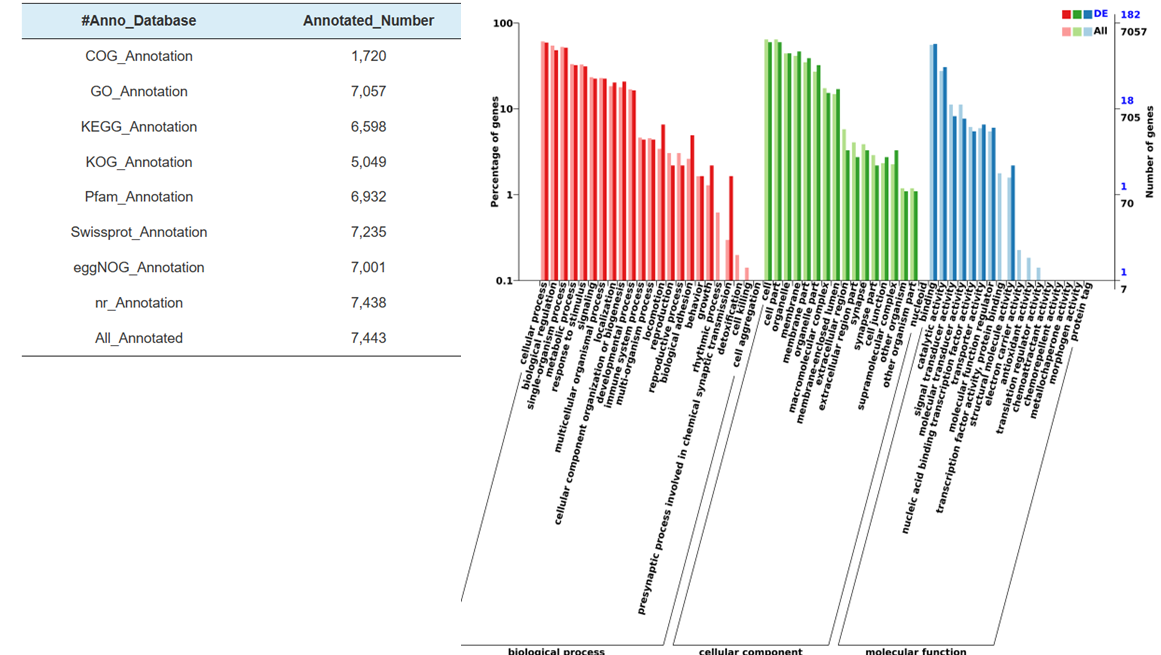
Funkcinis diferencijuotai išreikštų miRNR tikslo anotacija
Ištirkite tyrimų pažangą, kurią palengvino Bmkgene SRNR sekos nustatymo paslaugos per kuruojamą leidinių kolekciją.
Chen, H. ir kt. (2023) „Virusinės infekcijos slopina saponino biosintezę ir fotosintezę Panax Notoginseng“, Augalų fiziologija ir biochemija, 203, p. 108038. Doi: 10.1016/j.plaphy.2023.108038.
Li, H. ir kt. (2023 m.) „Augalas Fyve domenas turintys baltymai be baltymų yra susiję su mikroprocesoriniais komponentais, kad būtų slopinamos miRNR biogenezę“, EMBO ataskaitos, 24 (1). doi: 10.15252/Embr.202255037/Suppl_file/Empl202255037-Sup-0004-Sdatafig4.tif.
Yu, J. ir kt. (2023 m.) „MikroRNR AME-BANTAM-3P kontroliuoja lervų vyzdžių vystymąsi, nukreipdama į daugybinius epidermio augimo faktorių, panašių į 8 geną (mEGF8), medaus bičiuje, APIS mellifera“, Tarptautinis žurnalas „Molecular Sciences“, 24 (6), P), P 24 (6), P), P 24 (6), P) . 5726. Doi: 10.3390/ijms24065726/s1.
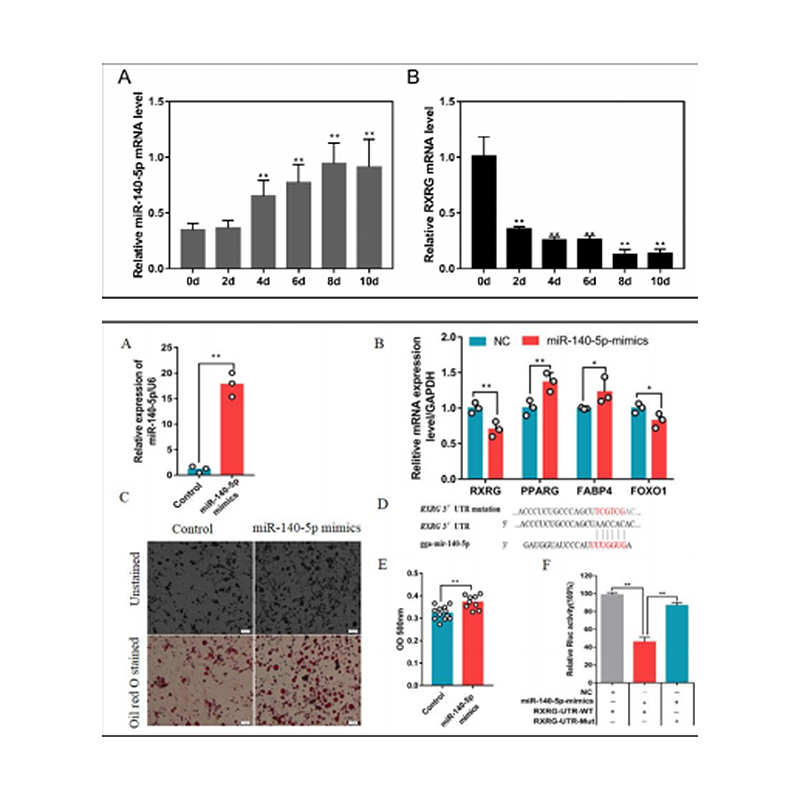
Zhang, M. et al. (2018) „Integruota miRNR ir genų, susijusių su mėsos kokybe, analizė atskleidžia, kad GGA-MIR-140-5P daro įtaką vištų raumenų nusėdimui į raumenis raumenis“, ląstelių fiziologija ir biochemija, 46 (6), p. 2421–2433. doi: 10.1159/000489649.