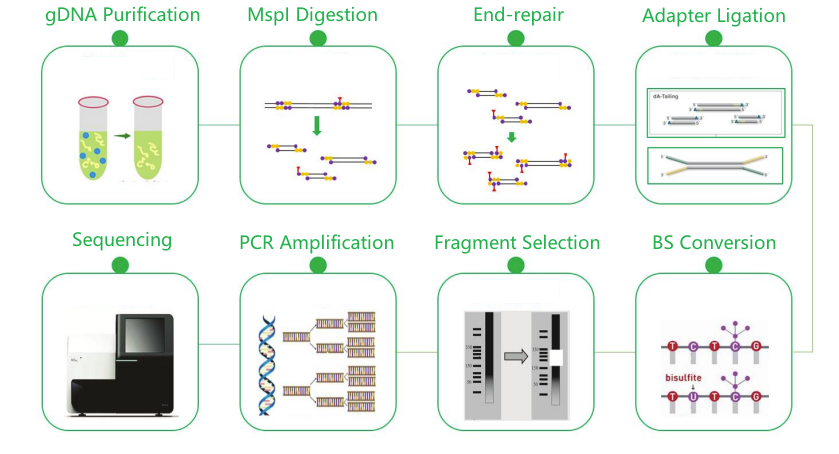
Sumažinto bisulfito sekos nustatymas (RRBS)
Paslaugos ypatybės
● Reikalingas etaloninis genomas.
● Bisulfito konversijos efektyvumui stebėti naudojama lambda DNR.
● Taip pat stebimas MspI virškinimo efektyvumas.
● Augalų mėginių virškinimas dvigubu fermentu.
● Sekos nustatymas naudojant Illumina NovaSeq.
Paslaugos privalumai
●Ekonomiška ir efektyvi WGBS alternatyva: leidžia atlikti analizę mažesnėmis sąnaudomis ir mažesniais mėginių reikalavimais.
●Pilna platforma:teikti vieno langelio puikias paslaugas nuo mėginių apdorojimo, bibliotekos konstravimo ir sekos nustatymo iki bioinformatinės analizės.
●Plati ekspertizė: sėkmingai užbaigus įvairių rūšių RRBS sekos sudarymo projektus, BMKGENE suteikia daugiau nei dešimtmečio patirtį, aukštos kvalifikacijos analizės komandą, išsamų turinį ir puikų palaikymą po pardavimo.
Paslaugos specifikacijos
| biblioteka | Sekos sudarymo strategija | Rekomenduojama duomenų išvestis | Kokybės kontrolė |
| MspI suardyta ir bisulfitu apdorota biblioteka | Illumina PE150 | 8 Gb | Q30 ≥ 85 % Bisulfito konversija > 99 % MspI pjovimo efektyvumas > 95 % |
Pavyzdiniai reikalavimai
| Koncentracija (ng/µL) | Bendras kiekis (µg) |
| |
| Genominė DNR | ≥ 30 | ≥ 1 | Ribotas skilimas arba užteršimas |
Aptarnavimo darbų eiga

Mėginio pristatymas

Bibliotekos statyba

Sekos nustatymas

Duomenų analizė
Paslaugos po pardavimo
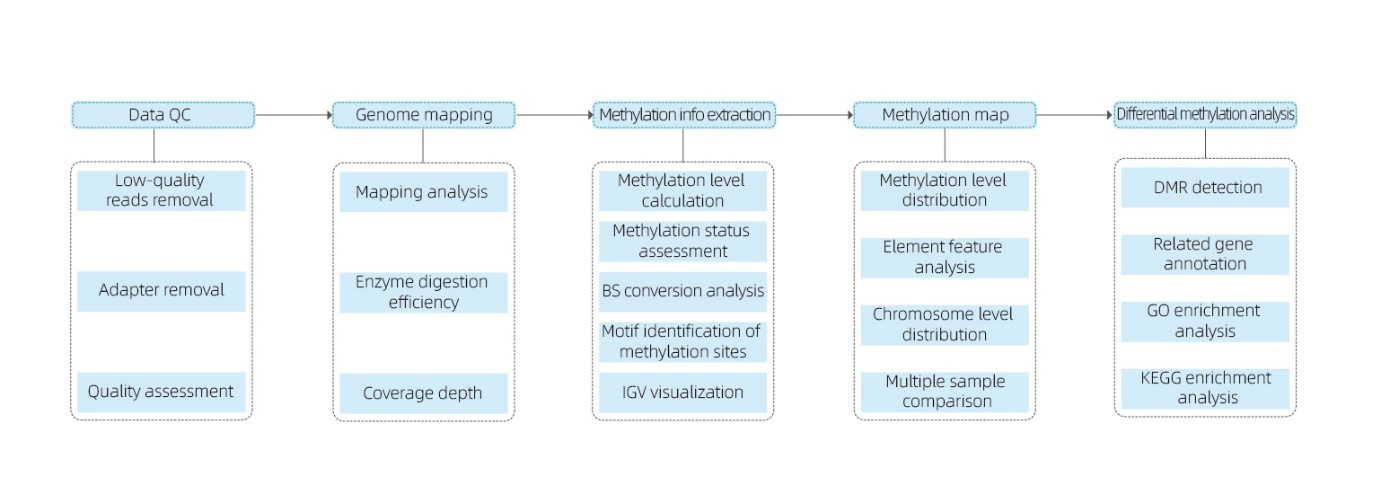
Apima šią analizę:
● Neapdorotos sekos kokybės kontrolė;
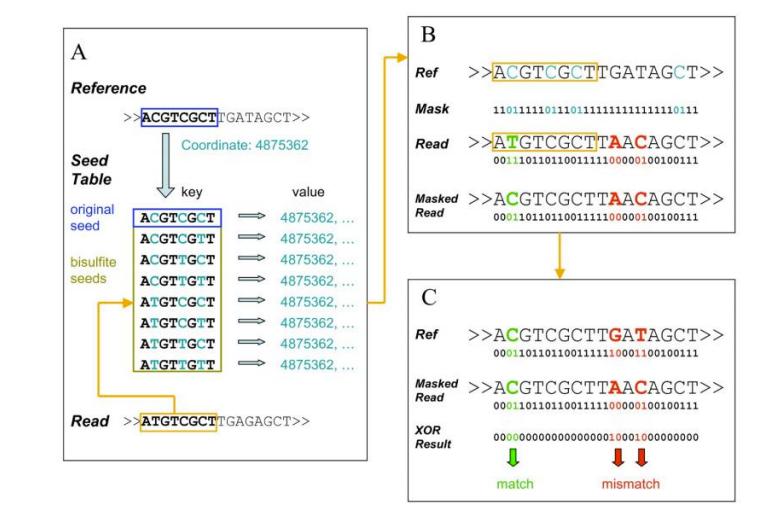
● Susiejimas su etaloniniu genomu;
● 5mC metilintų bazių aptikimas ir motyvų identifikavimas;
● Metilinimo pasiskirstymo ir mėginių palyginimo analizė;
● Diferencialiai metiluotų regionų (DMR) analizė;
● Su DMR susijusių genų funkcinė anotacija.
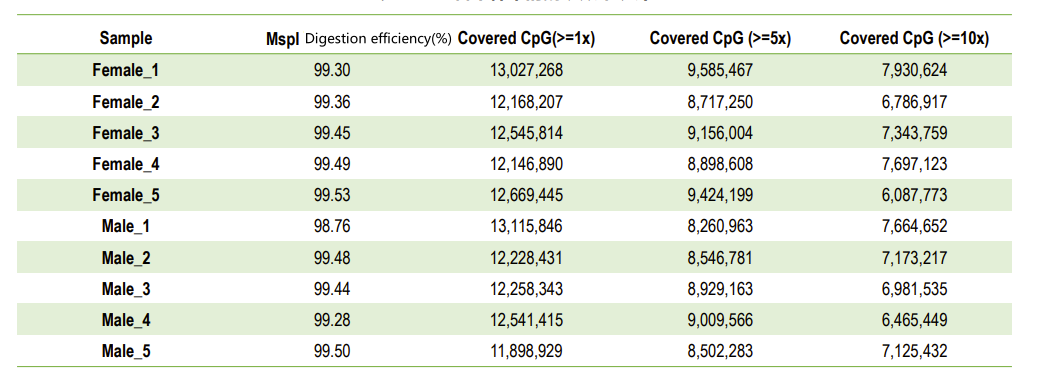
Kokybės kontrolė: virškinimo efektyvumas (genomo kartografijoje)
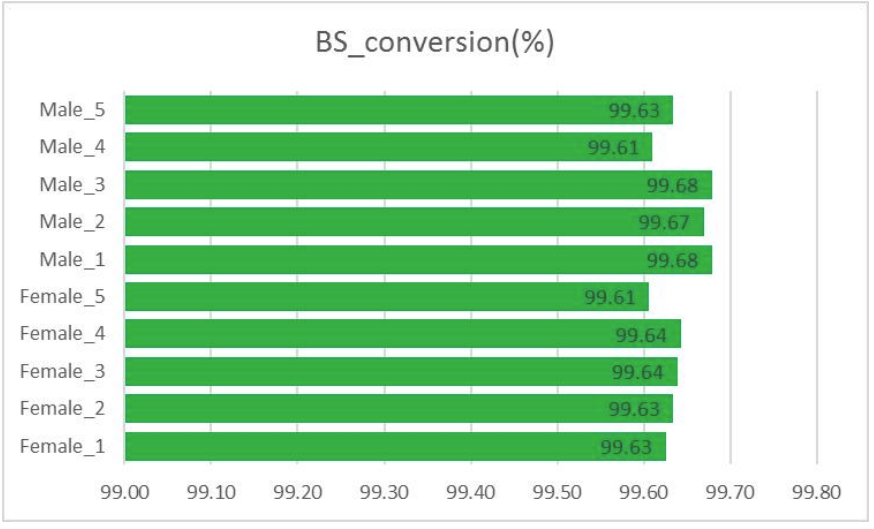
Kokybės kontrolė: bisulfito konversija (išgaunant informaciją apie metilinimą)
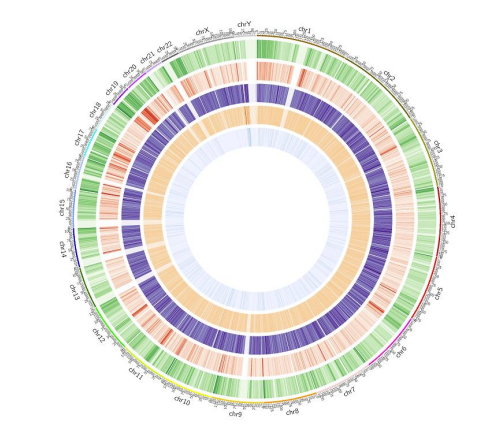
Metilinimo žemėlapis: 5mC metilinimo genomo pasiskirstymas
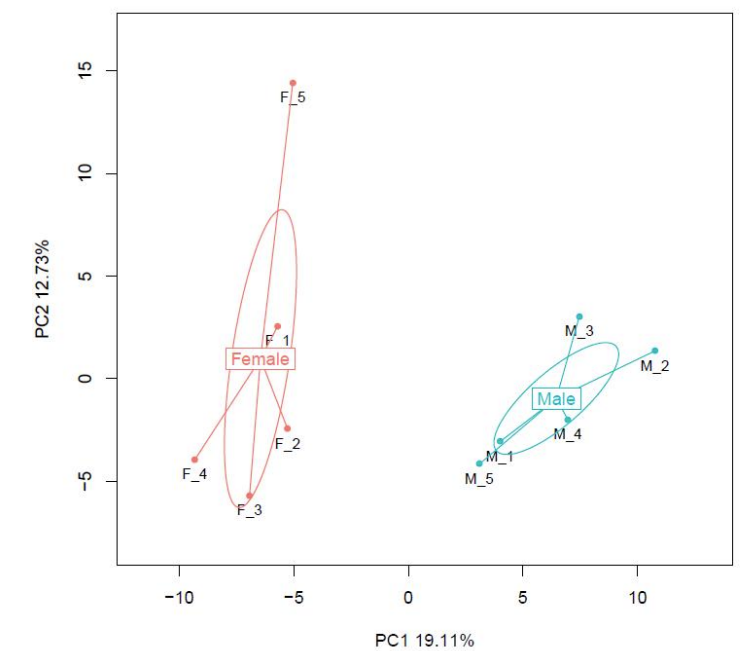
Pavyzdžio palyginimas: pagrindinių komponentų analizė
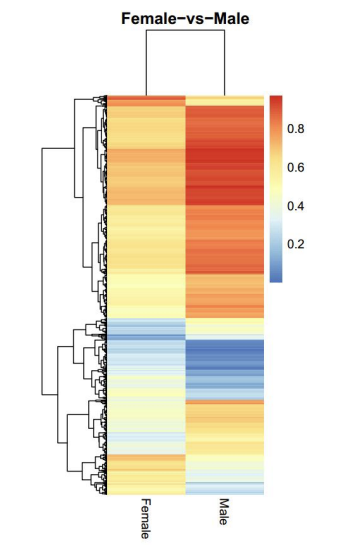
Diferencijiškai metiluotų regionų (DMR) analizė: šilumos žemėlapis
Naršykite mokslinių tyrimų pažangą, kurią palengvino BMKGene viso genomo bisulfito sekos nustatymo paslaugos per kuruojamą publikacijų rinkinį.
Li, Z. ir kt. (2022 m.) „Didelio tikslumo perprogramavimas į Leydig panašias ląsteles naudojant CRISPR aktyvavimą ir parakrininius veiksnius“,PNAS Nexus, 1 straipsnio 4 dalis. doi: 10.1093 / PNASNEXUS / PGAC179.
Tian, H. ir kt. (2023) „Kinų monozigotinių dvynių kūno sudėties genomo masto DNR metilinimo analizė“,Europos klinikinių tyrimų žurnalas, 53 (11), p. e14055. doi: 10.1111/ECI.14055.
Wu, Y. ir kt. (2022) „DNR metilinimas ir juosmens bei klubų santykis: epigenomo masto asociacijos tyrimas su Kinijos monozigotiniais dvyniais“,Endokrinologinių tyrimų žurnalas, 45(12), p. 2365–2376. doi: 10.1007/S40618-022-01878-4.