Hi-c pagrįsta chromatino sąveika
Aptarnavimo funkcijos
● „Illumina NovaSeq“ sekos nustatymas su PE150.
● Aptarnavimas reikalauja audinių mėginių, o ne ekstrahuotų nukleorūgščių, kad būtų galima susieti su formaldehidu ir išsaugoti DNR-baltymų sąveiką.
● „Hi-C“ eksperimentas apima lipnių galų apribojimą ir pabaigos taisymą biotinu, po to cirkuliarizuojant gautus neryškius galus, išsaugant sąveiką. Tada DNR nuleidžiama su streptavidino granulėmis ir išgryninama vėlesniam bibliotekos paruošimui.
Aptarnavimo pranašumai
●Optimalus apribojimo fermento dizainas: užtikrinti didelį HI-C efektyvumą skirtingoms rūšims, kurių sąveikos poros yra iki 93%.
●Didelės kompetencijos ir publikavimo įrašai:„Bmkgene“ turi didžiulę patirtį su> 2000 „Hi-C“ sekos projektų iš 800 skirtingų rūšių ir įvairių patentų. Daugiau nei 100 paskelbtų atvejų, kai kaupiamasis poveikio koeficientas viršija 900.
●Aukštos kvalifikacijos bioinformatikos komanda:Su vidaus patentais ir programine įranga autorių teisėmis, skirtomis „Hi-C“ eksperimentams ir duomenų analizei bei savarankiškai sukurtai vizualizacijos duomenų programinei įrangai.
●Palaikymas po pardavimo:Mūsų įsipareigojimas apima ne tik projekto pabaigą su 3 mėnesių aptarnavimo laikotarpiu po pardavimo. Per tą laiką mes siūlome projekto tolesnius veiksmus, pagalbą trikčių šalinimui ir klausimų ir atsakymų sesijoms, kad išspręstume bet kokius su rezultatus susijusius klausimus.
●Išsami anotacija: Mes naudojame kelias duomenų bazes, kad funkciškai komentuojame genus su identifikuotais variantais ir atliktume atitinkamą praturtinimo analizę, suteikdami įžvalgų apie kelis tyrimų projektus.
Aptarnavimo specifikacijos
| Biblioteka | Sekos strategija | Rekomenduojamas duomenų išvestis | „Hi-C“ signalo skiriamoji geba |
| „Hi-C“ biblioteka | „Illumina PE150“ | Chromatino kilpa: 150x TAD: 50x | Chromatino kilpa: 10 kb TAD: 40 kb |
Paslaugų reikalavimai
| Mėginio tipas | Reikalaujama suma |
| Gyvūnų audinys | ≥2g |
| Viso kraujo | ≥2ml |
| Grybeliai | ≥1g |
| Augalas- jaunas audinys | 1g/alikvotinė |
| Kultivuotos ląstelės | ≥1x107 |
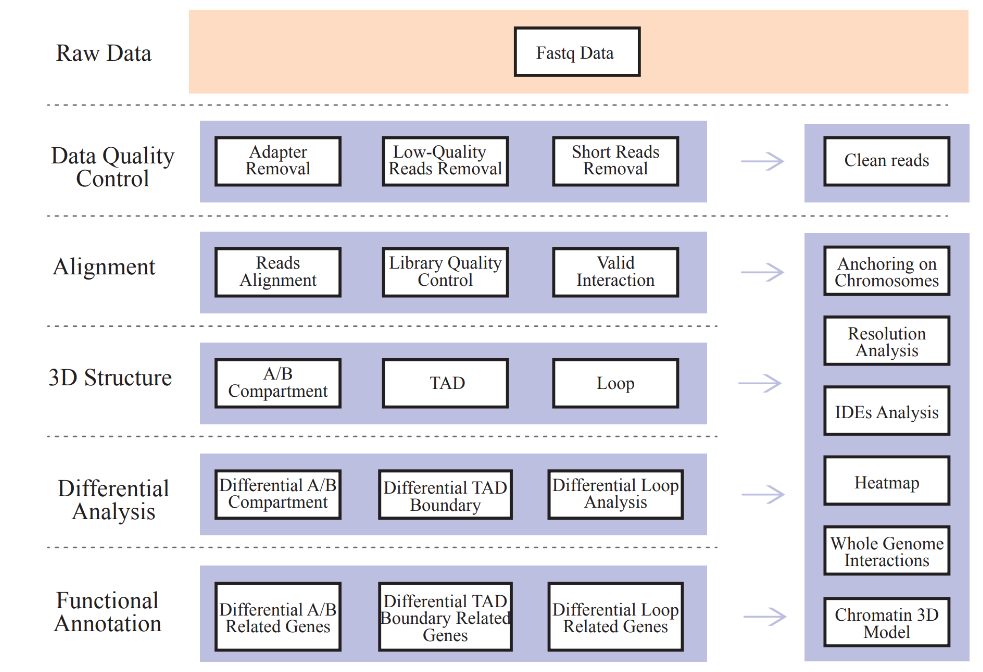
Apima šią analizę:
● Neapdoroti duomenys QC;
● Žemėlapis ir HI-C biblioteka QC: galiojančios sąveikos poros ir sąveikos skilimo eksponentai (IDE);
● Genomo sąveikos profiliavimas: cis/trans-analizė ir hi-c sąveikos žemėlapis;
● A/B skyrių pasiskirstymo analizė;
● TADS ir chromatino kilpų identifikavimas;
● 3D chromatino struktūros elementų diferencinė analizė tarp mėginių ir atitinkamas susijusių genų funkcinis anotacija.
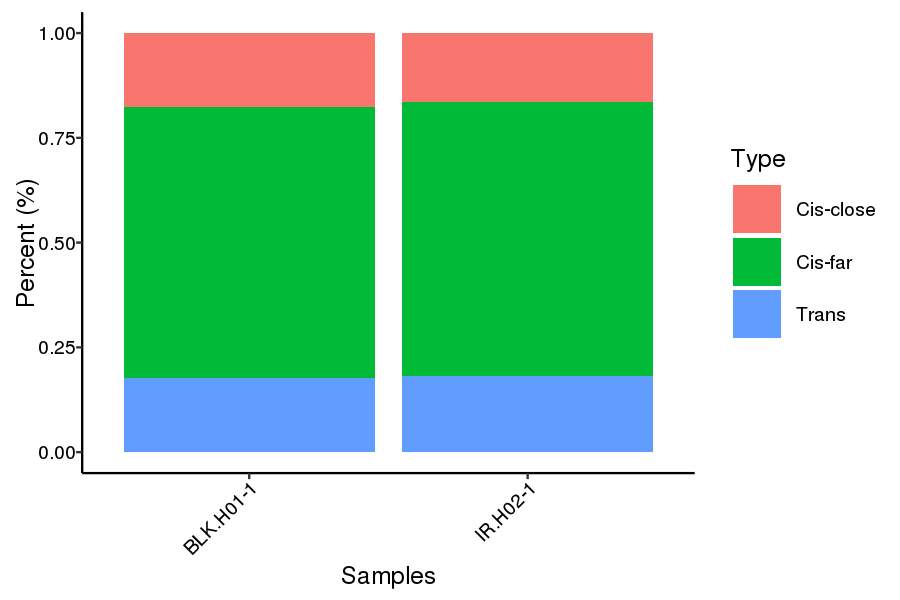
NVS ir trans -proporcijos pasiskirstymas
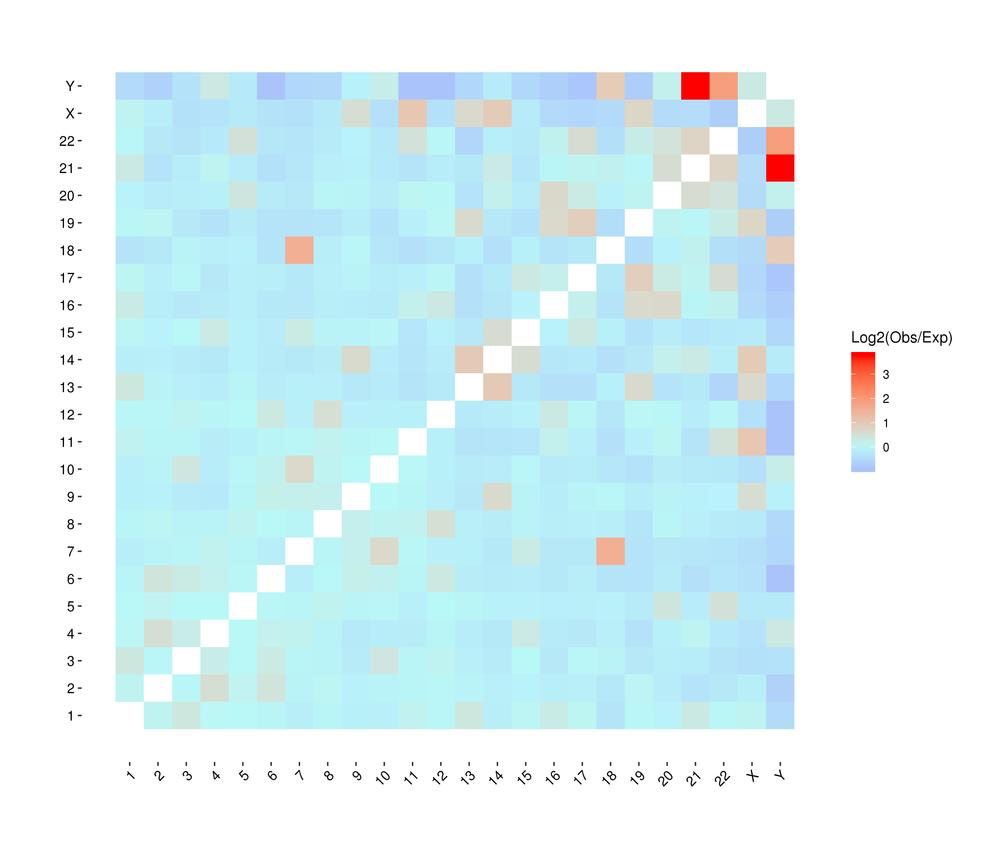
Chromosomų sąveikos tarp mėginių šilumos žemėlapis
A/B skyrių genomo pasiskirstymas visame pasaulyje
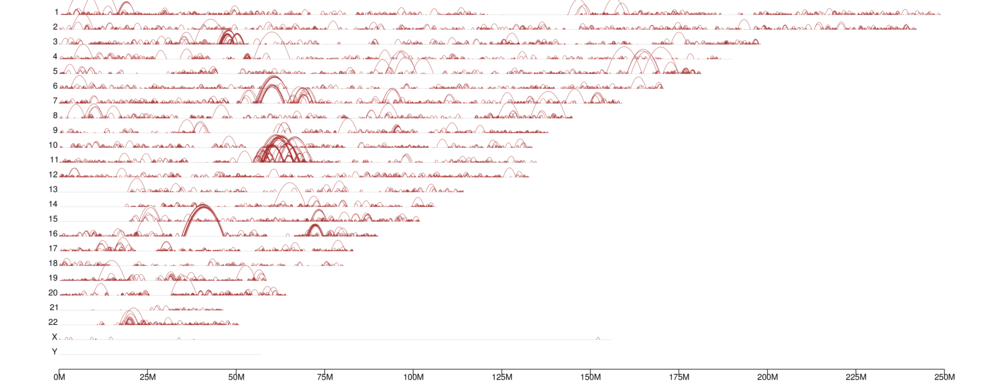
Genomo visos chromatino kilpų pasiskirstymas
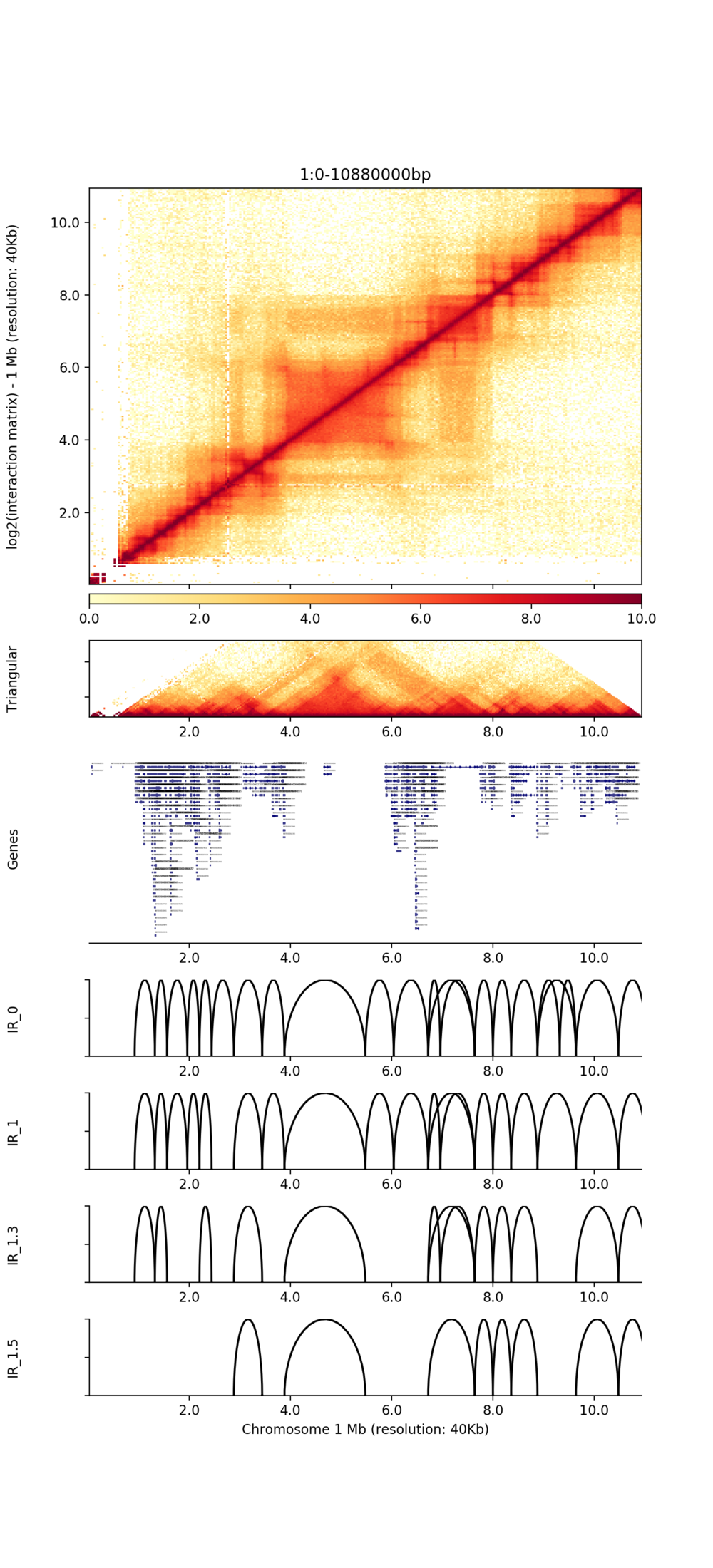
TADS vizualizacija
Ištirkite tyrimų pažangą, kurią palengvino BMKGENE „HI-C“ sekos nustatymo paslaugos, per kuruojamą leidinių kolekciją.
Meng, T. et al. (2021) „Lyginamoji integruota daugialypė omikos analizė nustato Ca2 kaip naują chordomos taikinį“,Neuro onkologija, 23 (10), p. 1709–1722. doi: 10.1093/neuonc/noAb156.
Xu, L. et al. (2021 m.) 3D dezorganizavimas ir genomo pertvarkymas suteikia įžvalgos apie NAFLD patogenezę integruotą hi-C, nanoporą ir RNR seką ',„Acta Pharmaceutica Sinica B“, 11 (10), p. 3150–3164. doi: 10.1016/j.apsb.2021.03.022.