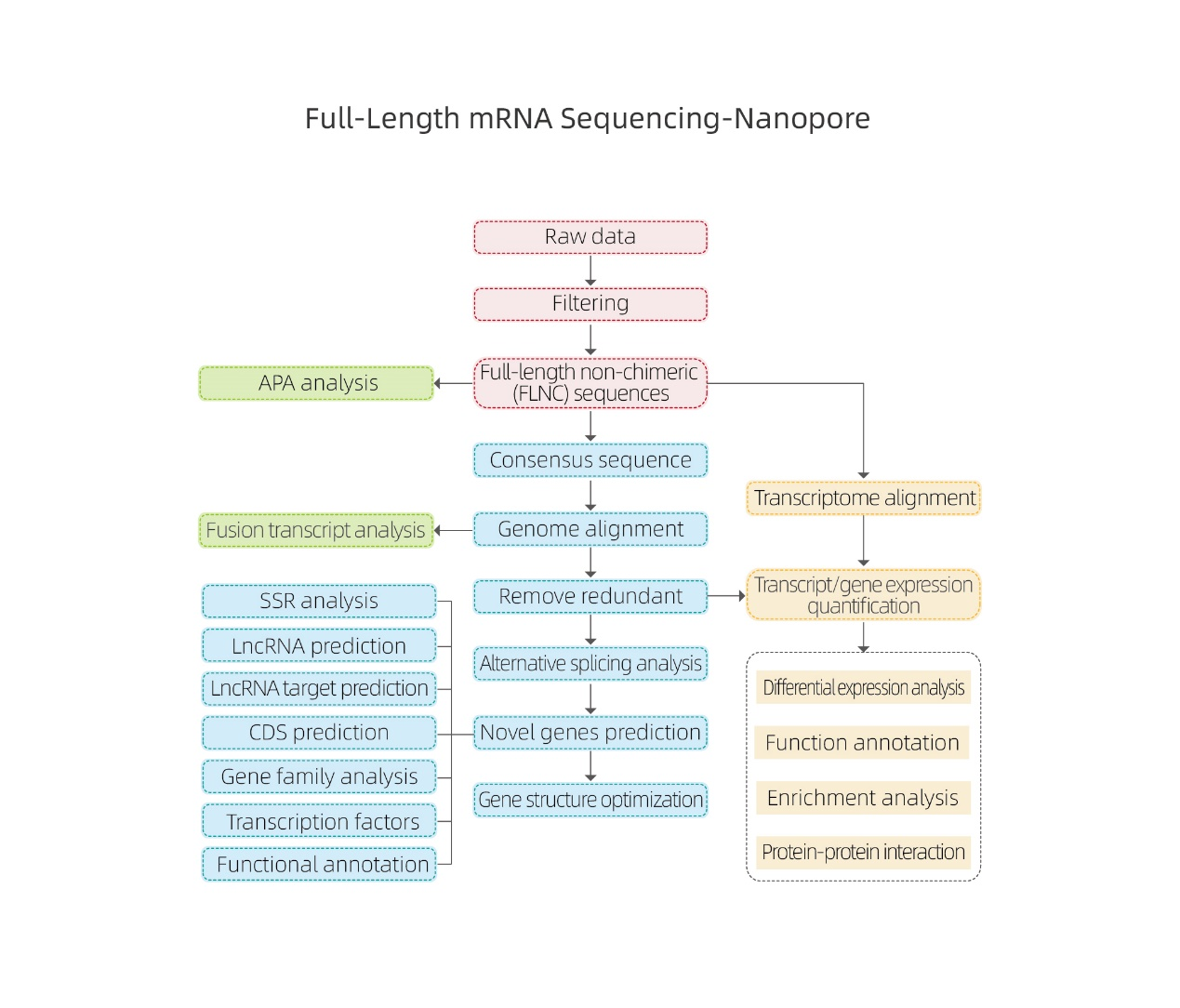
Viso ilgio mRNR sekos nustatymas-nanoporas
Savybės
● Poli-A mRNR surinkimas, po to cDNR sintezė ir bibliotekos paruošimas
● Viso ilgio nuorašų seka
● Bioinformatinė analizė, pagrįsta suderinimu su etaloniniu genomu
● Bioinformatinė analizė apima ne tik ekspresiją genų ir izoformų lygiu, bet ir lncRNR, genų susiliejimo, poliadenilinimo ir genų struktūros analizę.
Paslaugos privalumai
●Išraiškos kiekybinis įvertinimas izoformų lygiu: leidžia atlikti išsamią ir tikslią ekspresijos analizę, atskleidžiant pokyčius, kurie gali būti užmaskuoti analizuojant visą geno ekspresiją
●Sumažintas duomenų poreikis:Palyginti su naujos kartos sekos nustatymu (NGS), nanoporų sekos nustatymui keliami mažesni duomenų reikalavimai, todėl naudojant mažesnius duomenis galima pasiekti lygiaverčius genų ekspresijos kiekybinio prisotinimo lygius.
●Didesnis išraiškos kiekybinio įvertinimo tikslumas: tiek genų, tiek izoformų lygiu
●Papildomos transkriptinės informacijos identifikavimas: alternatyvus poliadenilinimas, suliejimo genai ir lcnRNR bei jų tiksliniai genai
●Plati ekspertizė: Mūsų komanda kiekvienam projektui suteikia daug patirties, įvykdžiusi daugiau nei 850 Nanopore pilno ilgio transkripto projektų ir apdorojusi daugiau nei 8000 pavyzdžių.
●Pagalba po pardavimo: mūsų įsipareigojimas apima ne tik projekto užbaigimą, bet ir 3 mėnesių aptarnavimo po pardavimo laikotarpį. Per šį laiką siūlome tolesnius projekto veiksmus, trikčių šalinimo pagalbą ir klausimų ir atsakymų sesijas, kad galėtume atsakyti į visas su rezultatais susijusias užklausas.
Pavyzdžių reikalavimai ir pristatymas
| biblioteka | Sekos nustatymo strategija | Rekomenduojami duomenys | Kokybės kontrolė |
| Poli A praturtintas | Illumina PE150 | 6/12 Gb | Vidutinis kokybės balas: Q10 |
Pavyzdiniai reikalavimai:
Nukleotidai:
| Konc. (ng/μl) | Kiekis (μg) | Grynumas | Sąžiningumas |
| ≥ 100 | ≥ 1,0 | OD260/280=1,7-2,5 OD260/230=0,5-2,5 Ant gelio rodomas ribotas baltymų ar DNR užterštumas arba jo nėra. | Augalams: RIN≥7,0; Gyvūnams: RIN≥7,5; 5,0≥28S/18S≥1,0; ribotas arba jo nėra |
● Augalai:
Šaknis, stiebas arba žiedlapis: 450 mg
Lapai arba sėklos: 300 mg
Vaisiai: 1,2 g
● Gyvūnas:
Širdis arba žarnynas: 300 mg
Viscera arba smegenys: 240 mg
Raumenys: 450 mg
Kaulai, plaukai arba oda: 1 g
● Nariuotakojai:
Vabzdžiai: 6g
Vėžiagyviai: 300 mg
● Visas kraujas: 1 vamzdelis
● Ląstelės: 106 ląstelės
Rekomenduojamas pavyzdžių pristatymas
Talpykla: 2 ml centrifugos mėgintuvėlis (Alavo folija nerekomenduojama)
Mėginio ženklinimas: Grupė+pakartojimas pvz. A1, A2, A3; B1, B2, B3.
Siuntimas:
1. Sausas ledas: Mėginiai turi būti supakuoti į maišus ir užkasti sausajame lede.
2. RNR stabilūs mėgintuvėliai: RNR mėginius galima džiovinti RNR stabilizavimo mėgintuvėlyje (pvz., RNAstable®) ir gabenti kambario temperatūroje.
Aptarnavimo darbų eiga
Nukleotidai:

Mėginio pristatymas

Bibliotekos statyba

Sekos nustatymas

Duomenų analizė

Paslaugos po pardavimo
Aptarnavimo darbų eiga
Audinys:

Eksperimento dizainas

Mėginio pristatymas

RNR ekstrahavimas

Bibliotekos statyba

Sekos nustatymas

Duomenų analizė

Paslaugos po pardavimo
● Neapdorotų duomenų apdorojimas
● Nuorašo identifikavimas
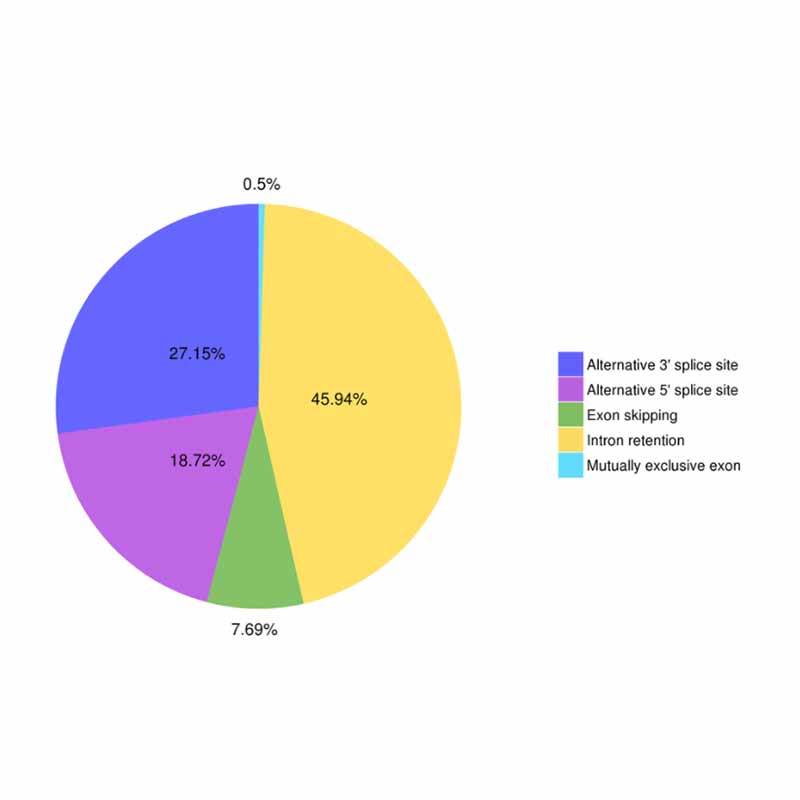
● Alternatyvus sujungimas
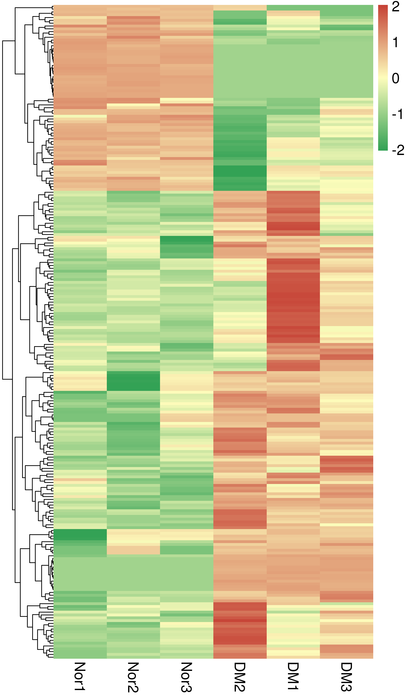
● Išraiškos kiekybinis įvertinimas genų lygiu ir izoformų lygiu
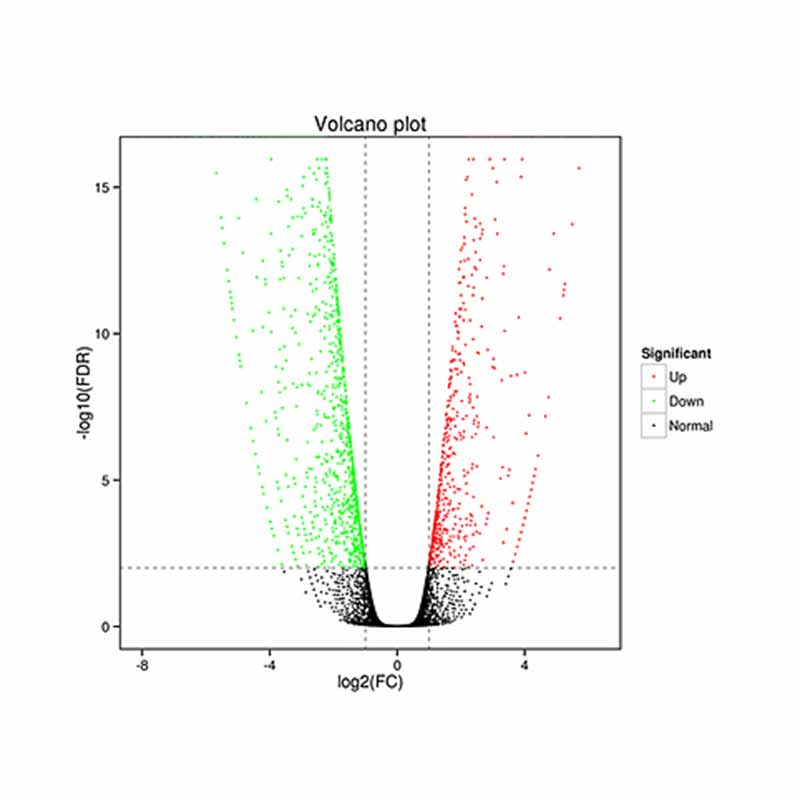
● Diferencinė išraiškų analizė
● Funkcijų anotacija ir praturtinimas (DEG ir DET)
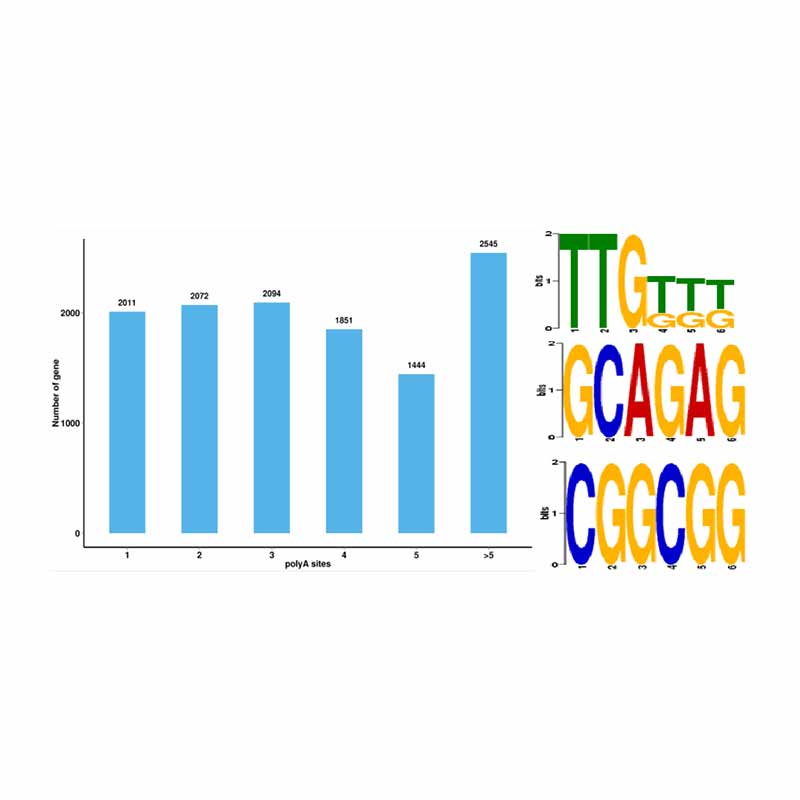
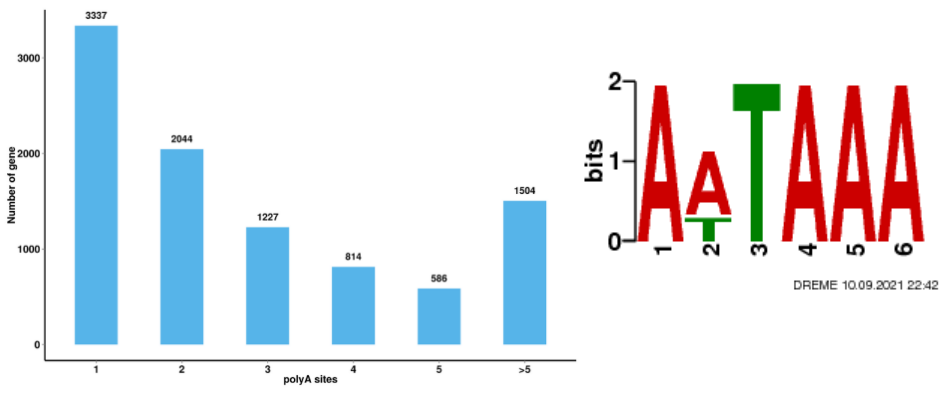
Alternatyvi sujungimo analizė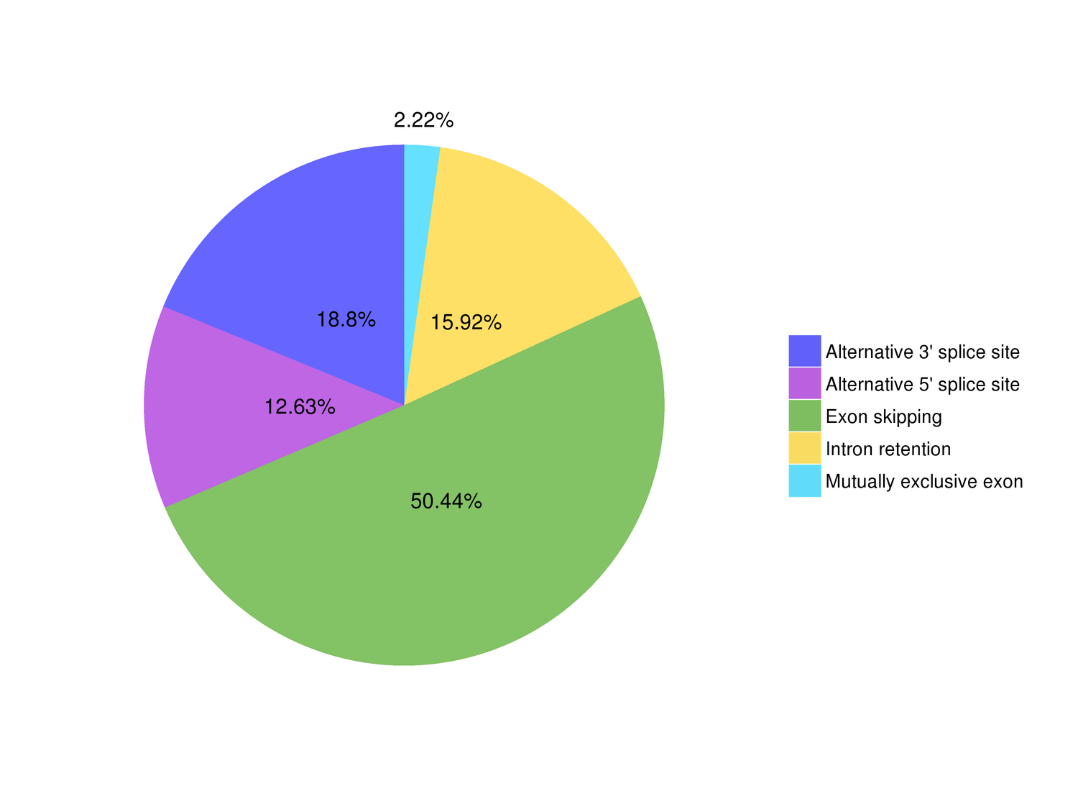 Alternatyvi poliadenilinimo analizė (APA)
Alternatyvi poliadenilinimo analizė (APA)
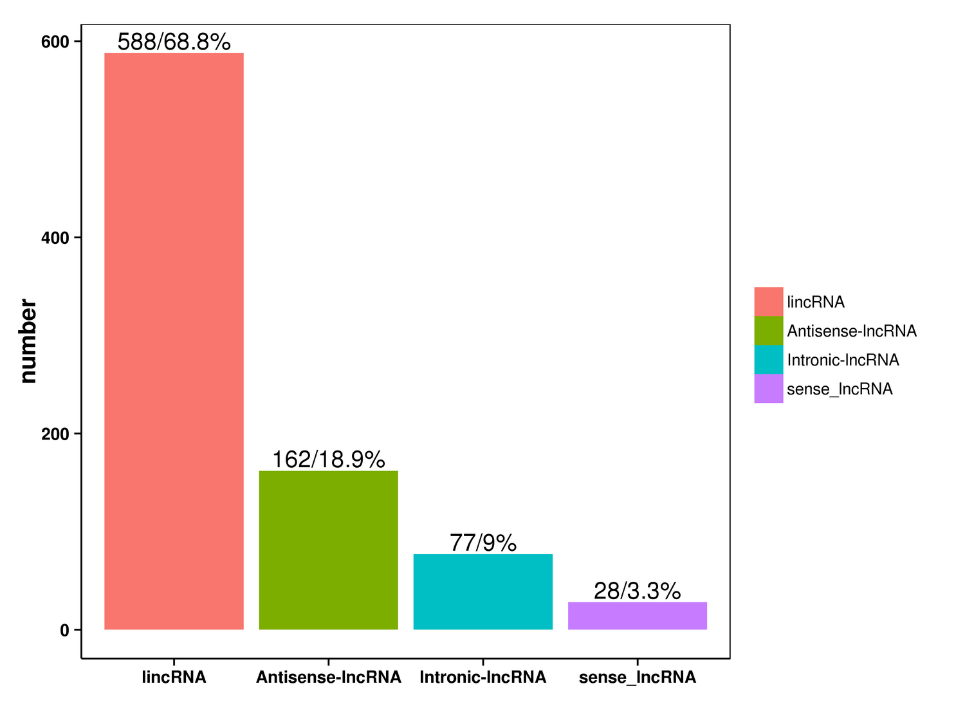
lncRNR prognozė
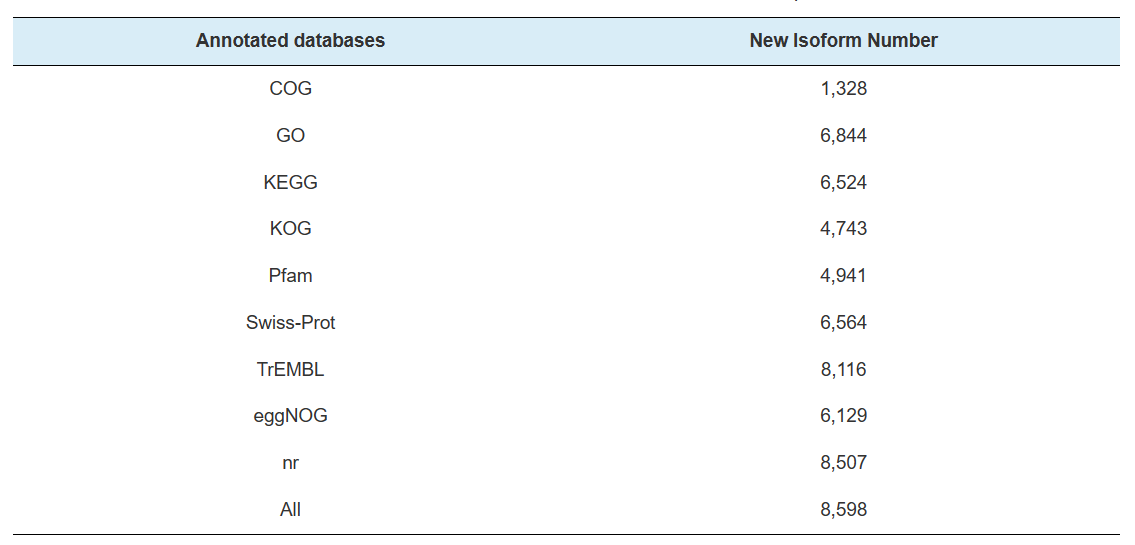
Naujų genų anotacija
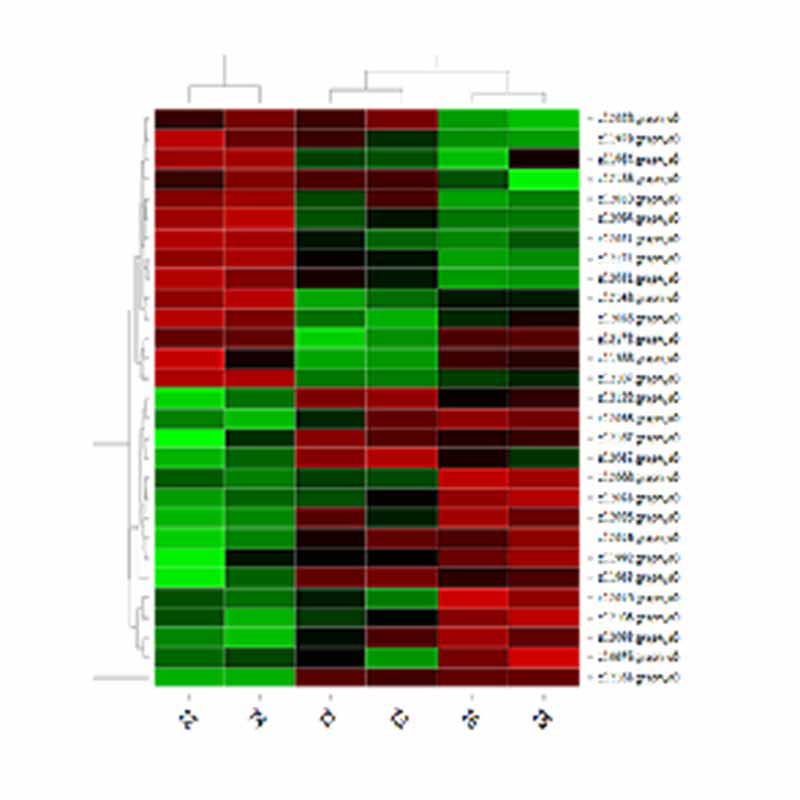
DET grupavimas
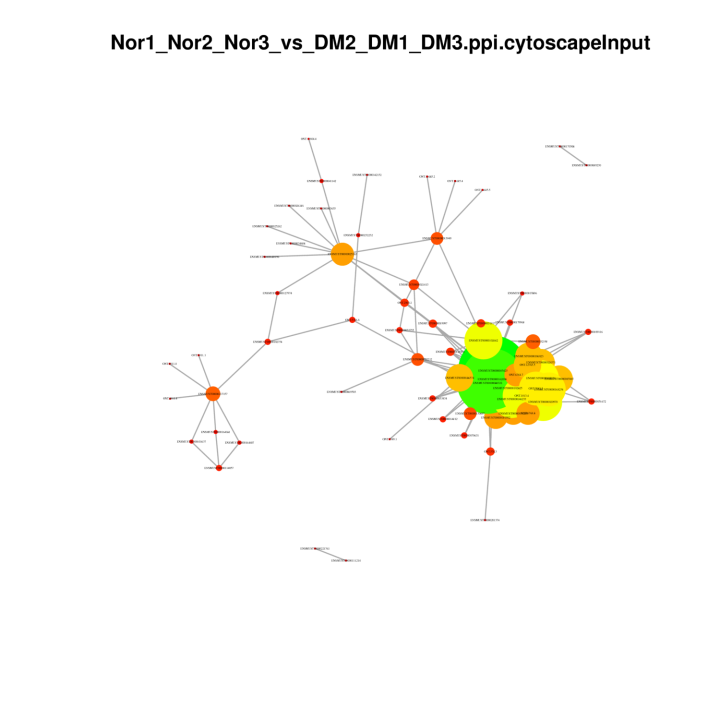
Baltymų ir baltymų tinklai DEG
Naršykite pažangą, kurią palengvino BMKGene Nanopore viso ilgio mRNR sekos nustatymo paslaugos per kuruojamą publikacijų kolekciją.
Gong, B. ir kt. (2023) „Epigenetinis ir transkripcinis sekrecinės kinazės FAM20C aktyvinimas kaip onkogenas sergant glioma“, „Journal of Genetics and Genomics“, 50 (6), p. 422–433. doi: 10.1016/J.JGG.2023.01.008.
Jis, Z. ir kt. (2023) „Viso ilgio limfocitų transkripto seka, reaguojanti į IFN-γ, atskleidžia Th1 iškreiptą imuninį atsaką plekšnėse (Paralichthys olivaceus)“, Fish & Shellfish Immunology, 134, p. 108636. doi: 10.1016/J.FSI.2023.108636.
Ma, Y. ir kt. (2023) „PacBio ir ONT RNR sekos nustatymo metodų lyginamoji analizė Nemopilema Nomurai nuodų identifikavimui“, Genomics, 115(6), p. 110709. doi: 10.1016/J.YGENO.2023.110709.
Yu, D. ir kt. (2023) „Nano-seq analizė atskleidžia skirtingą funkcinę tendenciją tarp egzosomų ir mikropūslelių, gautų iš hUMSC“, Stem Cell Research and Therapy, 14(1), p. 1–13. doi: 10.1186/S13287-023-03491-5/TABLES/6.