-

Metagenomic Sequencing -NGS
E Metagenom ass eng Sammlung vum Gesamtgenetesch Material vun enger gemëschter Gemeinschaft vun Organismen, sou wéi Ëmwelt- a mënschlech Metagenome. Et enthält Genome vu béid kultivéierbaren an onkultivéierbare Mikroorganismen. Shotgun metagenomesch Sequenzéierung mat NGS erméiglecht d'Studie vun dësen komplizéierte genomesche Landschaften, déi an Ëmweltproben agebonne sinn, andeems se méi wéi taxonomesch Profiléierung ubidden, och granulär Abléck an Arten Diversitéit, Iwwerfloss Dynamik, a komplex Bevëlkerungsstrukturen. Zousätzlech zu taxonomesche Studien, bitt Shotgun Metagenomics och eng funktionell Genomik Perspektiv, déi d'Exploratioun vu kodéierte Genen an hir putative Rollen an ökologesche Prozesser erméiglecht. Schlussendlech dréit d'Grënnung vu Korrelatiounsnetzwierker tëscht geneteschen Elementer an Ëmweltfaktoren zu engem holistesche Verständnis vun der komplizéierter Zesummespill tëscht mikrobiellen Gemeinschaften an hirem ökologeschen Hannergrond bäi. Als Schlussfolgerung steet metagenomesch Sequenzéierung als e pivotal Instrument fir d'genomesch Intricacies vun diversen mikrobiellen Gemeinschaften z'entdecken, déi villsäiteg Bezéiungen tëscht Genetik an Ökologie an dëse komplexe Ökosystemer beliichten.
Plattformen: Illumina NovaSeq an DNBSEQ-T7
-

Metagenomic Sequencing-TGS
E Metagenom ass eng Sammlung vum genetesche Material vun enger gemëschter Gemeinschaft vun Organismen, sou wéi Ëmwelt- a mënschlech Metagenome. Et enthält Genome vu béid kultivéierbaren an onkultivéierbare Mikroorganismen. Metagenomesch Sequenzéierung erméiglecht d'Studie vun dësen komplizéierte genomesche Landschaften, déi an ökologesche Proben agebonne sinn, andeems se méi wéi taxonomesch Profiler ubidden. Et bitt och eng funktionell Genomik Perspektiv andeems se déi kodéiert Genen an hir putativ Rollen an Ëmweltprozesser exploréieren. Wärend traditionell Gewier Approche mat Illumina Sequencing wäit an metagenomesche Studien benotzt goufen, hunn d'Entstoe vu Nanopore a PacBio laang gelies Sequencing d'Feld geännert. Nanopore a PacBio Technologie verbesseren downstream bioinformatesch Analysen, notamment Metagenome Assemblée, fir méi kontinuéierlech Versammlungen ze garantéieren. Berichter weisen datt Nanopore-baséiert a PacBio-baséiert Metagenomics erfollegräich komplett a zougemaach bakteriell Genome vu komplexe Mikrobiome generéiert hunn (Moss, EL, et al., Nature Biotech, 2020). D'Integratioun vun Nanopore Liesungen mat Illumina Reads bitt eng strategesch Approche fir Feelerkorrektur, wat dem Nanopore seng inherent niddereg Genauegkeet reduzéiert. Dës synergistesch Kombinatioun profitéiert d'Stäerkte vun all Sequenzéierungsplattform, bitt eng robust Léisung fir potenziell Aschränkungen ze iwwerwannen an d'Präzisioun an d'Zouverlässegkeet vu metagenomeschen Analysen ze förderen.
Plattform: Nanopore PromethION 48, Illumia a PacBio Revio
-

Ganz Genom Bisulfit Sequencing (WGBS)
Whole Genome Bisulfite Sequencing (WGBS) steet als Gold-Standard-Methodologie fir déif Exploratioun vun der DNA-Methylatioun, speziell déi fënneft Positioun am Zytosin (5-mC), e pivotal Reguléierer vum Genausdrock an der cellulärer Aktivitéit. De Prinzip, deen WGBS ënnersträicht, beinhalt d'Bisulfitbehandlung, induzéieren d'Konversioun vun onmethyléierten Zytosinen op Uracil (C bis U), wärend methyléiert Zytosinen onverännert bleiwen. Dës Technik bitt Single-Basis Resolutioun, wat d'Fuerscher erlaabt de Methylome ëmfaassend z'ënnersichen an anormal Methylatiounsmuster ze entdecken, déi mat verschiddene Konditiounen assoziéiert, notamment Kriibs. Andeems Dir WGBS beschäftegt, kënnen d'Wëssenschaftler onparalleléiert Abléck an genombreet Methylatiounslandschaften kréien, e nuancéiert Verständnis vun den epigeneteschen Mechanismen ubidden, déi verschidde biologesch Prozesser a Krankheeten ënnersträichen.
-

Assay fir Transposase-Accessible Chromatin mat High Throughput Sequencing (ATAC-seq)
ATAC-seq ass eng High-Throughput Sequenzéierungstechnik déi fir Genom-breet Chromatin Accessibilitéitsanalyse benotzt gëtt. Et benotzt e méi déif Verständnis vun de komplexe Mechanismen vun der globaler epigenetescher Kontroll iwwer Genausdrock. D'Method benotzt eng hyperaktiv Tn5 Transposase fir gläichzäiteg opzeechnen Chromatin Regiounen ze fragmentéieren an ze markéieren andeems se Sequenzenadapteren asetzen. Déi spéider PCR Amplifikatioun resultéiert an der Schafung vun enger Sequenzéierungsbibliothéik, déi eng ëmfaassend Identifikatioun vun oppene Chromatinregiounen ënner spezifesche Raumzäitbedéngungen erlaabt. ATAC-seq bitt eng holistesch Vue op zougänglech Chromatinlandschaften, am Géigesaz zu Methoden déi sech eleng op Transkriptiounsfaktor Bindungsplazen oder spezifesch Histon-modifizéiert Regiounen konzentréieren. Duerch d'Sequenzéierung vun dësen oppene Chromatinregiounen, weist ATAC-seq Regiounen méi wahrscheinlech fir aktiv Reguléierungssequenzen a potenziellen Transkriptiounsfaktor Bindungsplazen op, bitt wäertvoll Abléck an déi dynamesch Modulatioun vum Genausdrock iwwer de Genom.
-

16S/18S/ITS Amplicon Sequencing-PacBio
D'16S an 18S rRNA Genen, zesumme mat der Internal Transcribed Spacer (ITS) Regioun, déngen als pivotal molekulare Fangerofdrockmarker wéinst hirer Kombinatioun vun héich konservéierten an hypervariablen Regiounen, wat se wäertvoll Tools mécht fir prokaryotesch an eukaryotesch Organismen ze charakteriséieren. Amplifikatioun an Sequenzéierung vun dëse Regioune bidden eng Isolatiounsfräi Approche fir d'mikrobial Zesummesetzung an Diversitéit iwwer verschidden Ökosystemer z'ënnersichen. Wärend Illumina Sequencing typesch kuerz hypervariabel Regioune wéi V3-V4 vun 16S an ITS1 zielt, gouf bewisen datt eng super taxonomesch Annotatioun erreechbar ass andeems se déi ganz Längt vun 16S, 18S, an ITS Sequenzéieren. Dës ëmfaassend Approche féiert zu méi héije Prozentsaz vu präzis klasséierte Sequenzen, fir e Resolutiounsniveau z'erreechen deen sech op d'Aartidentifikatioun erstreckt. PacBio's Single-Molecule Real-Time (SMRT) Sequenzéierungsplattform steet eraus andeems se héich präzis laang Liesungen (HiFi) ubidden, déi d'Volllängt Ampliconen ofdecken, mat der Präzisioun vun der Illumina Sequencing rivaliséieren. Dës Fäegkeet erlaabt d'Fuerscher en onvergläichleche Virdeel ze erreechen - eng Panoramavue vun der genetescher Landschaft. Déi erweidert Ofdeckung erhéicht d'Resolutioun an der Speziesannotatioun wesentlech, besonnesch a bakteriellen oder Pilzgemeinschaften, wat e méi déif Verständnis vun de Schwieregkeete vu mikrobiellen Populatiounen erméiglecht.
-

16S/18S/ITS Amplicon Sequencing-NGS
Amplicon Sequenzéierung mat Illumina Technologie, speziell op d'16S, 18S, an ITS genetesch Marker gezielt, ass eng mächteg Method fir d'Phylogenie, d'Taxonomie, an d'Arten Heefegkeet bannent mikrobielle Gemeinschaften z'entdecken. Dës Approche implizéiert d'Sequenzéierung vun den hypervariablen Regiounen vun Haushaltsgeneteschen Marker. Ursprénglech agefouert als molekulare Fangerofdrock vunWoesen et alan 1977, dëser Technik huet microbiome profiling revolutionéiert vun Isolatioun-gratis Analysen. Duerch d'Sequenzéierung vun 16S (Bakterien), 18S (Pilze), an Internal Transcribed Spacer (ITS, Pilze), kënnen d'Fuerscher net nëmmen reichend Arten identifizéieren, awer och selten an onidentifizéierter. Breet ugeholl als pivotal Tool, Amplicon Sequenzéierung ass instrumental ginn fir differenziell mikrobielle Kompositioune iwwer verschidden Ëmfeld ze erkennen, dorënner de mënschleche Mond, Darm, Hocker, an doriwwer eraus.
-

Bakteriell a Pilz ganz Genom Re-Sequencing
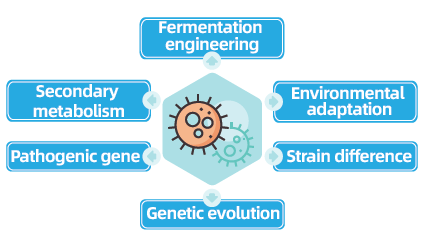
Bakteriell a Pilz Ganzgenom Re-Sequenzéierungsprojete si pivotal fir mikrobiell Genomik ze förderen andeems se d'Réalisatioun an de Verglach vu mikrobiellen Genomen erméiglechen. Dëst erliichtert Fermentatiounstechnik, d'Optimiséierung vun industrielle Prozesser, an d'Exploratioun vu sekundäre Metabolismusweeër. Ausserdeem ass Pilz a bakteriell Re-Sequenzéierung entscheedend fir d'Ëmweltadaptatioun ze verstoen, Stämme optimiséieren an d'genetesch Evolutiounsdynamik z'entdecken, mat breet Implikatiounen an der Medizin, der Landwirtschaft an der Ëmweltwëssenschaft.
-

PacBio-Full-Length 16S/18S/ITS Amplicon Sequencing
Amplicon (16S / 18S / ITS) Plattform ass mat Joeren Erfarung an der mikrobieller Diversitéitsprojetanalyse entwéckelt, déi standardiséierter Basisanalyse a personaliséierter Analyse enthält: Basisanalyse deckt den Mainstream Analyseinhalt vun der aktueller mikrobieller Fuerschung, den Analyseinhalt ass räich an ëmfaassend, an d'Analyseresultater ginn a Form vu Projetsberichter presentéiert; Den Inhalt vun der personaliséierter Analyse ass divers. Echantillon kënnen ausgewielt ginn a Parameteren kënne flexibel no dem Basisanalysebericht a Fuerschungszwecker festgeluecht ginn, fir personaliséiert Ufuerderungen ze realiséieren. Windows Betribssystem, einfach a séier.
-

PacBio-Voll-Längt Transkriptom (Non-Referenz)
Wann Dir Pacific Biosciences (PacBio) Isoform Sequenzéierungsdaten als Input hëlt, ass dës App fäeg Transkriptsequenzen vu voller Längt z'identifizéieren (ouni Versammlung). Andeems Dir voll Längt Sequenzen géint Referenzgenom kartéiert, kënne Transkriptiounen duerch bekannte Genen optimiséiert ginn, Transkriptiounen, Kodéierungsregiounen, asw. Gemeinsam Analyse mat NGS Transkriptom Sequenzéierungsdaten erméiglecht méi ëmfaassend Annotatioun a méi präzis Quantifikatioun am Ausdrock um Transkriptniveau, wat gréisstendeels downstream differentiell Ausdrock a funktionell Analyse profitéiert.
-

Reduced Representation Bisulfite Sequencing (RRBS)
Reduzéiert Representatioun Bisulfite Sequencing (RRBS) ass entstanen als eng kosteneffektiv an effizient Alternativ zu Whole Genome Bisulfite Sequencing (WGBS) an der DNA Methylatiounsfuerschung. Wärend WGBS iwwergräifend Abléck ubitt andeems de ganze Genom bei enger eenzeger Basisopléisung ënnersicht gëtt, kënnen hir héich Käschte e limitéierende Faktor sinn. RRBS mitigéiert strategesch dës Erausfuerderung andeems se e representativen Deel vum Genom selektiv analyséiert. Dës Methodik baséiert op der Beräicherung vun CpG Insel-räich Regiounen vun MspI Spaltung gefollegt vun Gréisst Auswiel vun 200-500/600 bps Fragmenter. Dofir sinn nëmmen Regiounen proximal zu CpG Inselen sequenzéiert, während déi mat wäitem CpG Inselen aus der Analyse ausgeschloss sinn. Dëse Prozess, kombinéiert mat Bisulfit-Sequenzéierung, erlaabt eng héichopléisend Detektioun vun der DNA-Methylatioun, an d'Sequenzéierungs Approche, PE150, konzentréiert sech speziell op d'Enn vun den Inserts anstatt d'Mëtt, wat d'Effizienz vun der Methylatiounsprofiléierung erhéicht. De RRBS ass en onschätzbare Tool dat kosteneffizient DNA Methylatiounsfuerschung erméiglecht an d'Wëssen iwwer epigenetesch Mechanismen fördert.
-

Prokaryotesch RNA Sequencing
RNA Sequencing erméiglecht déi ëmfaassend Profiléierung vun all RNA Transkriptiounen bannent Zellen ënner spezifesche Bedéngungen. Dës opzedeelen Technologie déngt als e mächtegt Tool, enthüllt komplizéiert Genausdrockprofiler, Genstrukturen a molekulare Mechanismen verbonne mat diverse biologesche Prozesser. Breet ugeholl an fundamental Fuerschung, klinescher Diagnostik, an Drogenentwécklung, RNA Sequencing bitt Abléck an d'Intricacies vun der cellulärer Dynamik an der genetescher Reguléierung. Eis prokaryotesch RNA Probe Veraarbechtung ass fir prokaryotesch Transkriptomen ugepasst, involvéiert rRNA Ausarmung a Richtungsbibliothéik Virbereedung.
Plattform: Illumina NovaSeq
-

Metatranskriptom Sequenzéierung
Mat Hëllef vun Illumina Sequencing Technologie, BMKGENE's Metatranscriptome Sequencing Service enthüllt den dynamesche Genausdrock vun enger diverser Palette vu Mikroben, spannen Eukaryoten op Prokaryoten a Viren, bannent natierlechen Ëmfeld wéi Buedem, Waasser, Mier, Hocker, an den Darm. Eise ëmfaassende Service erlaabt d'Fuerscher sech a komplexe mikrobielle Gemeinschaften komplett Genausdrockprofiler ze verdéiwen. Nieft der taxonomescher Analyse erliichtert eise Metatranskriptom Sequenzéierungsservice d'Erfuerschung a funktionell Beräicherung, léisst d'Liicht op differenziell ausgedréckte Genen an hir Rollen. Entdeckt e Räichtum vu biologeschen Abléck wéi Dir déi komplex Landschafte vum Genausdrock, taxonomescher Diversitéit, a funktioneller Dynamik an dësen verschiddenen Ëmweltnische navigéiert.
-

De novo Fungal Genome Assemblée
BMKGENE bitt villsäiteg Léisunge fir Pilzgenomen, këmmere sech op verschidde Fuerschungsbedierfnesser a gewënschte Genom Vollständegkeet. D'Benotzung vun kuerz-liesen Illumina Sequenzen eleng erlaabt d'Generatioun vun engem Entworf Genom. Kuerz Liesen a laang gelies Sequenzéierung mat Nanopore oder Pacbio gi kombinéiert fir e méi raffinéiert Pilzgenom mat méi laange Contigs. Ausserdeem verbessert d'Integratioun vun Hi-C Sequenzéierung d'Fäegkeeten weider, wat d'Erreeche vun engem komplette Chromosom-Niveau Genom erméiglecht.