Hi-C Baséiert Genom Assemblée
Service Fonctiounen
● Sequencing op Illumina NovaSeq mat PE150.
● Service erfuerdert Tissueproben, amplaz vun extrahéierten Nukleinsäuren, fir mat Formaldehyd ze crosslinken an d'DNA-Protein Interaktiounen ze konservéieren.
● Den Hi-C Experiment beinhalt d'Restriktioun an d'Ennreparatur vun de plakeg Enden mat Biotin, gefollegt duerch d'Zirkulariséierung vun de resultéierende stompegen Ennen, während d'Interaktiounen erhalen. D'DNA gëtt dann mat Streptavidin Perlen erofgezunn a gereinegt fir déi spéider Bibliothéikpräparatioun.
Service Virdeeler

Iwwersiicht vun Hi-C
(Lieberman-Aiden E et al.Wëssenschaft, 2009)
●Eliminéieren de Besoin fir genetesch Bevëlkerungsdaten:Hi-C ersetzt déi wesentlech Informatioun déi néideg ass fir Contig Verankerung.
●Héich Marker Dicht:féiert zu engem héije Contig Verankerungsverhältnis iwwer 90%.
●Extensiv Expertise a Verëffentlechungsrecords:BMKGene huet grouss Erfahrung mat méi wéi 2000 Fäll vun Hi-C Genome Assemblée vun 1000 verschidden Arten a verschidde Patenter. Iwwer 200 publizéiert Fäll hunn en akkumulativen Impaktfaktor vun iwwer 2000.
●Héichqualifizéiert Bioinformatik Team:mat internen Patenter a Software-Urheberrechter fir Hi-C Experimenter an Datenanalyse, déi selbstentwéckelt Visualiséierungsdatensoftware erméiglecht manuell Blockbeweegung, ëmgedréint, zréckzéien an nei ze maachen.
●Post-Verkaf Support:Eist Engagement erstreckt sech iwwer d'Réalisatioun vum Projet mat enger 3-Mount After-Sale Service Period. Wärend dëser Zäit bidde mir de Projet Suivi, Troubleshooting Hëllef, a Q&A Sessiounen fir all Ufroen am Zesummenhang mat de Resultater unzegoen.
●Iwwergräifend Annotatioun: Mir benotze verschidde Datenbanken fir d'Genen mat identifizéierten Variatiounen funktionell annotéieren an déi entspriechend Beräicherungsanalyse auszeféieren, Abléck iwwer verschidde Fuerschungsprojeten ze bidden.
Service Spezifikatioune
| Bibliothéik Virbereedung | Sequenzéierungsstrategie | Recommandéiert Datenausgang | Qualitéitskontroll |
| Hi-C Bibliothéik | Illumina NovaSeq PE150 | 100x vun | Q30 ≥ 85% |
Prouf Ufuerderunge
| Tissue | Néideg Betrag |
| Déier Viscera | ≥ 2g |
| Déier Muskel | |
| Mammalian Blutt | ≥ 2 ml |
| Gefligel / Fësch Blutt | |
| Planz - Frësch Leaf | ≥ 3g |
| Kulturéiert Zellen | ≥ 1x107 |
| Insekt | ≥ 2g |
Service Work Flow

Experimenter Design

Echantillon Liwwerung

Bibliothéik Bau

Sequenzéieren

Donnéeën Analyse

No-Verkaf Servicer
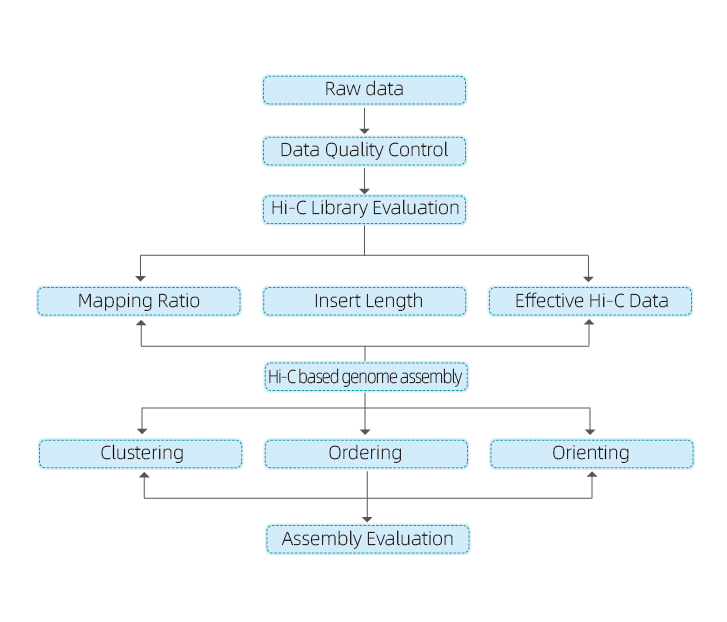
1) Matière Daten QC
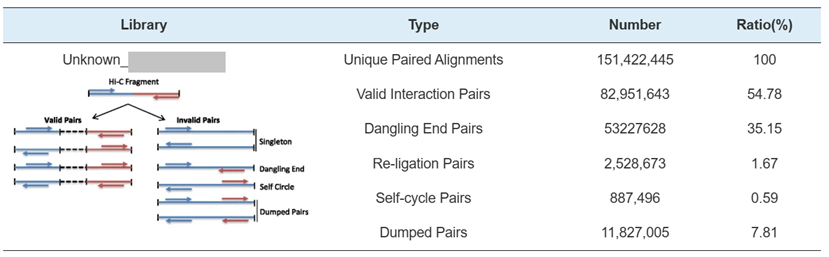
2) Hi-C Bibliothéik QC: Schätzung vun valabel Hi-C Interaktiounen
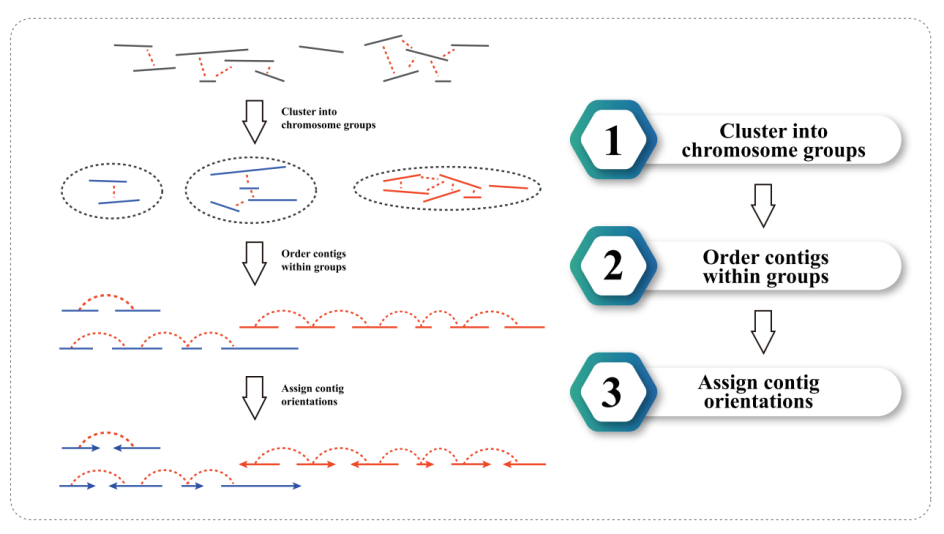
3) Hi-C Assemblée: Clustering vu Contigs a Gruppen, gefollegt vu Contig Uerdnung bannent all Grupp an zougewisen Contig Orientéierung
4) Hi-C Evaluatioun
Hi-C Bibliothéik QC - Schätzung vun Hi-C gëlteg Interaktiounsparen
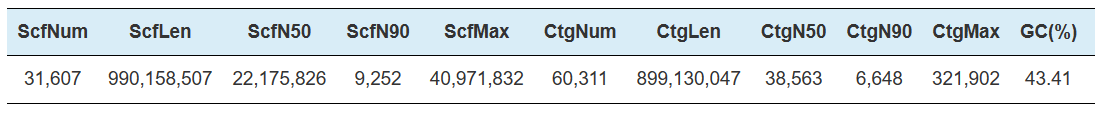
Hi-C Assemblée - Statistiken
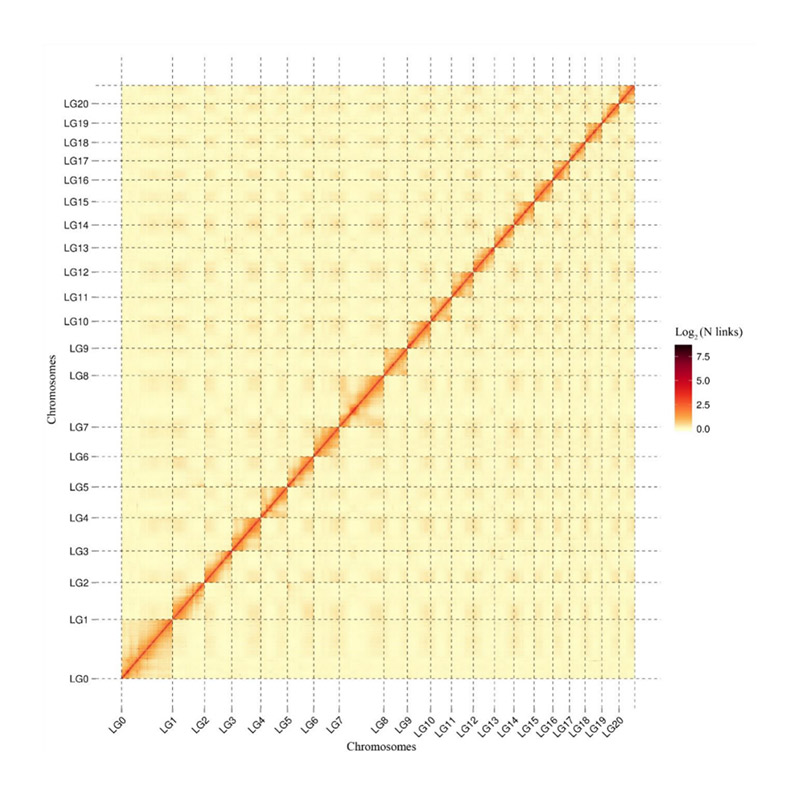
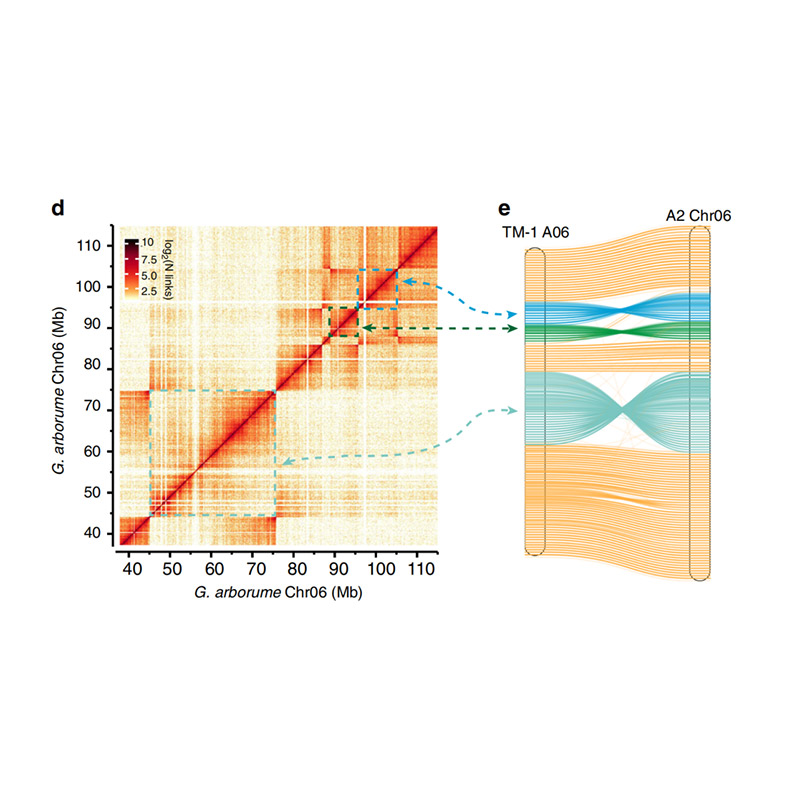
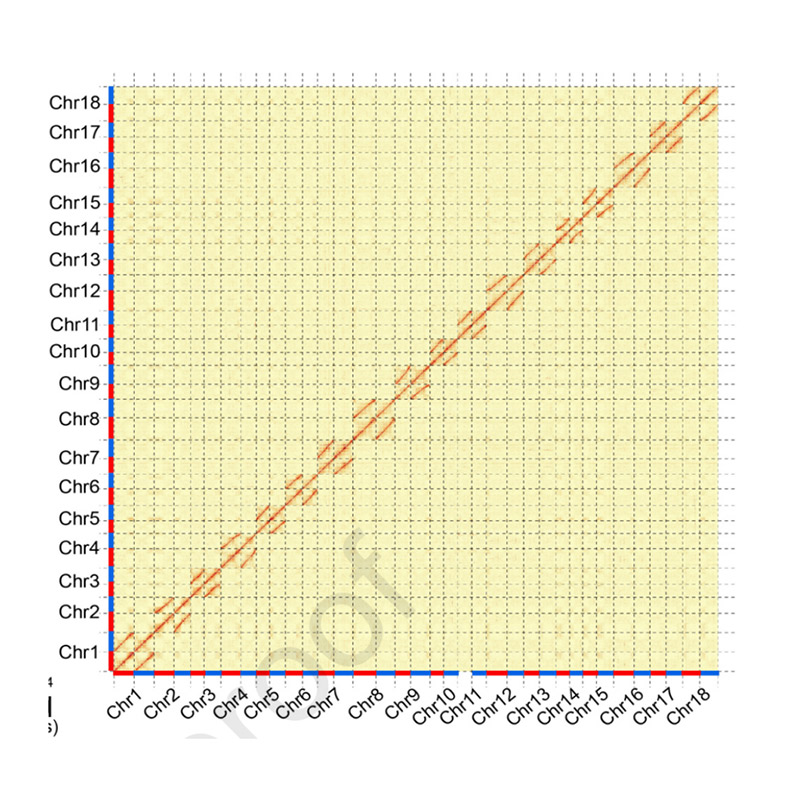
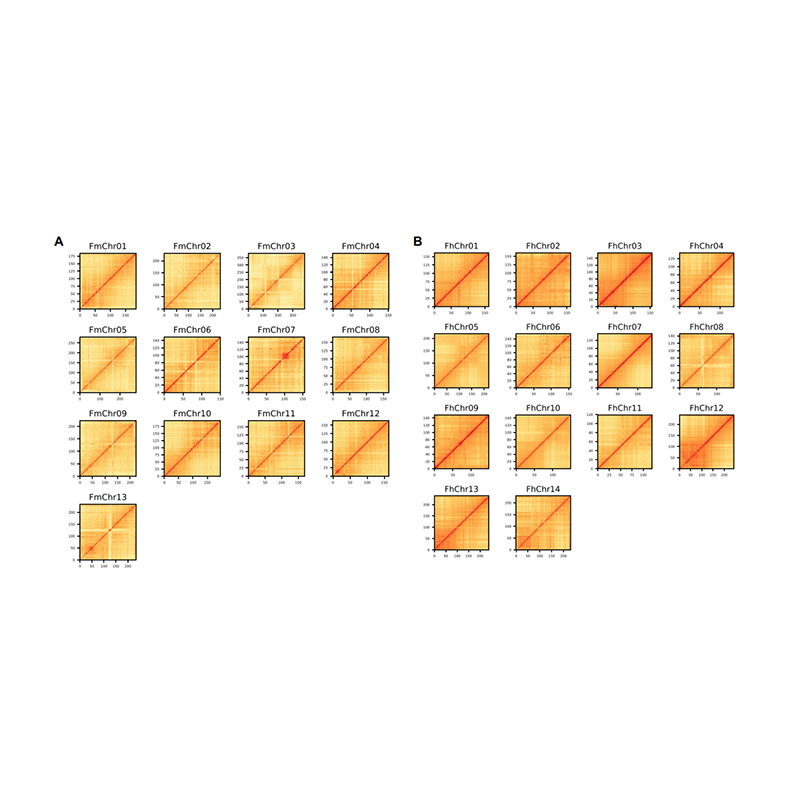
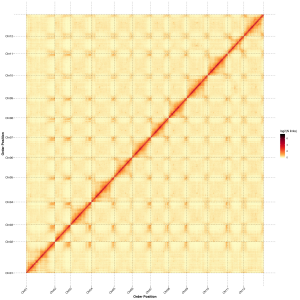
Post-Assemblée Evaluatioun - Hëtzt Kaart vun Signal Intensitéit tëscht Poubellen
Entdeckt d'Fortschrëtter erliichtert vum BMKGene's Hi-C Assemblée Servicer duerch eng curéiert Sammlung vu Publikatiounen.
Tian, T. et al. (2023) 'Genome Assemblée an genetesch Dissection vun engem prominente dréchent-resistente Maiskemplasma', Nature Genetics 2023 55:3, 55(3), S. 496–506. doi: 10.1038/s41588-023-01297-y.
Wang, ZL et al. (2020) 'A Chromosome-Scale Assembly of the Asian Honeybee Apis cerana Genome', Frontiers in Genetics, 11, p. 524140. doi: 10.3389/FGENE.2020.00279/BIBTEX.
Zhang, F. et al. (2023) 'Entdeckung vun der Evolutioun vun der Tropane Alkaloid Biosynthese duerch Analyse vun zwee Genomen an der Solanaceae Famill', Nature Communications 2023 14:1, 14(1), S. 1-18. doi: 10.1038/s41467-023-37133-4.
Zhang, X, et al. (2020) 'Genomen vum Banyan Tree a Pollinator Wasp bidden Abléck an Fig-Wesp Coevolution', Zell, 183(4), S. 875-889.e17. doi: 10.1016/J.CELL.2020.09.043