Bulkéiert segregant Analyse
Service Virdeeler

Takagi et al., De Planz Journal, 2013
● richteg Lokaliséierungen: Mixen Bëscher mat 30 + 30 + 30 bis 200 Seite fir Hannergrond Geräischer ze minimiséieren; Net-synonym mutatants-baséiert Kandidatin Regioun Prognosen.
● Fir eng Analyse: Pikter wiert, d'Funioun Invissioun, doriwwerzéckséiweg, Speed, etc.
● Méi séier Ëffnungszäit: Rapid Gen Lokalisatioun bannent 45 Aarbechtsdeeg.
● Viskier hunn d'Rees nëmme bei Tourhike vun der Rees bäibréngen, mat engem öpsproochege Kanner, Bësch, Fliuchtten, agerk.
Service Spezifikatioune
Populatioun:
Segregéierend Prilly vun Eltere mat opposéierende Phenotypen.
zB F2 Progony, Backcrossing (BC), recommandant Inbredlinn (Ril)
Mixen Pool
Fir qualitativ Streck: 30 bis 50 Persounen (minimum 20) / bulk
Fir quantitativ Traumis: Top 5% op 10% Eenzelen mat entweder extrem phenotypen an der ganzer Bevëlkerung (minimum 30 + 30).
Recommandéiert SEKENTING TIMET
Op d'mannst 20x / Elterendeel an 1x / Nofolger Individuum (z. B. fir Nofolger vun 30 + 30 Eenzelpersoun, Sequenzen Déift 30x pro Vulk)
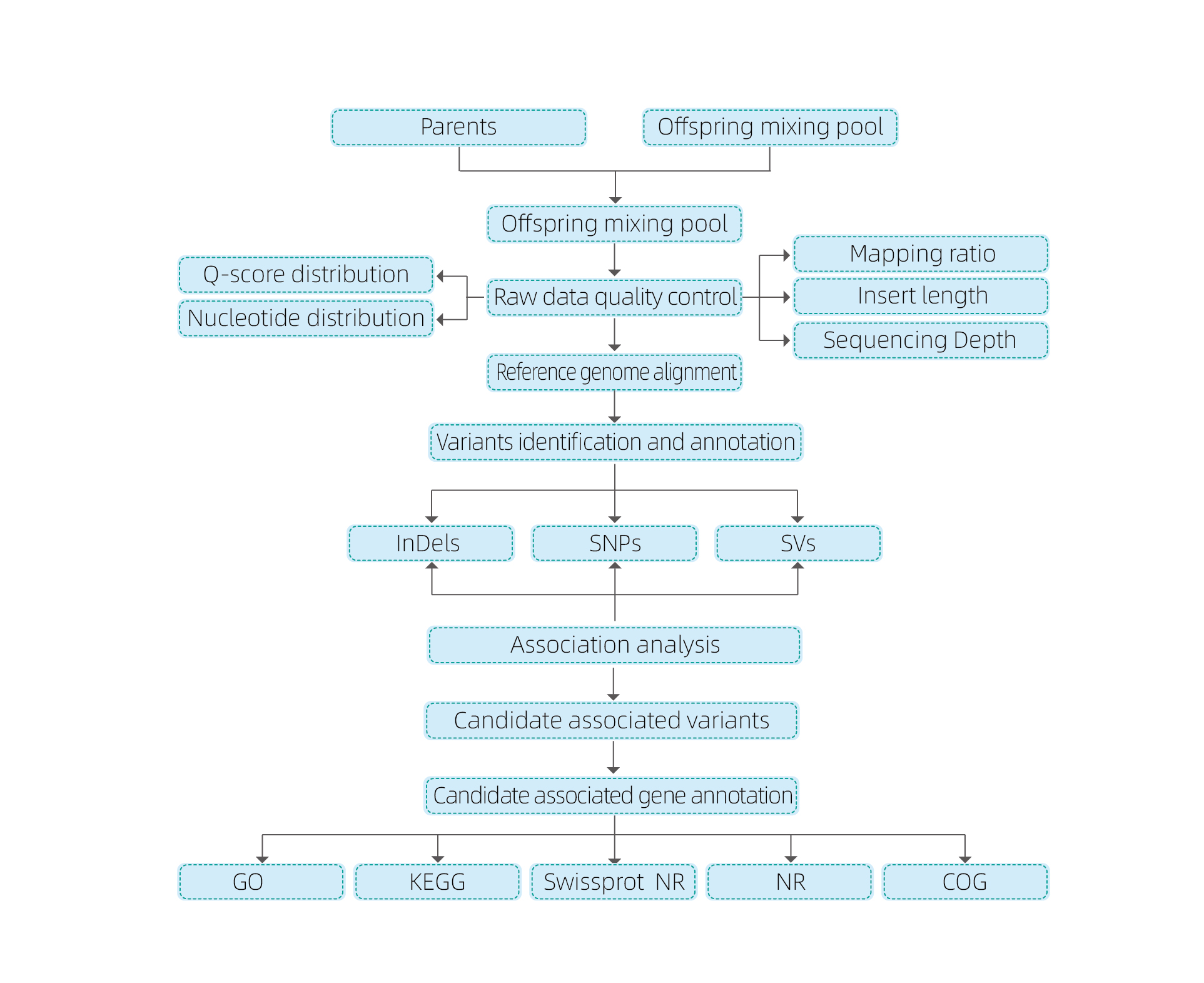
Biinformatatik Analysen
● Ganzt Genome resequem
● Datefinanzéierung
● shp / Dialier ruffen
● bännereverräftungscorverting
● Kandidat VERKAUF FORMATIOUN Annotatioun

Probe Ufuerderunge a Liwwerung
Probe Ufuerderungen:
Cukleogen:
| gdna Probe | Tissue Probe |
| Konzentratioun: ≥30 NG / μl | Planzen: 1-2 g |
| Betrag: ≥2 μg (Volume ≥15 μl) | Déieren: 0,5-1 g |
| Puritéit: OD260 / 280 = 1,6-2,5 | Ganz Blutt: 1,5 ml |
Service Aarbecht fléisst

Experiment Design

Géif Problem Liwwerung

RVA Extraktioun

Bibliothéik Konstruktioun

Sequencing

Daten Analyse

No-Verkaaf Servicer
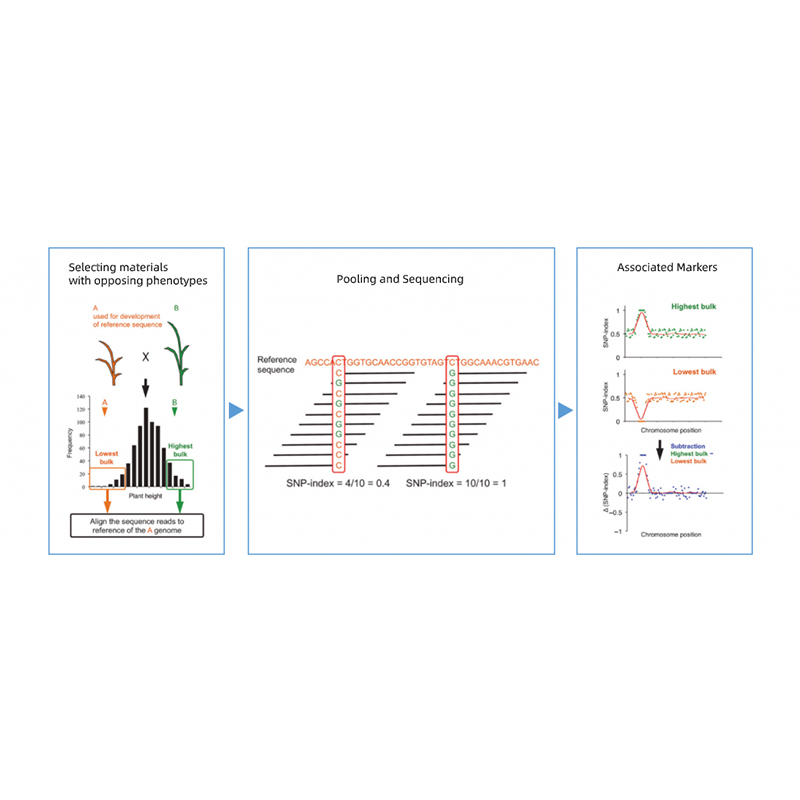
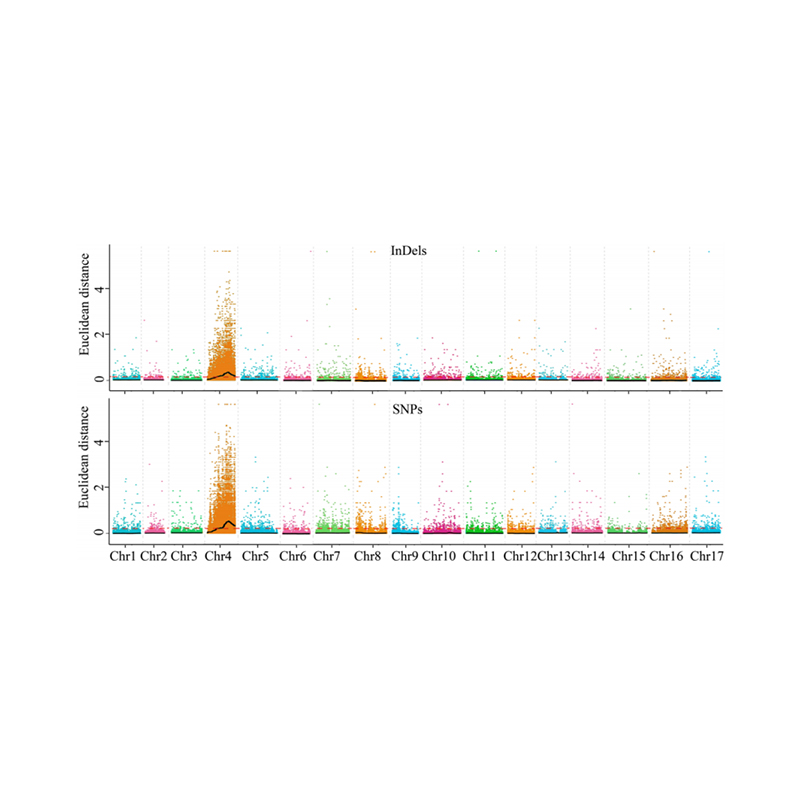
1.association Analyse Base op Euclidean Distanz (ed) fir d'Kandidat Regioun ze identifizéieren. An der folgender Figur
X-Achs: Chromosome Nummer; All Punkt representéiert en ed Wäert vun engem snp. Déi schwaarz Line entsprécht dem ed Wäert. E méi héije edste Wäert weist eng méi bedeitendst Associatioun tëscht dem Site an de Phenotyp. Red Dash Line representéiert Dréiheld vu bedeitende Associatioun.

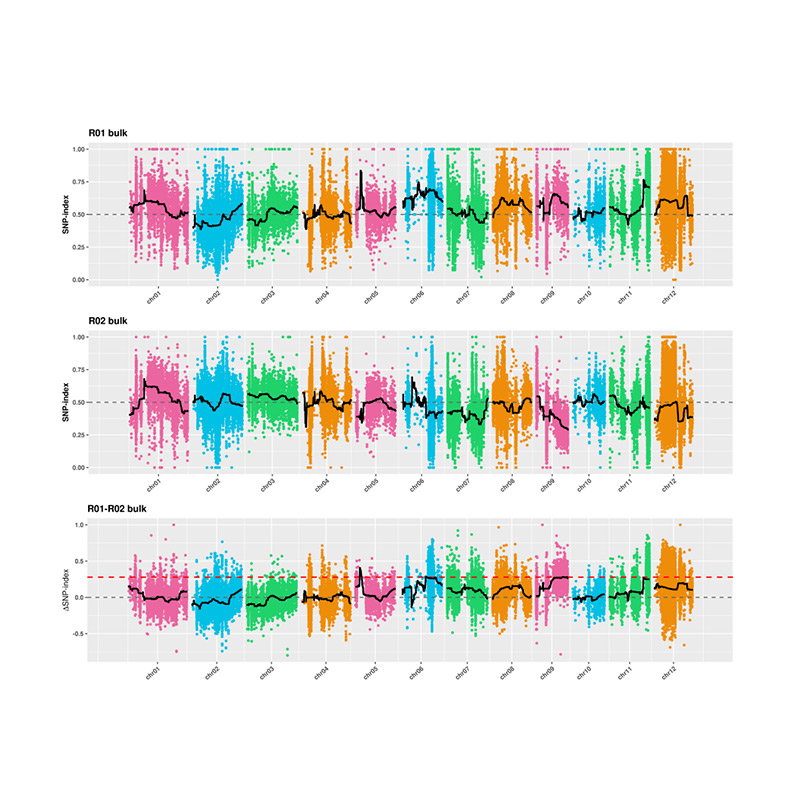
2.association Analyse baséiert kee Snp-Index
X-Achs: Chromosome Nummer; All Punkt representéiert de Snp-Indexwäert. Déi schwaarz Linn steet fir gepasst Snp-Indexwäert. De Wäert huet de Wäert dat méi bedeitend Sënn behalen.

BMK Fall
Den Haapteffekt quantitativen Trait locus fnl7.1
Verëffentlecht: Planzebiotechnologie Journal, 2020
Sequencing Strategie:
Elteren (Jin5-508, YN): ganz Genomen resequeming fir 34 × an 20 ×.
DNA Pools (50 laang-nack an 50 Short-Nack): Resizencing fir 61 × an 52 ×
Schlësselresultater
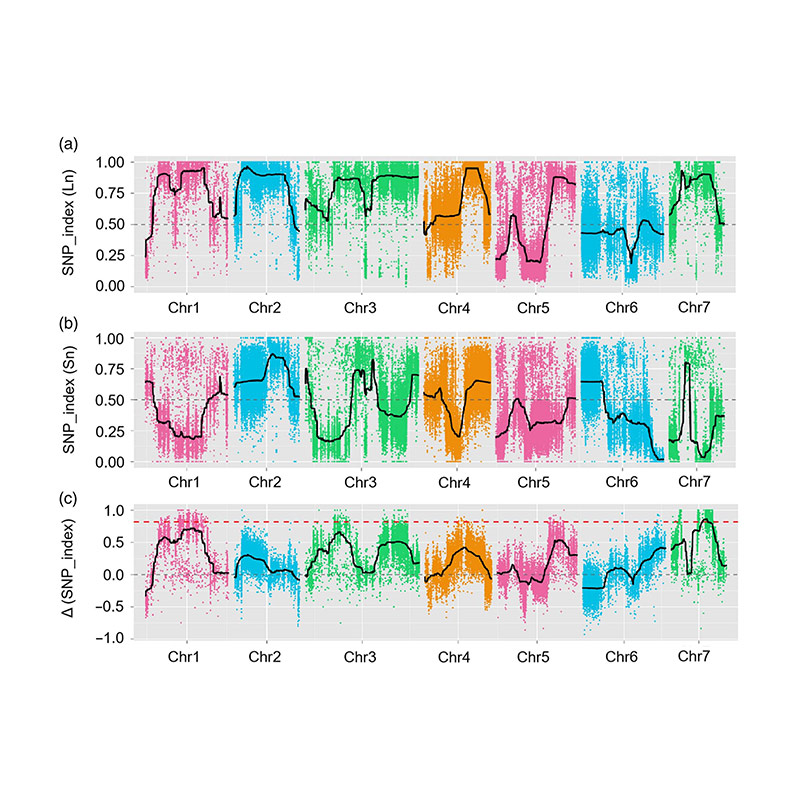
An dëser Etude, segregéiert Populatioun (F2 an F2: 3) gouf generéiert duerch laang-Hals Gurke Line Line Jin5-508 a Short-Hals Yn. Zwee DNA Poole goufe vu 50 extrem laang-Hals Individuen a 50 extrem Kuerzmaäich gebaut. Major-Effekt QTL gouf op de Chr07 duerch BSA Analyse an traditionell qtl Kartéierung identifizéiert. De Kandidat Regioun war weider vu Fine-Mapping, Gen Ausdrock-Quantitéit an iwwerwaachtent Experimenter, ginn de CSFNNNNNNUL -1,1. Zousätzlech, Polymorphismus am CSFNLLU7.1 Promoteur ass mat entspriechende Ausdrock fonnt ginn. Weider phylogenetesch Analyse huet sech virgeschloen datt fnl7.1 Locus ganz wahrscheinlech aus Indien hierkënnt.
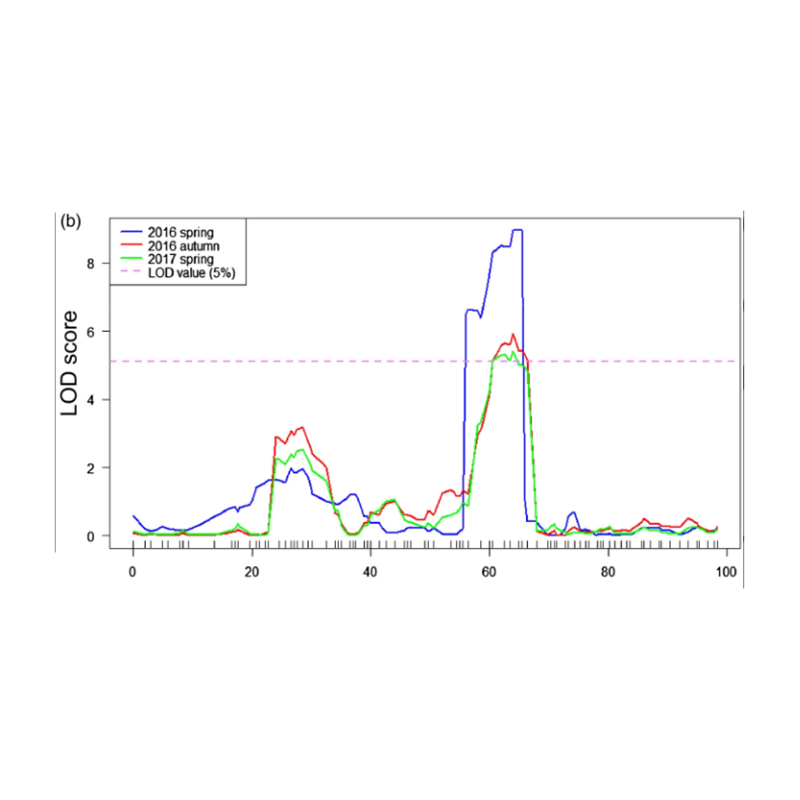
 QTL-Mapping an der BSA Analyse fir d'Kandidat Regioun ze identifizéieren ass mat Gurken Halslängt ze identifizéieren |  Lod Profiler vu Gurken Hals-Längt QTL identifizéiert um Chr07 |
Xu, x., et al. "De Majoreffekt quantitativ Trait locus fnl7.1 Iwwer e spéiden Embryogenesis Iwwerflossesch Protein assoziéiert mat Uruffer Hals Längt am Gurken." Planzebiotechnologie Journal 18.7 (2020).