게놈 전체 연관 분석
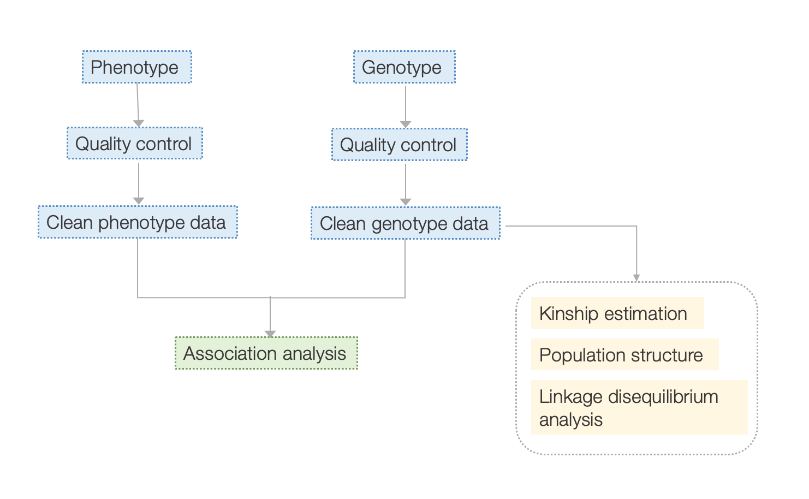
워크 플로

서비스 장점
●광범위한 전문 지식 및 출판 기록: GWAS에 대한 경험이 축적 된 Bmkgene은 인구 GWAS 연구에서 수백 개의 종 프로젝트를 완료했으며, 연구원들이 100 개가 넘는 기사를 출판하도록 지원했으며 누적 영향 요인은 500에 도달했습니다.
● 포괄적 인 생물 정보학 분석: 워크 플로에는 SNP-Trait 연관 분석이 포함되어 있으며, 후보 유전자 세트 및 해당 기능 주석을 전달합니다.
●고도로 숙련 된 생물 정보학 팀 및 짧은 분석주기: Advanced Genomics Analysis에서 훌륭한 경험을 통해 BMKGENE의 팀은 신속한 처리 시간으로 포괄적 인 분석을 제공합니다.
●판매 후 지원 :우리의 약속은 프로젝트 완료를 넘어 3 개월간의 사후 서비스 기간으로 확장됩니다. 이 기간 동안 우리는 프로젝트 후속 조치, 문제 해결 지원 및 Q & A 세션을 제공하여 결과와 관련된 쿼리를 해결합니다.
서비스 사양 및 요구 사항
| 시퀀싱 유형 | 권장 인구 규모 | 시퀀싱 전략 | 뉴클레오티드 요구 사항 |
| 전체 게놈 시퀀싱 | 200 샘플 | 10x | 농도 : ≥ 1 ng/ µl 총액 30ng 저하 또는 오염이 제한적이거나 없음 |
| 특정 로커스 증폭 된 조각 (SLAF) | 태그 깊이 : 10x 태그 수 : <400 MB : WGS가 권장됩니다 <1GB : 100K 태그 1GB > 2GB : 300K 태그 최대 500k 태그 | 농도 ≥ 5 ng/µl 총 금액 ≥ 80 ng Nanodrop OD260/280 = 1.6-2.5 아가 로스 겔 : 분해 또는 오염이 제한적이거나 제한적입니다
|
재료 선택



다양한 품종, 아종, 랜드 레이스/유전자 뱅크/혼합 가족/야생 자원
다양한 품종, 아종, 랜드 레이스
하프 시브 가족/풀 시브 가족/야생 자원
서비스 작업 흐름

실험 설계

샘플 전달

RNA 추출

도서관 건설

시퀀싱

데이터 분석

애프터 판매 서비스
다음 분석을 포함합니다.
- 게놈 전체 연관 분석 : LM, LMM, Emmax, Fastlmm 모델
- 후보 유전자의 기능적 주석
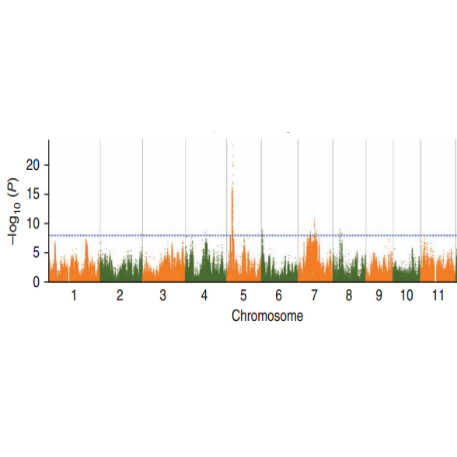
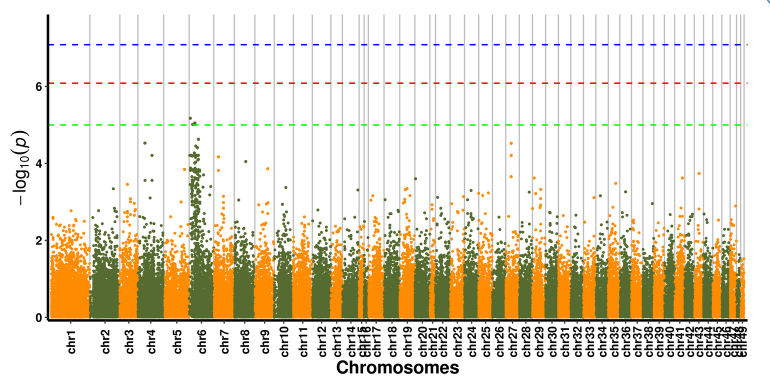
SNP-TRAIT Association Analysis-맨해튼 플롯
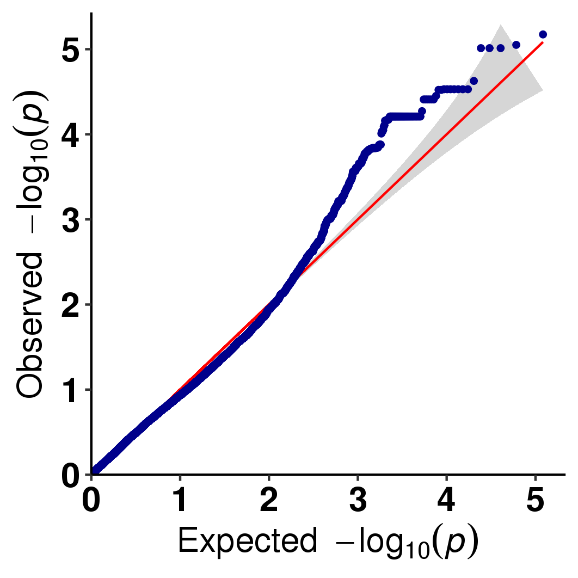
SNP-trait 연관 분석-QQ 플롯
선별 된 출판물 모음을 통해 BMKGENE의 DE GWAS 서비스가 촉진하는 진보를 탐색하십시오.
LV, L. et al. (2023) '게놈 전체 연관 연구에 의한 면도기 조개 sinonovacula complericta에서 암모니아 내성의 유전 적 기초에 대한 통찰력',양식, 569, p. 739351. doi : 10.1016/j.aquaculture.2023.739351.
Li, X. et al. (2022) '398 개의 Fextail Millet Accession의 다중 생물 분석은 가축화, 대사 산물 특성 및 항 염증 효과와 관련된 게놈 영역을 나타냅니다.',분자 식물, 15 (8), pp. 1367–1383. doi : 10.1016/j.molp.2022.07.003.
Li, J. et al. (2022) '가뭄 환경에서 헐리스의 게놈 전체 연관성 매핑',식물 과학의 국경, 13, p. 924892. doi : 10.3389/fpls.2022.924892/bibtex.
Zhao, X. et al. (2021) '설포 트랜스퍼 라제를 암호화하는 GMST1은 대두 모자이크 바이러스 균주 G2 및 G3'에 대한 내성을 부여합니다.식물, 세포 및 환경, 44 (8), pp. 2777–2792. doi : 10.1111/pce.14066.