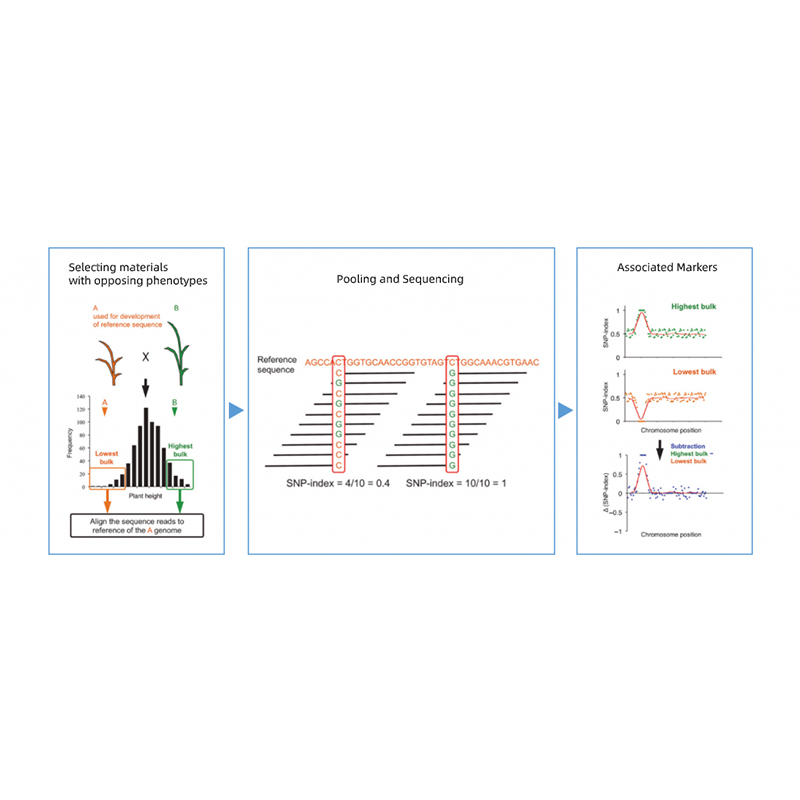
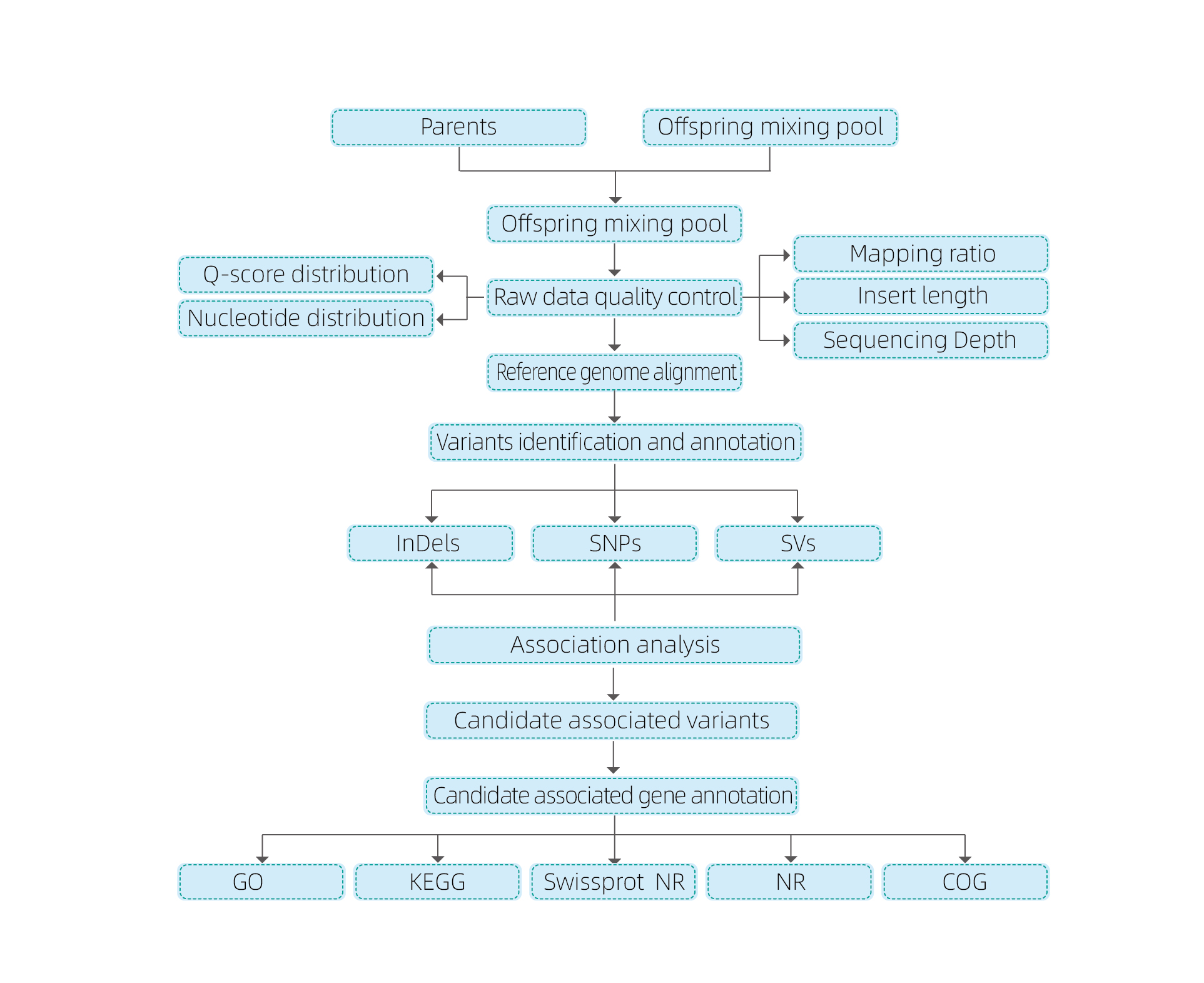
대량 분리 분석
서비스 장점

Takagi et al., The Plant Journal, 2013
● 정확한 현지화 : 배경 소음을 최소화하기 위해 벌크가 30+30 ~ 200+200 인 개인과 혼합; 비 동의 돌연변이 체 기반 후보 영역 예측.
● 포괄적 인 분석 : NR, Swissprot, Go, Kegg, Cog, Kog 등을 포함한 심층적 인 후보 유전자 기능 주석.
● 더 빠른 처리 시간 : 근무일 기준 45 일 이내에 빠른 유전자 국소화.
● 광범위한 경험 : BMK는 작물, 수생 제품, 숲, 꽃, 과일 등과 같은 다양한 종을 다루는 수천 가지 특성 지역화에 기여했습니다.
서비스 사양
인구:
반대 표현형을 가진 부모의 자손을 분리합니다.
EG F2 자손, 백 크로스 (BC), 재조합 근친 교배소 (RIL)
믹싱 풀
질적 특성 : 30 ~ 50 명의 개인 (최소 20)/벌크
정량적 tratis : 전체 인구에서 극도의 표현형을 가진 상위 5% ~ 10% 개인 (최소 30+30).
권장 시퀀싱 깊이
최소 20x/parent 및 1x/offspring 개인 (예 : 30+30 개별의 자손 혼합 풀의 경우 시퀀싱 깊이는 벌크 당 30 배입니다)
생물 정보학 분석
● 전체 게놈 재현
● 데이터 처리
● SNP/indel 전화
● 후보 지역 선별
● 후보 유전자 기능 주석

샘플 요구 사항 및 전달
샘플 요구 사항 :
뉴클레오티드 :
| GDNA 샘플 | 조직 샘플 |
| 농도 : ≥30 ng/μl | 식물 : 1-2 g |
| 양 : ≥2 μg (Volumn ≥15 μl) | 동물 : 0.5-1 g |
| 순도 : OD260/280 = 1.6-2.5 | 전혈 : 1.5 ml |
서비스 작업 흐름

실험 설계

샘플 전달

RNA 추출

도서관 건설

시퀀싱

데이터 분석

애프터 판매 서비스
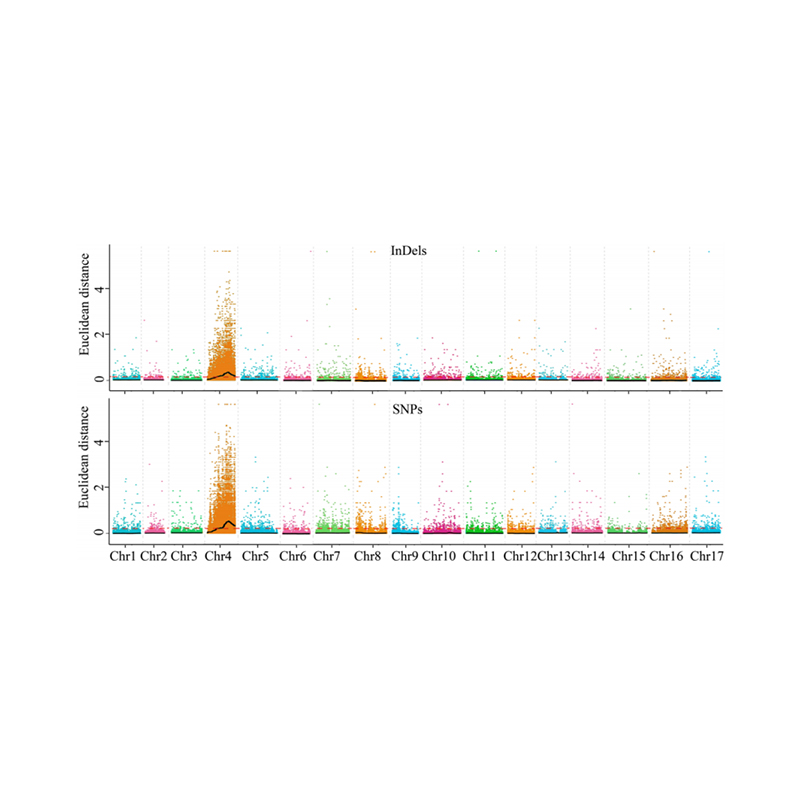
1. 후보 영역을 식별하기위한 유클리드 거리 (ED)의 협회 분석 기반. 다음 그림에서
X 축 : 염색체 수; 각 점은 SNP의 에드 값을 나타냅니다. 블랙 라인은 적합한 ed 값에 해당합니다. 더 높은 ED 값은 사이트와 표현형 사이의 더 중요한 연관성을 나타냅니다. 빨간 대시 라인은 유의미한 연관의 임계 값을 나타냅니다.

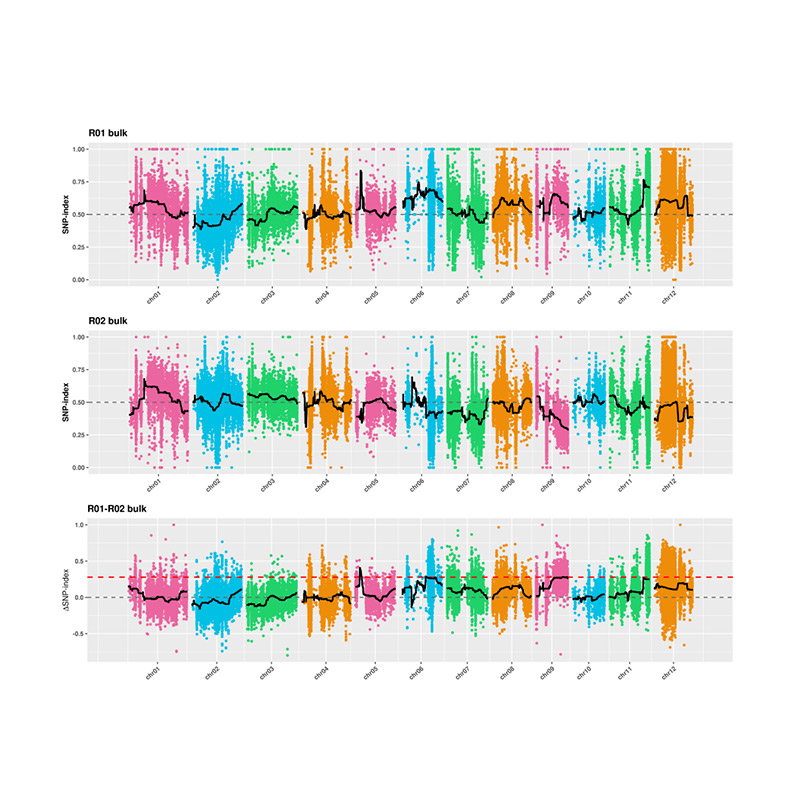
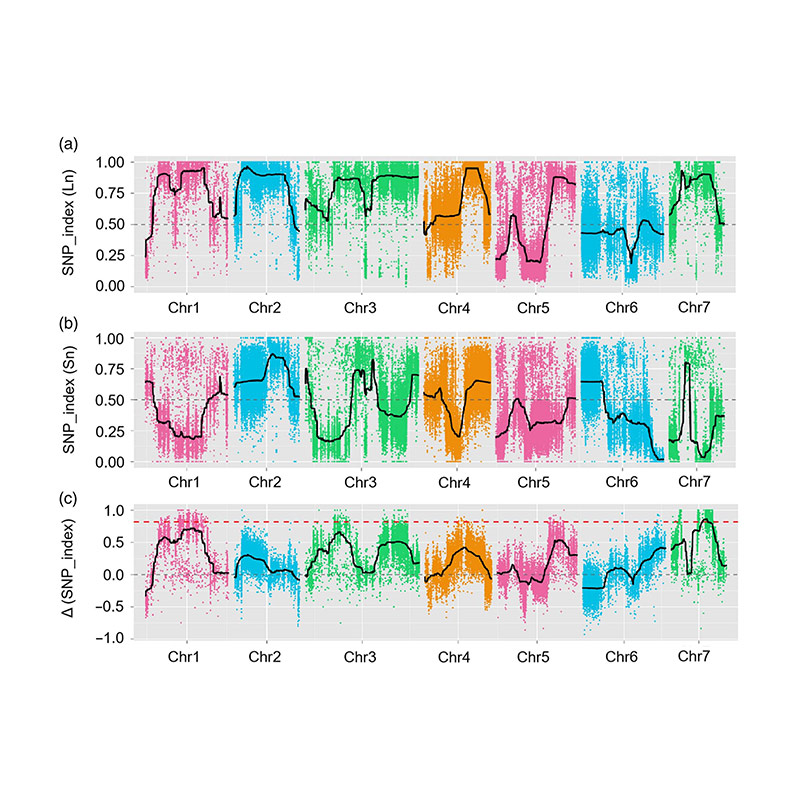
2. SNP-Index를 기반으로 연관성 분석
X 축 : 염색체 수; 각 점은 SNP-Index 값을 나타냅니다. 블랙 라인은 적합한 SNP-Index 값을 나타냅니다. 가치가 클수록 협회가 더 중요합니다.

BMK 케이스
주요 효과 정량적 특성 유전자좌 FNL7.1은 오이의 과일 목 길이와 관련된 늦은 배아 생성 풍부 단백질을 암호화합니다.
게시 : Plant Biotechnology Journal, 2020
시퀀싱 전략 :
부모 (JIN5-508, YN) : 34 × 및 20 ×에 대한 전체 게놈 재판매.
DNA 풀 (50 개의 장거리 및 50 개의 짧은 목) : 61x 및 52 ×에 대한 재조정
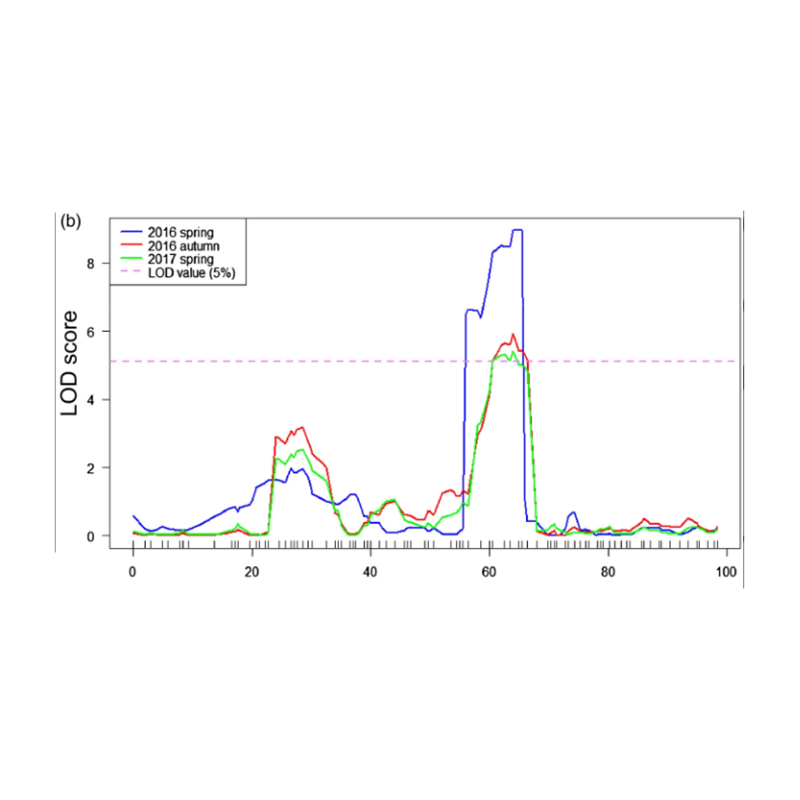
주요 결과
이 연구에서, 장거리 오이 라인 JIN5-508 및 단락 YN을 가로 질러 분리 된 모집단 (F2 및 F2 : 3)이 생성되었습니다. 2 개의 DNA 풀은 50 명의 극한 장거리 자 개체와 50 명의 극한 단락 개인에 의해 구성되었습니다. 주요 효과 QTL은 BSA 분석 및 전통적인 QTL 매핑에 의해 Chr07에서 확인되었다. 후보 영역은 미세 맵핑, 유전자 발현 정량화 및 형질 전환 실험에 의해 더욱 좁아졌으며, 이는 목 길이, CSFNL7.1을 제어하는 주요 유전자를 밝혀냈다. 또한, CSFNL7.1 프로모터 영역에서의 다형성은 상응하는 발현과 관련이있는 것으로 밝혀졌다. 추가 계통 발생 학적 분석은 FNL7.1 유전자좌가 인도에서 유래 될 가능성이 높다고 제안했다.
 오이 목 길이와 관련된 후보 영역을 식별하기위한 BSA 분석에서의 QTL 맵핑 |  CHR07에서 확인 된 오이 목 길 QTL의 LOD 프로파일 |
Xu, X., et al. "주요 효과 정량적 특성 유전자 좌위 FNL7.1은 오이의 과일 목 길이와 관련된 늦은 배아 생성 풍부 단백질을 암호화합니다." Plant Biotechnology Journal 18.7 (2020).