BMKMANU S1000 Transcriptome מרחבי
BMKMANU S1000 תכנית תעתיק מרחבית טכנית
תכונות
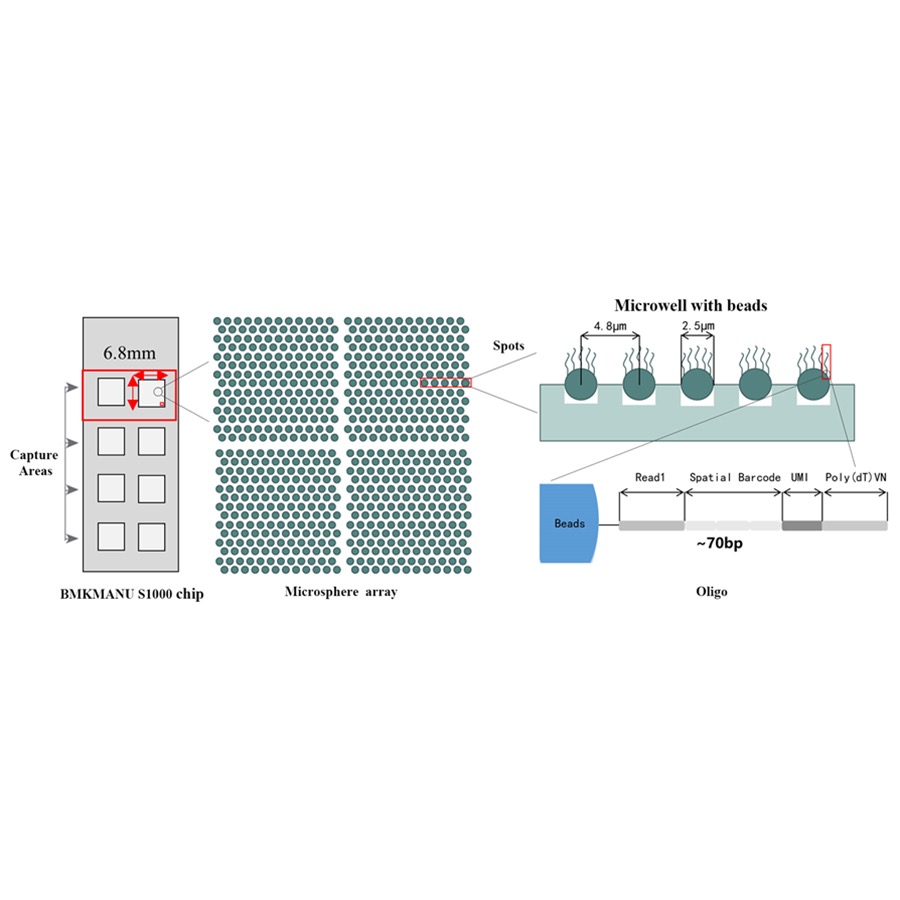
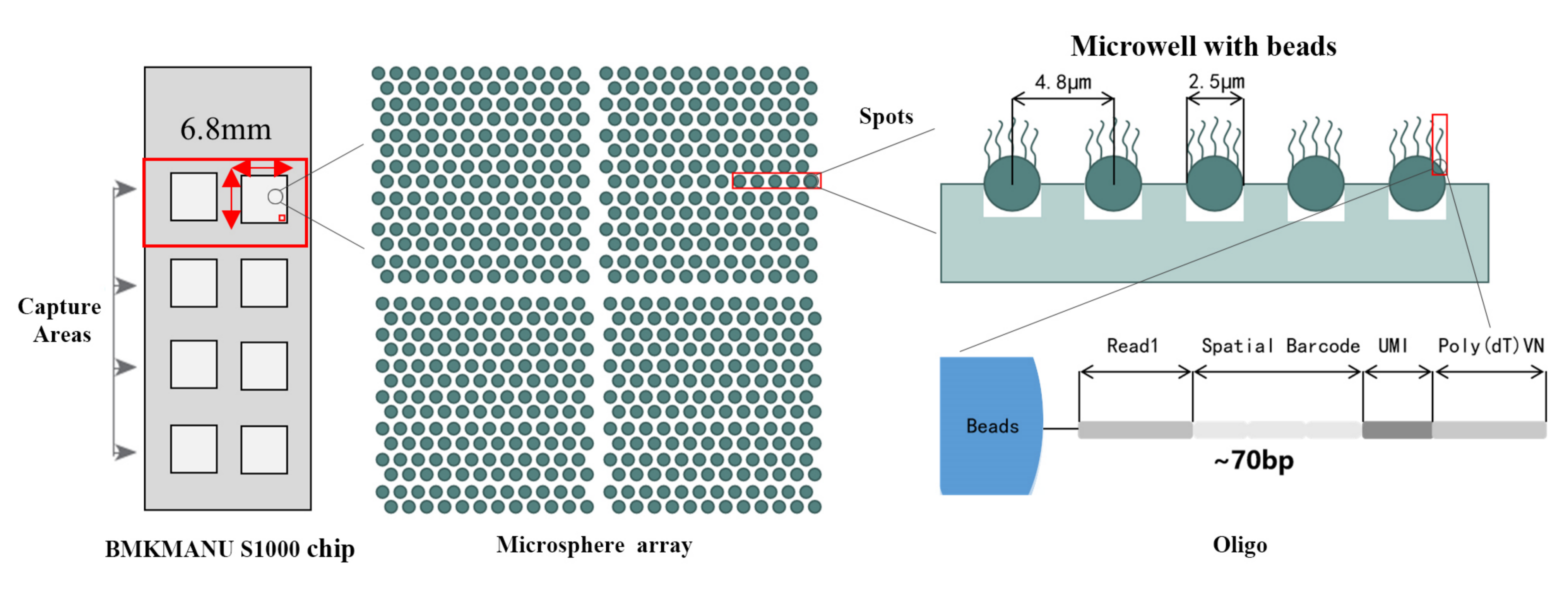
● רזולוציה: 5 מיקרומטר
● קוטר ספוט: 2.5 מיקרומטר
● מספר נקודות: כ-2 מיליון
● 3 פורמטים אפשריים של אזור לכידה: 6.8 מ"מ * 6.8 מ"מ, 11 מ"מ * 11 מ"מ או 15 מ"מ * 20 מ"מ
● כל חרוז ברקוד טעון בפריימרים המורכבים מ-4 חלקים:
זנב poly(dT) ל-mRNA priming וסינתזת cDNA
מזהה מולקולרי ייחודי (UMI) לתיקון הטיית הגברה
ברקוד מרחבי
רצף מחייב של פריימר רצף חלקי לקרוא 1
● H&E וצביעה ניאון של חתכים
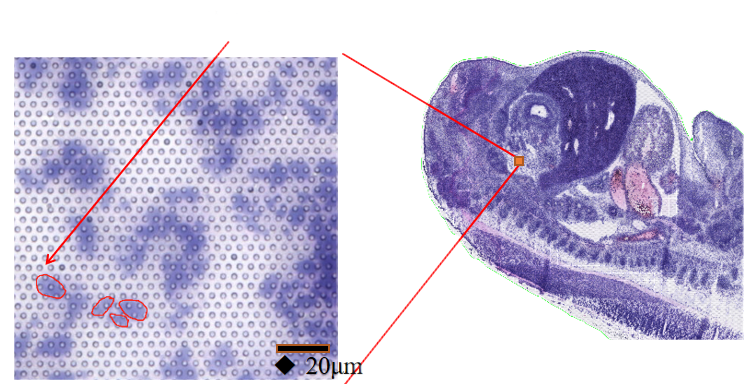
● אפשרות לשימושטכנולוגיית פילוח תאים: אינטגרציה של צביעת H&E, צביעה פלואורסצנטית ורצף RNA כדי לקבוע את הגבולות של כל תא והקצאה נכונה של ביטוי גנים לכל תא.
היתרונות של BMKMANU S1000
●רזולוציה תת-סלולרית: כל אזור לכידה הכיל יותר מ-2 מיליון נקודות ברקוד מרחביות בקוטר של 2.5 מיקרומטר ומרווח של 5 מיקרומטר בין מרכזי הנקודה, מה שמאפשר ניתוח תעתיק מרחבי ברזולוציה תת-תאית (5 מיקרומטר).
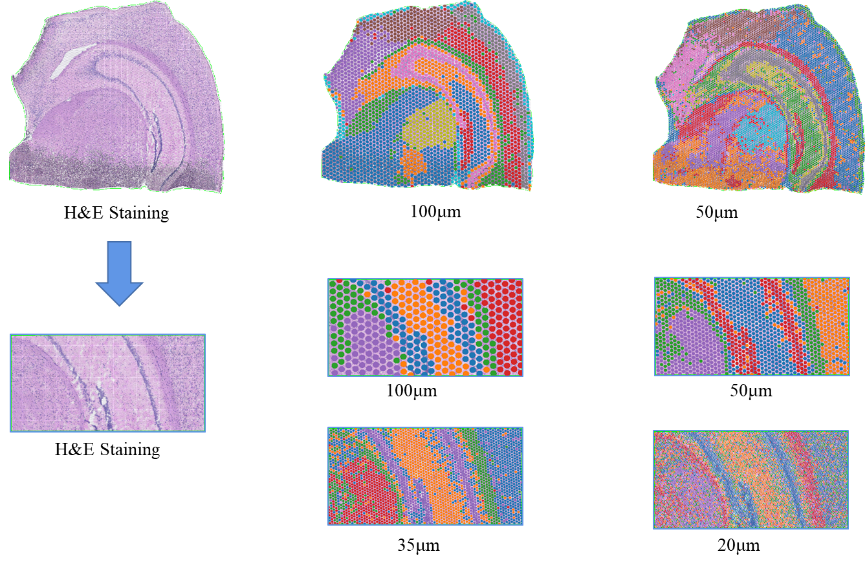
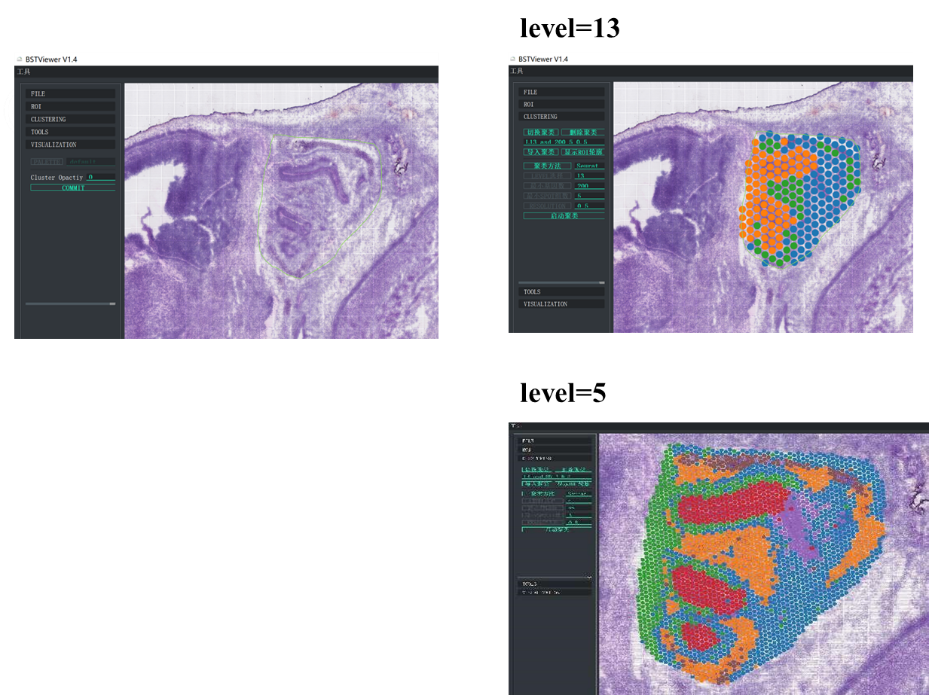
●ניתוח רזולוציה מרובת רמות:ניתוח רב-שכבתי גמיש הנעים בין 100 מיקרומטר ל-5 מיקרומטר כדי לפתור תכונות רקמה מגוונות ברזולוציה אופטימלית.
●אפשרות להשתמש בטכנולוגיית פילוח תאים "שלוש בשקופית אחת":שילוב של צביעת פלואורסצנטי, צביעת H&E ורצף RNA בשקופית אחת, אלגוריתם הניתוח "שלושה-ב-אחד" שלנו מעצים את הזיהוי של גבולות תאים עבור תעתיק מבוססי תאים הבאים.
●תואם לפלטפורמות רצף מרובות: גם NGS וגם רצף לקריאה ארוכה זמינים.
●עיצוב גמיש של 1-8 אזור לכידה פעילה: גודל אזור הלכידה גמיש, וניתן להשתמש ב-3 פורמטים (6.8 מ"מ * 6.8 מ"מ, 11 מ"מ * 11 מ"מ ו-15 מ"מ * 20 מ"מ)
●שירות חד פעמי: הוא משלב את כל השלבים המבוססים על ניסיון ומיומנויות, כולל ניתוח קריו, צביעה, אופטימיזציה של רקמות, ברקוד מרחבי, הכנת ספרייה, רצף וביואינפורמטיקה.
●ביואינפורמטיקה מקיפה והדמיה ידידותית למשתמש של תוצאות:החבילה כוללת 29 ניתוחים ו-100+ דמויות באיכות גבוהה, בשילוב עם שימוש בתוכנה שפותחה בעצמה כדי להמחיש ולהתאים אישית את פיצול התאים וצבירת נקודות.
●ניתוח נתונים ויזואליזציה מותאמים אישית: זמין עבור בקשות מחקר שונות
●צוות טכני מיומן במיוחד: עם ניסיון בלמעלה מ-250 סוגי רקמות ו-100+ מינים כולל אדם, עכבר, יונק, דגים וצמחים.
●עדכונים בזמן אמת על כל הפרויקט: עם שליטה מלאה בהתקדמות הניסוי.
●ניתוח משותף אופציונלי עם רצף mRNA חד-תא
מפרט שירות
|
לִטעוֹם דרישות
| סִפְרִיָה |
אסטרטגיית רצף
| נתונים מומלצים | בקרת איכות |
| דגימות קריו משובצות ב-OCT, 3 בלוקים לדגימה | ספריית cDNA S1000 | Illumina PE150 (פלטפורמות אחרות זמינות) | 100K PE קריאות לכל 100 uM (60-150 ג'יגה-בייט) | RIN>7 |
לפרטים נוספים על הדרכה לדוגמא וזרימת עבודה של שירות, אל תהסס לדבר עם אמומחה BMKGENE
זרימת עבודה בשירות
בשלב הכנת הדגימה, מבוצע ניסוי מיצוי RNA בתפזורת ראשונית כדי להבטיח שניתן להשיג RNA באיכות גבוהה. בשלב אופטימיזציית הרקמות החתכים מוכתמים ומומחזים ותנאי החדירה לשחרור mRNA מהרקמה עוברים אופטימיזציה. לאחר מכן, הפרוטוקול האופטימלי מיושם במהלך בניית הספרייה, ואחריו רצף וניתוח נתונים.
זרימת העבודה המלאה של השירות כוללת עדכונים בזמן אמת ואישורי לקוחות כדי לשמור על לולאת משוב תגובה, המבטיחה ביצוע חלק של הפרויקט.
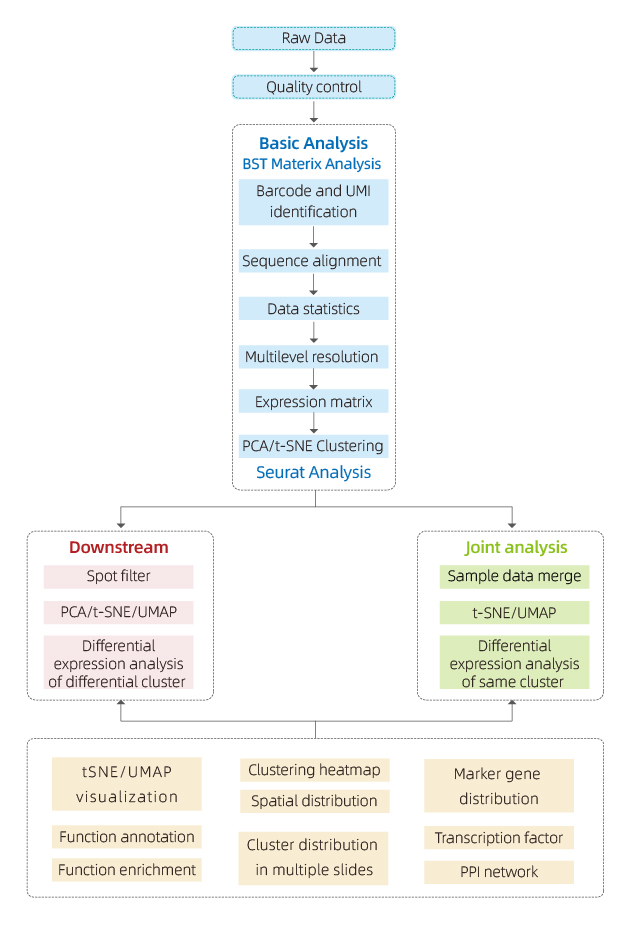
הנתונים שנוצרו על ידי BMKMANU S1000 מנותחים באמצעות התוכנה "BSTMatrix", אשר תוכננה באופן עצמאי על ידי BMKGENE, ויוצרת מטריקס של ביטוי גנים. משם, מופק דוח סטנדרטי הכולל בקרת איכות נתונים, ניתוח מדגם פנימי וניתוח בין קבוצות.
● בקרת איכות נתונים:
פלט נתונים וחלוקת ציוני איכות
זיהוי גנים לכל נקודה
כיסוי רקמות
● ניתוח מדגם פנימי:
עושר גנים
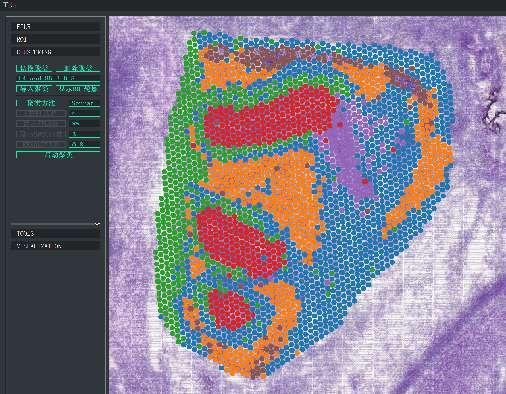
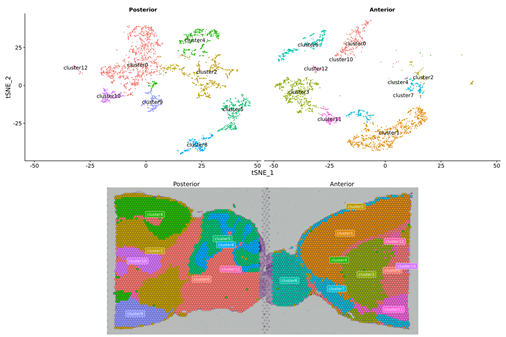
צבירת נקודות, כולל ניתוח מימדים מופחת
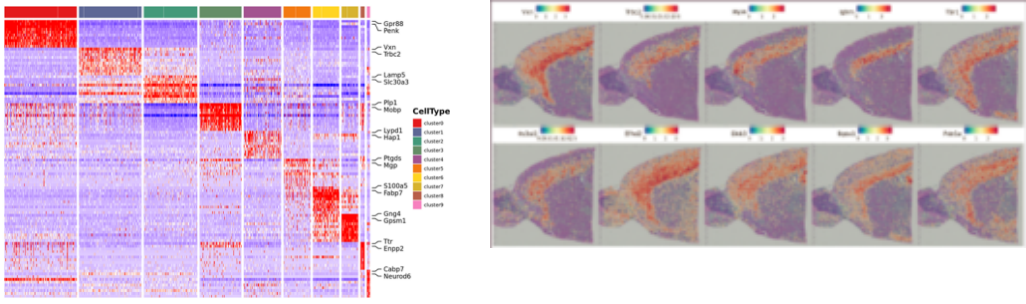
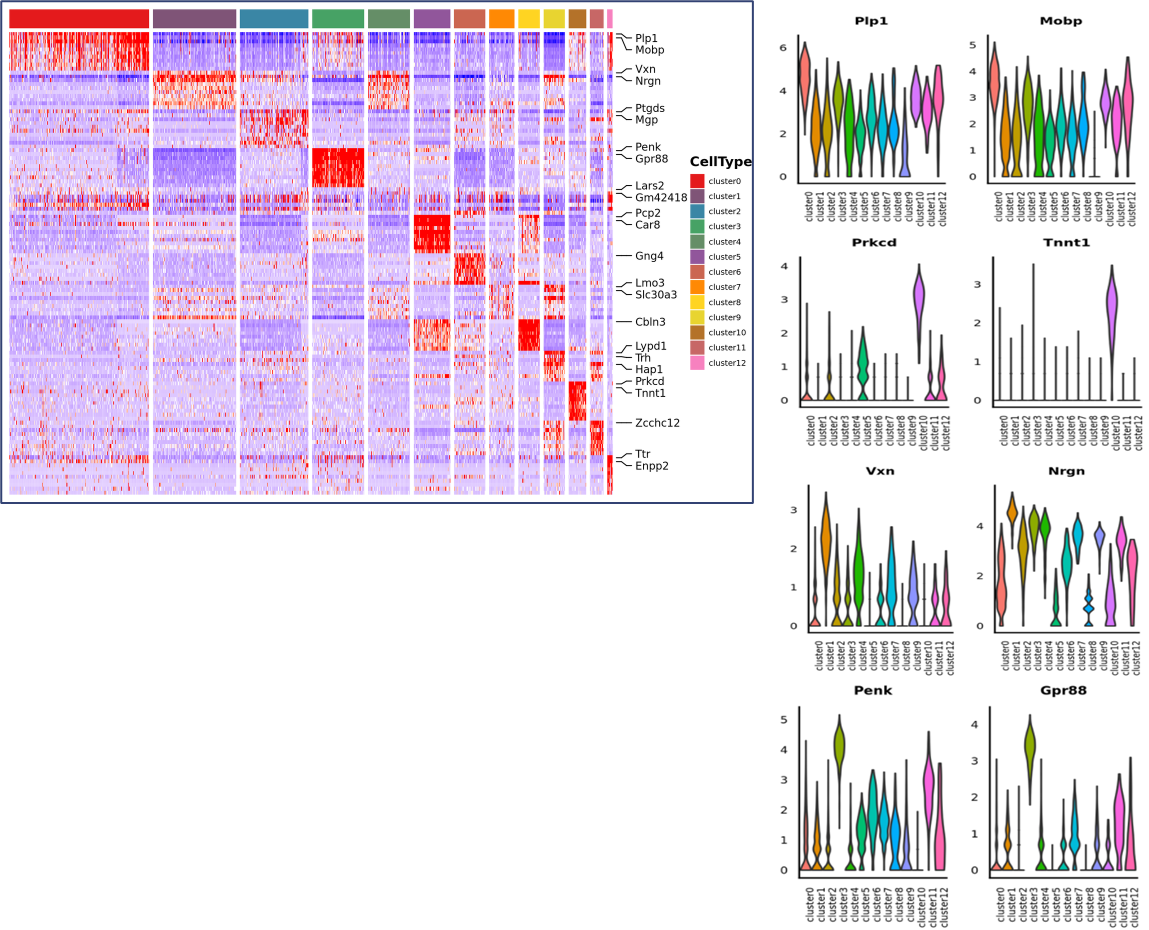
ניתוח ביטוי דיפרנציאלי בין אשכולות: זיהוי גנים של סמן
ביאור פונקציונלי והעשרה של גנים סמנים
● ניתוח בין קבוצות:
שילוב מחדש של כתמים משתי הדגימות (למשל חולים ובקרה) ומקבץ מחדש
זיהוי גני סמן עבור כל אשכול
ביאור פונקציונלי והעשרה של גנים סמנים
ביטוי דיפרנציאלי של אותו אשכול בין קבוצות
בנוסף, BMKGENE שפותחה "BSTViewer" הוא כלי ידידותי למשתמש המאפשר למשתמש לדמיין את ביטוי הגנים ואת התקבצות נקודות ברזולוציות שונות.
BMKGene פיתחה תוכנה להדמיה ידידותית למשתמש
צבירת נקודות BSTViewer ברזולוציה מרובת רמות
BSTcellViewer: פיצול תאים אוטומטי וידני
ניתוח מדגם פנימי
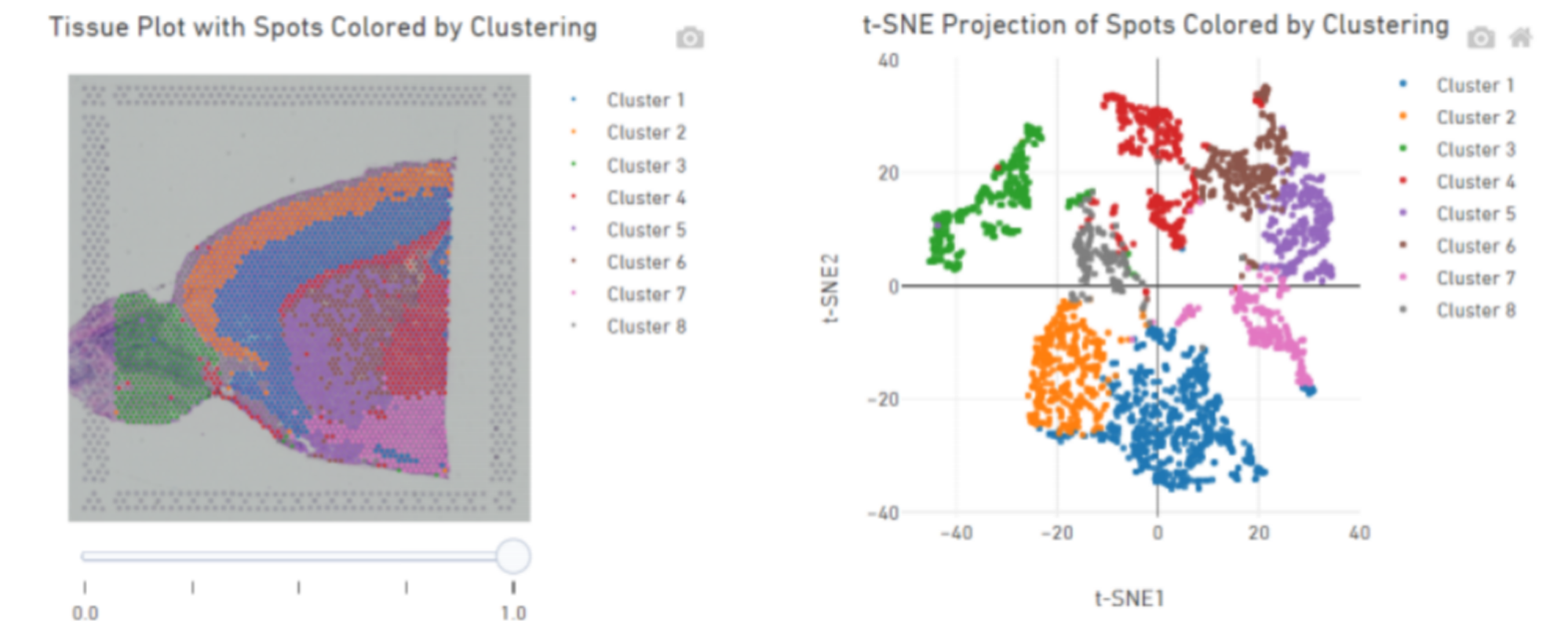
מקבץ נקודות:
זיהוי גני סמן והתפלגות מרחבית:
ניתוח בין קבוצות
שילוב נתונים משתי הקבוצות ומקבץ מחדש:
גנים סמן של אשכולות חדשים:
חקור את ההתקדמות שמאפשרת שירותי התעתיק המרחבי של BMKGene עם טכנולוגיית BMKManu S1000 בפרסום מוצג זה:
שיר, X. et al. (2023) "תעתוק מרחבי חושפת תאי כלורנכימה המושרים על ידי אור המעורבים בקידום התחדשות יורה בקאלוס של עגבניות",הליכים של האקדמיה הלאומית למדעים של ארצות הברית של אמריקה, 120(38), עמ'. e2310163120. doi: 10.1073/pnas.2310163120
אתה, י' ועוד. (2023) 'השוואה שיטתית של שיטות תעתיק מרחביות מבוססות רצף',bioRxiv, עמ' . 2023.12.03.569744. doi: 10.1101/2023.12.03.569744.