Sequenziamento del trascrittoma intero - Illumina
Caratteristiche
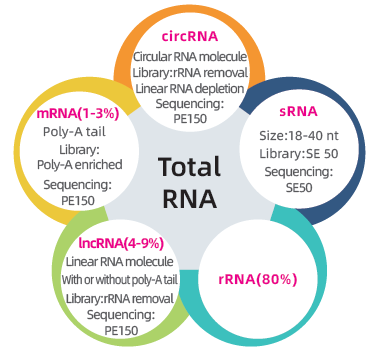
● Doppia libreria per sequenziare il trascrittoma completo: esaurimento dell'RRNA seguito dalla preparazione della libreria PE150 e dalla selezione delle dimensioni seguita dalla preparazione della libreria SE50
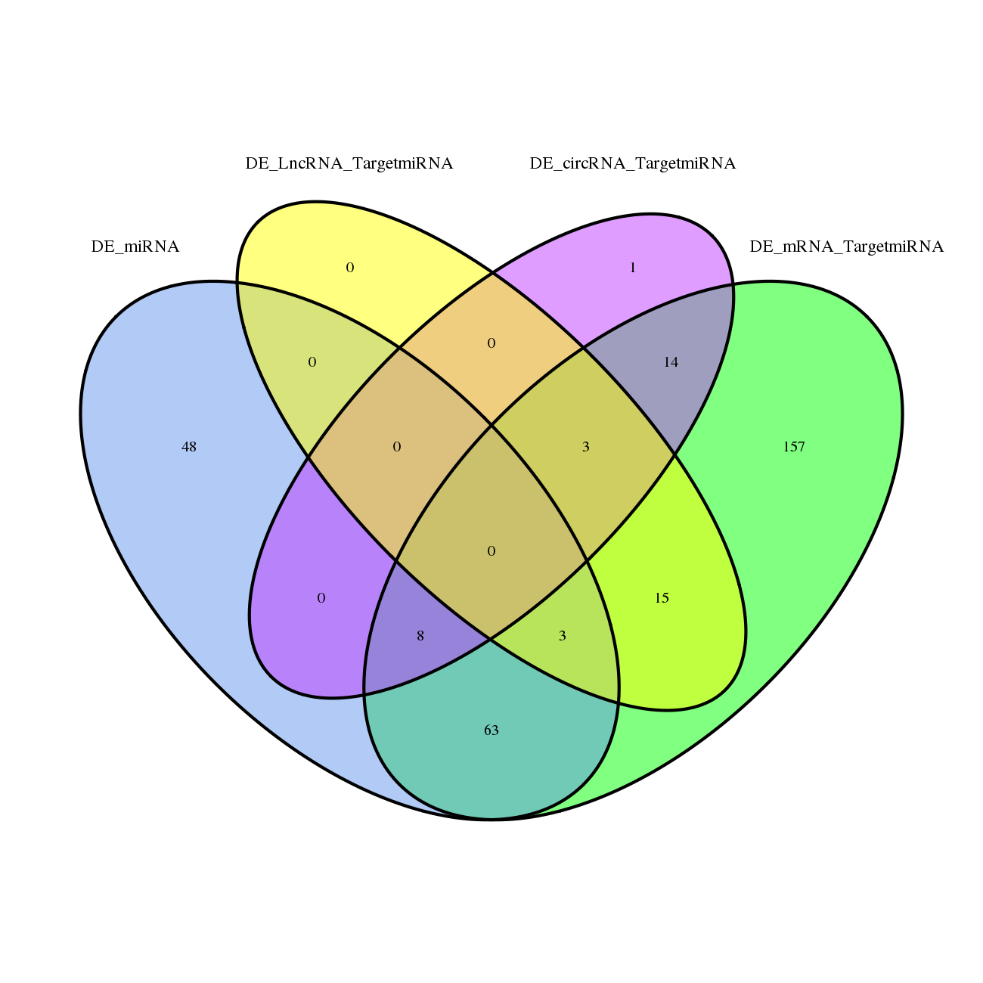
● Analisi bioinformatica completa di mRNA, lncRNA, circrNA e miRNA in report bioinformatici separati
● Analisi congiunta di tutta l'espressione dell'RNA in un rapporto combinato, inclusa l'analisi delle reti CERNA.
Vantaggi del servizio
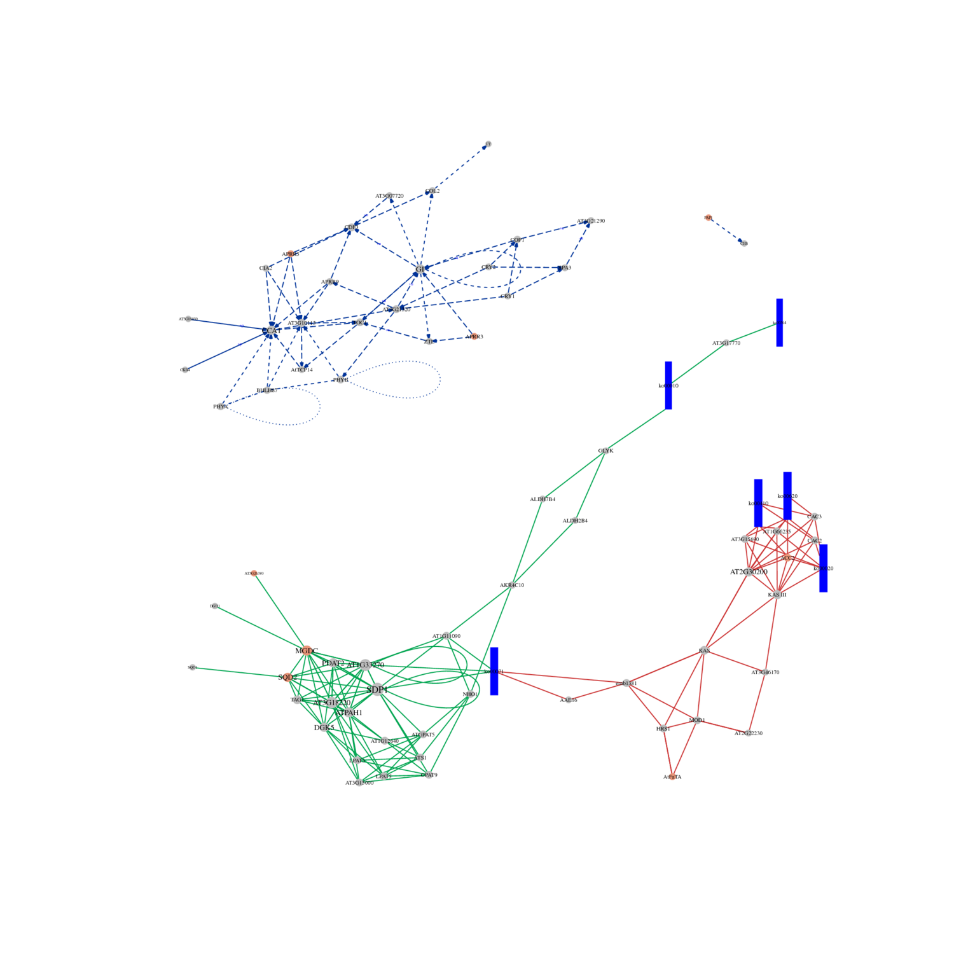
●Analisi approfondita delle reti regolatori: L'analisi della rete CERNA è abilitata dal sequenziamento articolare di mRNA, lncRNA, circrNA e miRNA e da un flusso di lavoro bioinformatico esaustivo.
●Annotazione completa: Utilizziamo più database per annotare funzionalmente i geni espressi in modo differenziato (DEG) ed eseguire l'analisi di arricchimento corrispondente, fornendo approfondimenti sui processi cellulari e molecolari alla base della risposta del trascrittoma.
●Vasta competenza: Con una storia di chiusura con successo oltre 2100 progetti di trascrittoma intero in vari domini di ricerca, il nostro team porta una vasta esperienza in ogni progetto.
●Controllo di qualità rigoroso: Implementiamo i punti di controllo di base in tutte le fasi, dalla preparazione di campioni e libreria al sequenziamento e alla bioinformatica. Questo meticoloso monitoraggio garantisce la consegna di risultati costantemente di alta qualità.
●Supporto post-vendita: Il nostro impegno si estende oltre il completamento del progetto con un periodo di servizio post-vendita di 3 mesi. Durante questo periodo, offriamo un follow-up del progetto, assistenza per la risoluzione dei problemi e sessioni di domande e risposte per affrontare eventuali domande relative ai risultati
Requisiti di esempio e consegna
| Biblioteca | Strategia di sequenziamento | Dati consigliati | Controllo di qualità |
| rRNA impoverito | Illumina PE150 | 16 GB | Q30≥85% |
| Dimensione selezionata | Illumina SE50 | 10-20 m letture |
Requisiti del campione:
Nucleotidi:
| CONC. (NG/μL) | Importo (μg) | Purezza | Integrità |
| ≥ 80 | ≥ 1,6 | OD260/280 = 1.7-2.5 OD260/230 = 0,5-2,5 Contaminazione da proteina o DNA limitata o nessuna mostrata su gel. | Rin≥6.0 5,0≥28s/18S≥1.0; Elevazione limitata o non basale |
Consegna del campione consigliata
Contenitore: tubo di centrifuga da 2 ml (foglio di stagno non è raccomandato)
Etichettatura del campione: gruppo+replicare EG A1, A2, A3; B1, B2, B3.
Spedizione:
1. ICE a secco: i campioni devono essere imballati in borse e sepolti in ghiaccio a secco.
2. Tubi rnastable: i campioni di RNA possono essere essiccati nel tubo di stabilizzazione dell'RNA (es. RNastable®) e spediti a temperatura ambiente.
Flusso di lavoro di servizio

Design dell'esperimento

Consegna del campione

Estrazione di RNA

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Servizi post-vendita
Bioinformatica
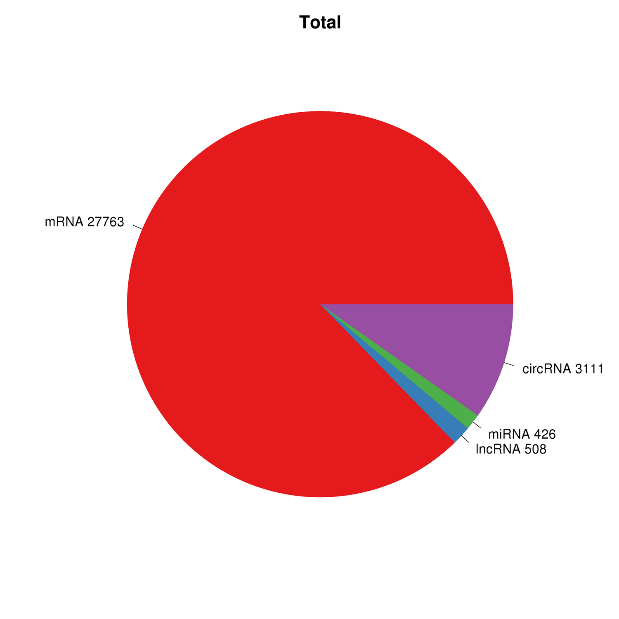
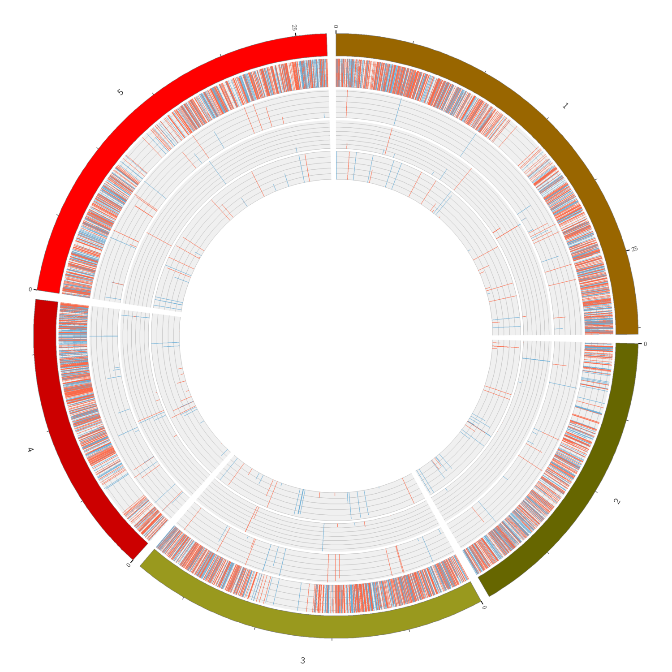
Panoramica dell'espressione dell'RNA
Geni espressi in modo differenziato
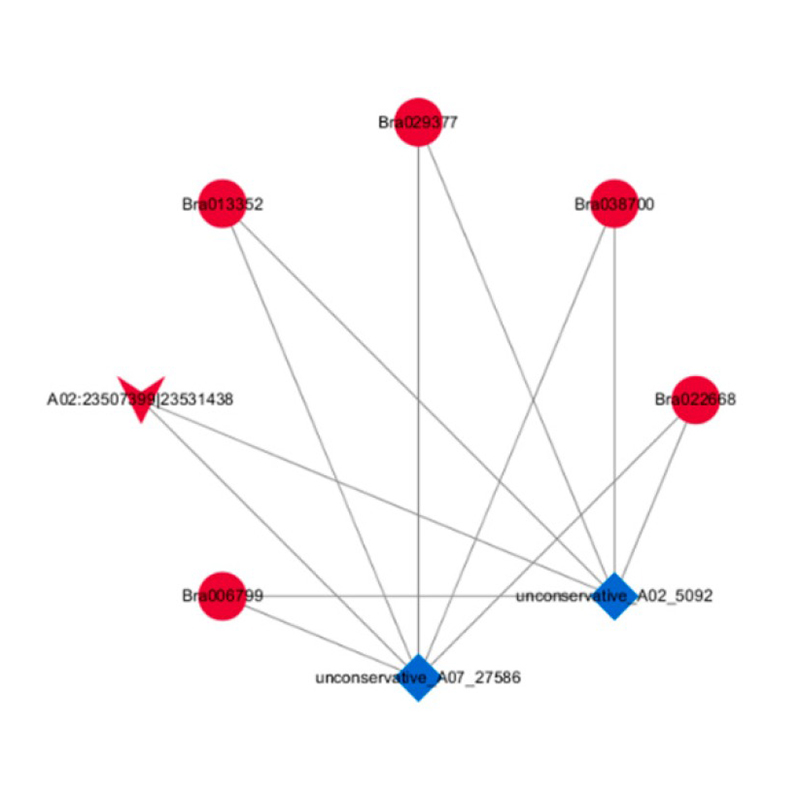
Analisi Cerna
Esplora i progressi della ricerca facilitati dai servizi di sequenziamento del trascrittoma di BMKGENE attraverso una raccolta curata di pubblicazioni.
Dai, Y. et al. (2022) "Profili di espressione completi di mRNA, lncRNA e miRNA nella malattia di Kashin-Beck identificati per sequenziamento dell'RNA", omics molecolare, 18 (2), pagg. 154–166. doi: 10.1039/d1mo00370d.
Liu, N. Nan et al. (2022) "Analisi dei trascrittomi a tutta lunghezza della resistenza a freddo delle API Cerana nel monte Changbai durante il periodo di svernamento.", Gene, 830, pp. 146503–146503. doi: 10.1016/j.gene.2022.146503.
Wang, XJ et al. (2022) "priorità basata sull'integrazione multi-OMIC delle reti di regolazione dell'RNA endogena in competizione nel carcinoma polmonare a piccole cellule: caratteristiche molecolari e candidati ai farmaci", Frontiers in Oncology, 12, p. 904865. Doi: 10.3389/fonc.2022.904865/bibtex.
Xu, P. et al. (2022) "L'analisi integrata dei profili di espressione lncRNA/CircrNA-MRNA-MRNA rivelano nuove intuizioni sui potenziali meccanismi in risposta ai nematodi a nodo radicale in arachidi", BMC Genomics, 23 (1), pp. 1–12. doi: 10.1186/s12864-022-08470-3/figure/7.
Yan, Z. et al. (2022) "Il sequenziamento dell'RNA per tutto il transscriptoma evidenzia i meccanismi molecolari associati al mantenimento della qualità post-tharvest nei broccoli mediante irradiazione a LED rossa", Biologia e tecnologia Postharvest, 188, p. 111878. Doi: 10.1016/j.postharvbio.2022.111878.