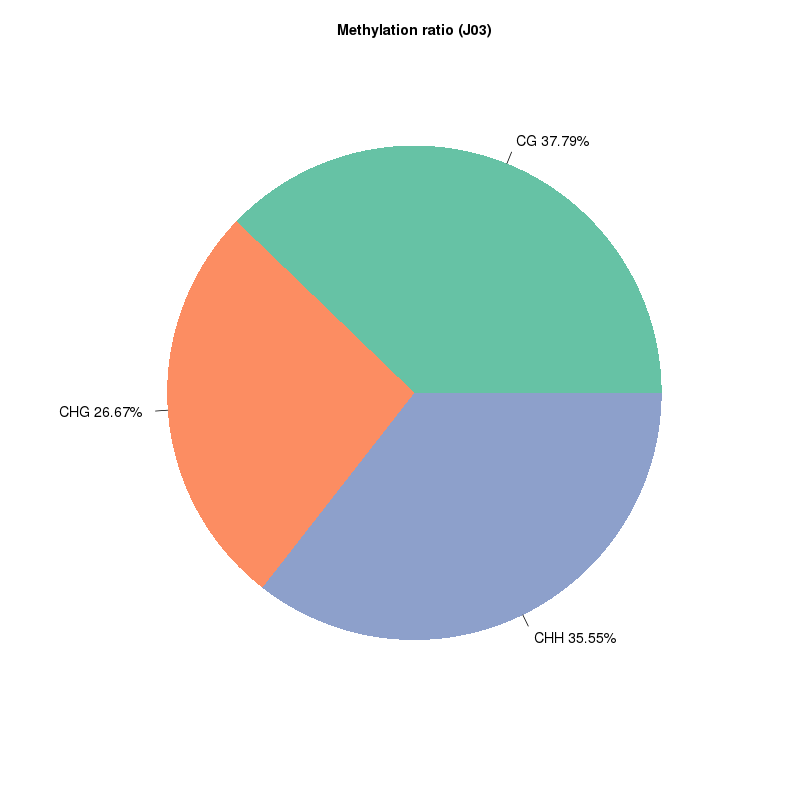Sequenziamento bisulico del genoma intero (WGBS)
Caratteristiche del servizio
● Richiede un genoma di riferimento.
● Il DNA Lambda viene aggiunto per monitorare l'efficienza di conversione del bisolfito.
● Sequenziamento su Illumina novaseq.
Vantaggi del servizio
●Gold Standard per la ricerca di metilazione del DNA: Questa tecnologia di elaborazione della conversione della metilazione matura ha un'alta precisione e una buona riproducibilità.
●Ampia copertura e risoluzione a base singola:Rilevazione di siti di metilazione a livello di genoma.
●Piattaforma completa:Fornire un servizio unico eccellente dalla lavorazione del campione, costruzione della libreria, sequenziamento all'analisi bioinformatica.
●Vasta competenza: Con i progetti di sequenziamento WGBS completati con successo in una vasta gamma di specie, BMKGENE porta oltre un decennio di esperienza, un team di analisi altamente qualificato, contenuti completi e un eccellente supporto post-vendita.
●Possibilità di unirsi all'analisi della trascrittomica: consentendo l'analisi integrata di WGBS con altri dati OMICS come l'RNA-seq.
Specifiche del campione
| Biblioteca | Strategia di sequenziamento | Output dei dati consigliato | Controllo di qualità |
| Bisulfito trattato | Illumina PE150 | 30x profondità | Q30 ≥ 85% Conversione del bisolfito> 99% |
Requisiti del campione
| Concentrazione (ng/µl) | Importo totale (µg) | Requisiti aggiuntivi | |
| DNA genomico | ≥ 5 | ≥ 400 ng | Degrado o contaminazione limitata |
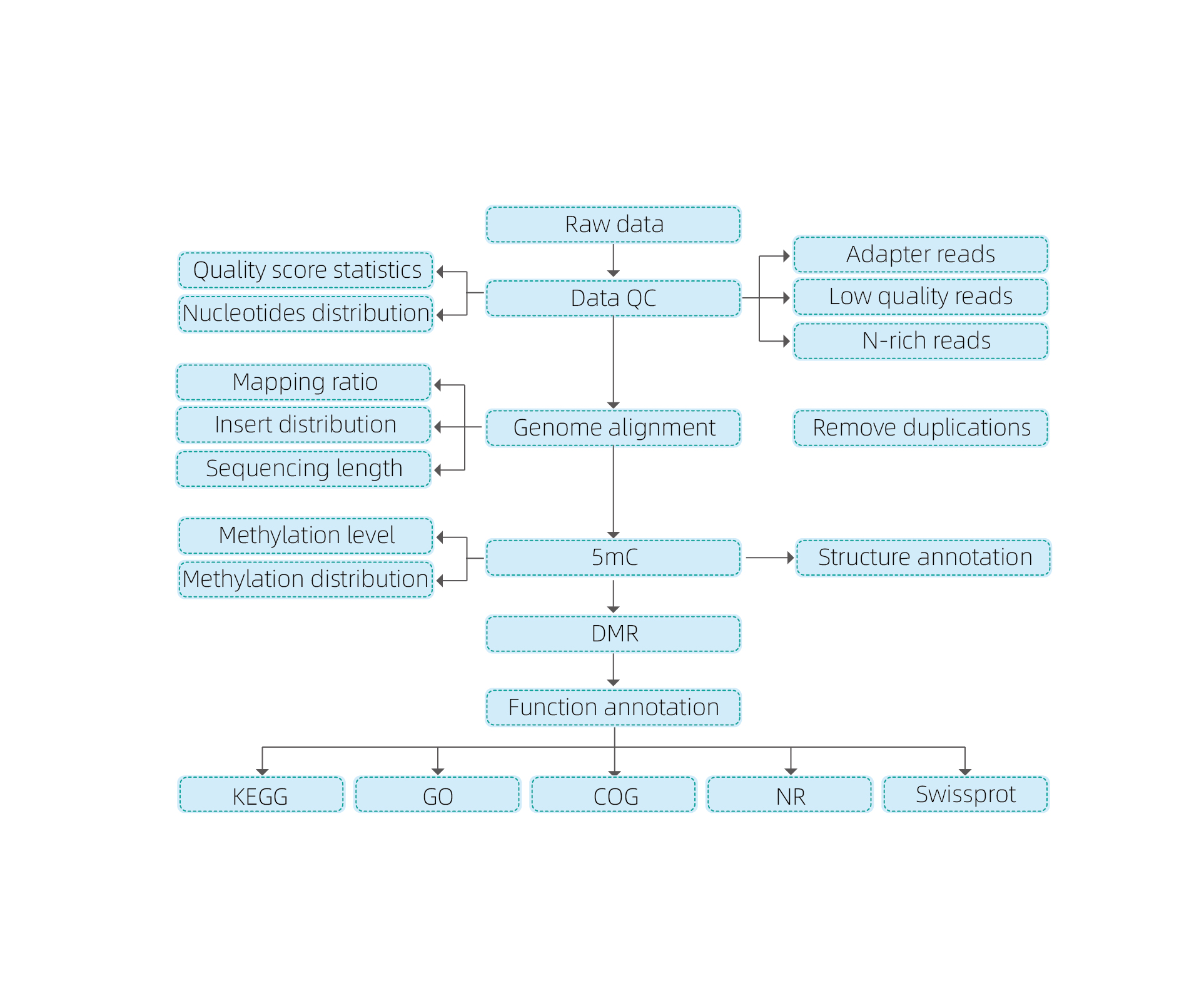
Flusso di lavoro di servizio

Consegna del campione

Estrazione del DNA

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Consegna dei dati
Include la seguente analisi:
● Controllo di qualità del sequenziamento grezzo;
● Mappatura al genoma di riferimento;
● Rilevazione di basi metilate da 5 mc;
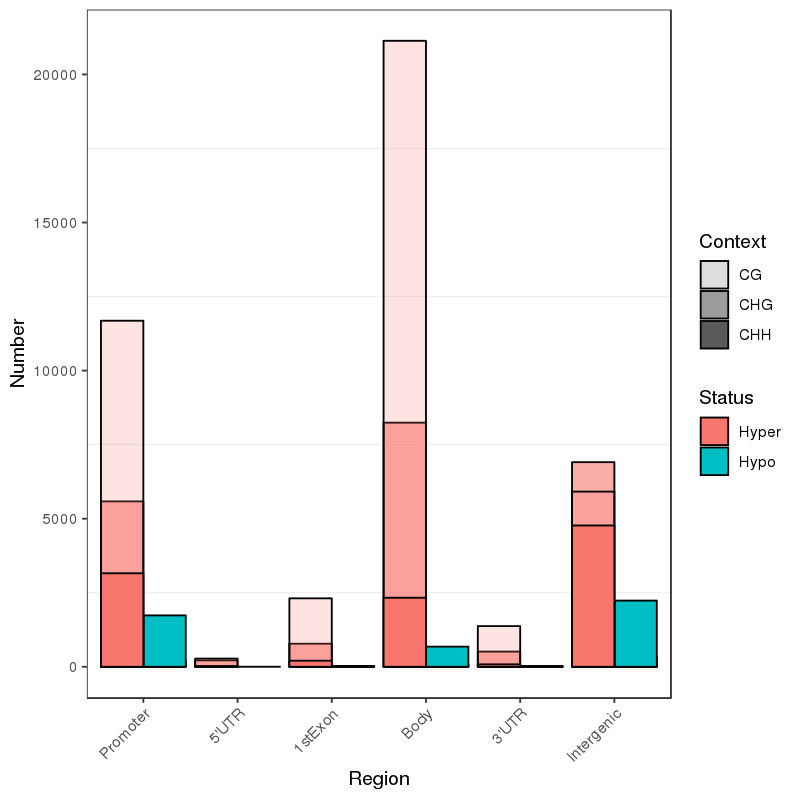
● Analisi della distribuzione e dell'annotazione della metilazione;
● Analisi delle regioni differenziate metilate (DMR);
● Annotazione funzionale dei geni associati ai DMR.
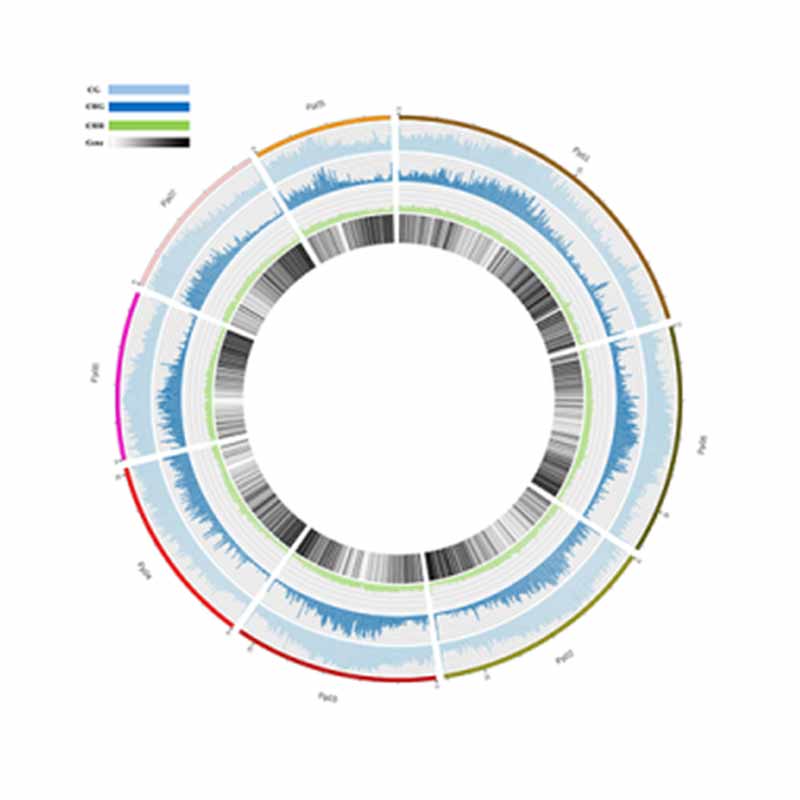
Rilevazione della metilazione 5mc: tipi di siti metilati
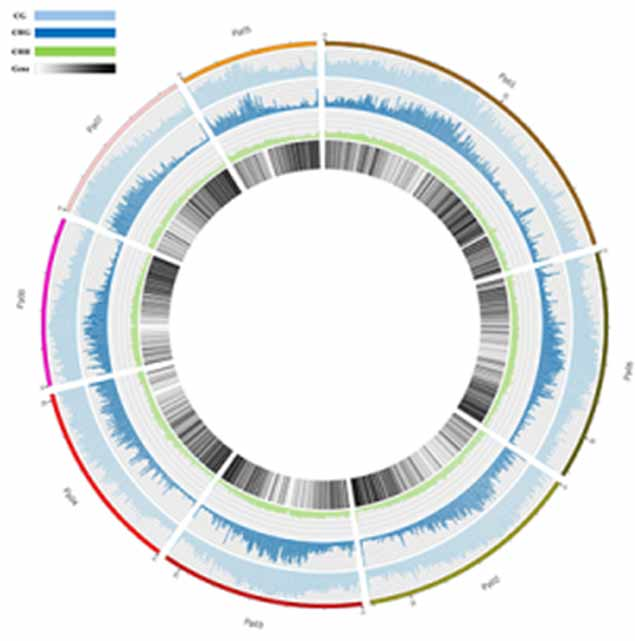
Mappa della metilazione. Distribuzione a livello del genoma della metilazione 5mc
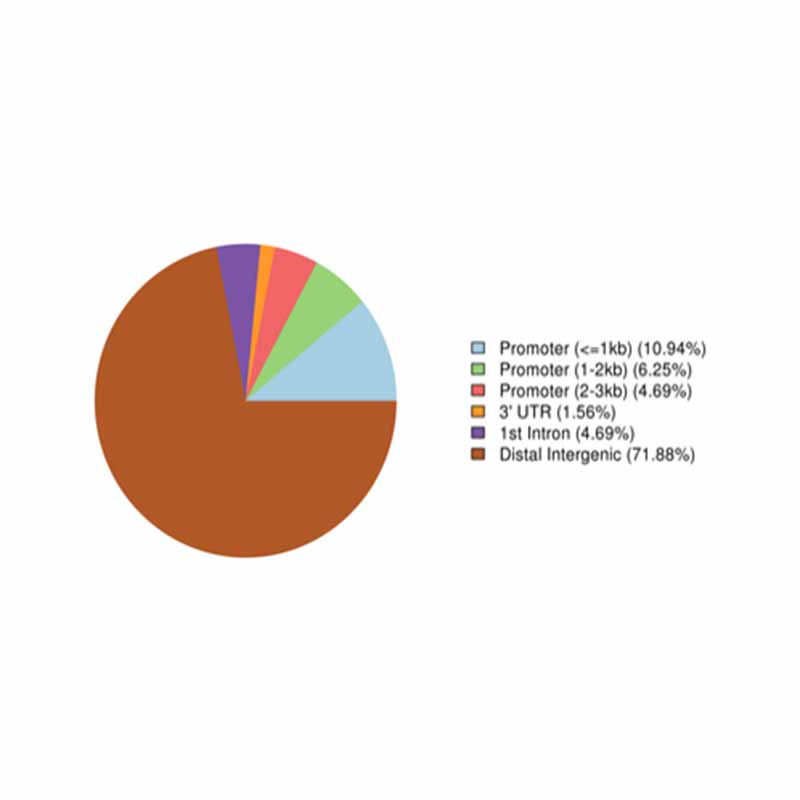
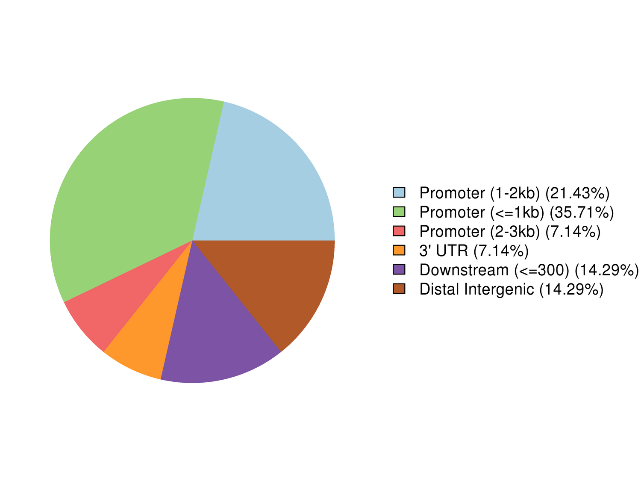
Annotazione di regioni altamente metilate
Regioni differenzialmente metilate: geni associati
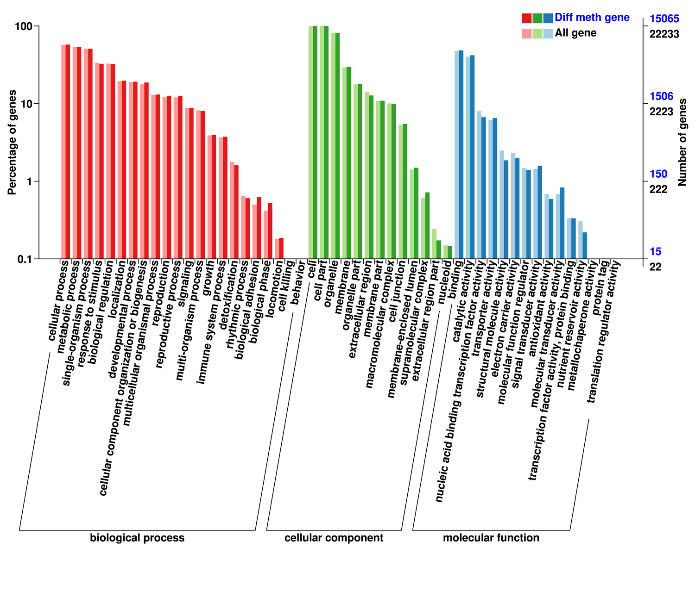
Regioni differenzialmente metilate: annotazione dei geni associati (ontologia genica)
Esplora i progressi della ricerca facilitati dagli interi servizi di sequenziamento del bisolfito del genoma di BMKGENE attraverso una raccolta curata di pubblicazioni.
Fan, Y. et al. (2020) "Analisi dei profili di metilazione del DNA durante lo sviluppo del muscolo scheletrico delle pecore usando il sequenziamento del bisolfito a genoma intero",Genomica BMC, 21 (1), pagg. 1–15. doi: 10.1186/s12864-020-6751-5.
Zhao, X. et al. (2022) "Nuove perturbazioni di metilazione dell'acido deossiribonucleico nei lavoratori esposti al cloruro di vinile",Tossicologia e salute industriale, 38 (7), pagg. 377–388. doi: 10.1177/07482337221098600
Zuo, J. et al. (2020) "Rapporti tra metilazione del genoma, livelli di RNA non codificanti, mRNA e metaboliti nella maturazione dei frutti di pomodoro",The Plant Journal, 103 (3), pagg. 980-994. doi: 10.1111/tpj.14778.