Piccolo sequenziamento dell'RNA-illumina
Caratteristiche
● La preparazione della libreria include un passaggio di selezione delle dimensioni
● Analisi bioinformatica incentrata sulla previsione di miRNA e i loro obiettivi
Vantaggi del servizio
●Analisi bioinformatica completa:Abilitando l'identificazione di miRNA sia noti che nuovi, identificazione di obiettivi di miRNA e corrispondente annotazione funzionale e arricchimento con più database (Kegg, Go)
●Controllo di qualità rigoroso: Implementiamo i punti di controllo di base in tutte le fasi, dalla preparazione di campioni e libreria al sequenziamento e alla bioinformatica. Questo meticoloso monitoraggio garantisce la consegna di risultati costantemente di alta qualità.
●Supporto post-vendita: Il nostro impegno si estende oltre il completamento del progetto con un periodo di servizio post-vendita di 3 mesi. Durante questo periodo, offriamo un follow-up del progetto, assistenza per la risoluzione dei problemi e sessioni di domande e risposte per affrontare eventuali domande relative ai risultati.
●Vasta competenza: Con una storia di chiusura con successo su più progetti SRNA che coprono oltre 300 specie in vari settori di ricerca, il nostro team porta una vasta esperienza in ogni progetto.
Requisiti di esempio e consegna
| Biblioteca | Piattaforma | Dati consigliati | Dati QC |
| Dimensione selezionata | Illumina SE50 | 10 m-20m | Q30≥85% |
Requisiti del campione:
Nucleotidi:
| CONC. (NG/μL) | Importo (μg) | Purezza | Integrità |
| ≥ 80 | ≥ 0,8 | OD260/280 = 1.7-2.5 OD260/230 = 0,5-2,5 Contaminazione da proteina o DNA limitata o nessuna mostrata su gel. | Rin≥6.0; 5,0≥28s/18S≥1.0; Elevazione limitata o non basale |
● Piante:
Radice, stelo o petalo: 450 mg
Foglia o seme: 300 mg
Frutto: 1,2 g
● Animale:
Cuore o intestino: 450 mg
Visceri o cervello: 240 mg
Muscolo: 600 mg
Ossa, capelli o pelle: 1,5 g
● Artropodi:
Insetti: 9g
Crustacea: 450 mg
● sangue intero: 2 tubi
● Celle: 106 cellule
● siero e plasma:6 ml
Consegna del campione consigliata
Contenitore: tubo di centrifuga da 2 ml (foglio di stagno non è raccomandato)
Etichettatura del campione: gruppo+replicare EG A1, A2, A3; B1, B2, B3.
Spedizione:
1. ICE a secco: i campioni devono essere imballati in borse e sepolti in ghiaccio a secco.
2. Tubi rnastable: i campioni di RNA possono essere essiccati nel tubo di stabilizzazione dell'RNA (EG RNastable®) e spediti a temperatura ambiente.
Flusso di lavoro di servizio

Design dell'esperimento

Consegna del campione

Estrazione di RNA

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Servizi post-vendita
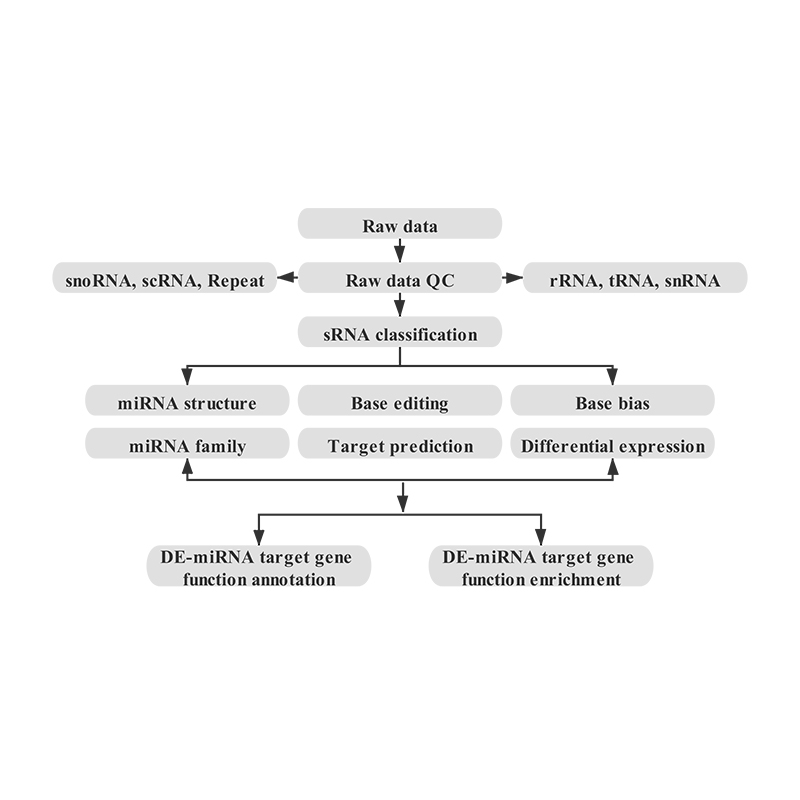
Bioinformatica
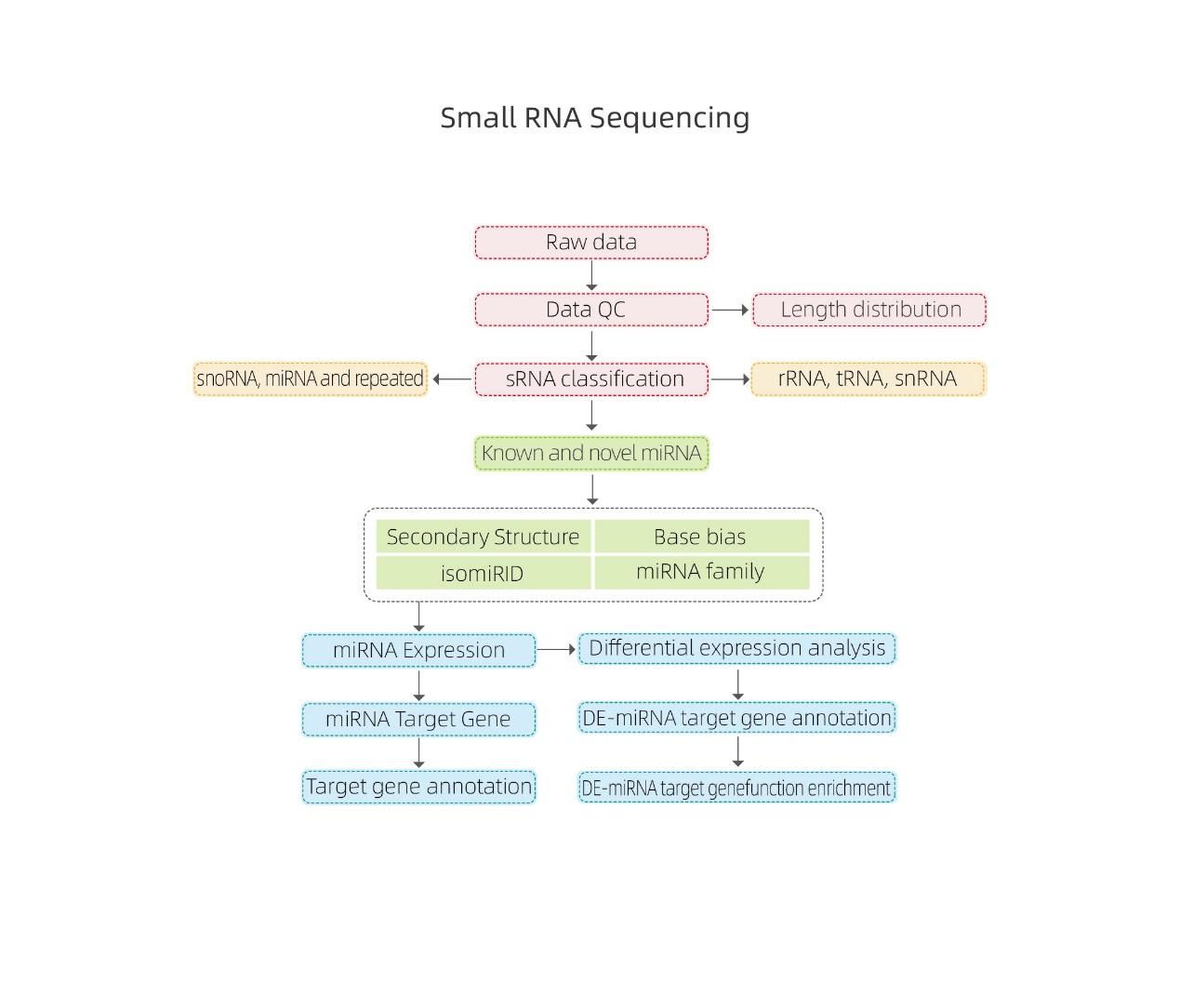 ● Controllo della qualità dei dati grezzi
● Controllo della qualità dei dati grezzi
● Classificazione SRNA
● Allineamento a un genoma di riferimento
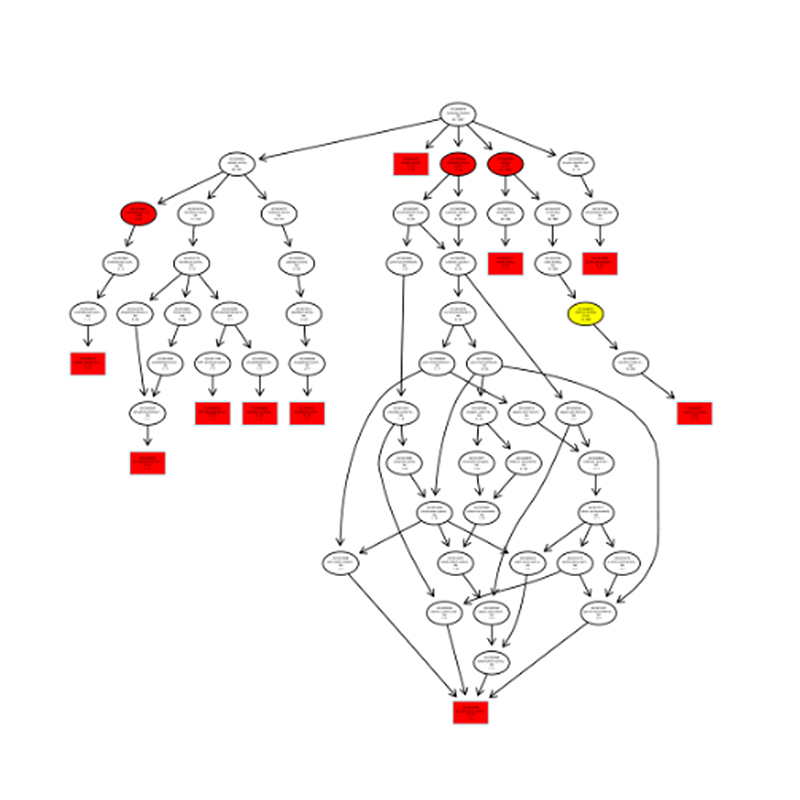
● Identificazione di miRNA noto e nuovo
● Analisi dell'espressione del miRNA differenziale
● Annotazione funzionale degli obiettivi di miRNA
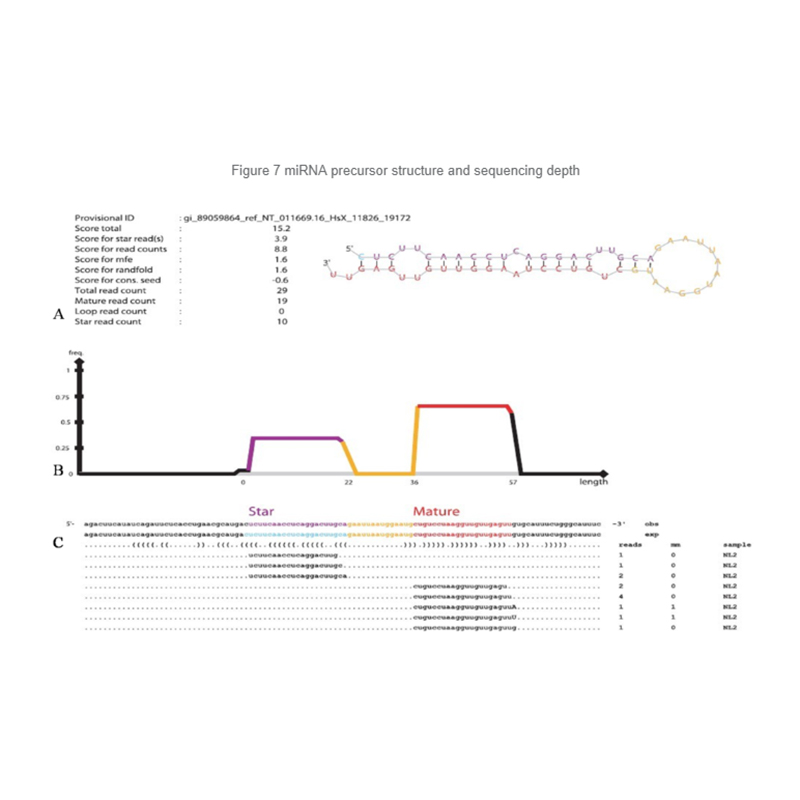
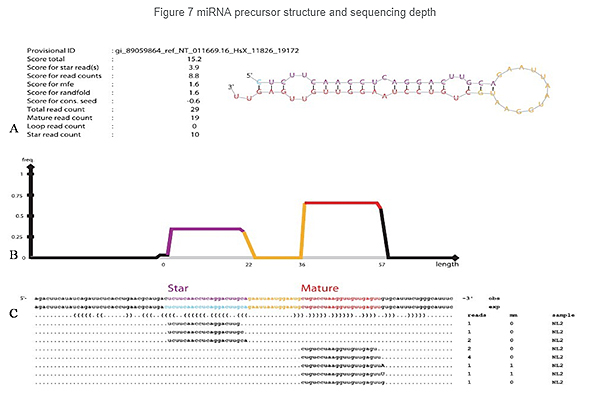
Identificazione del miRNA: struttura e profondità
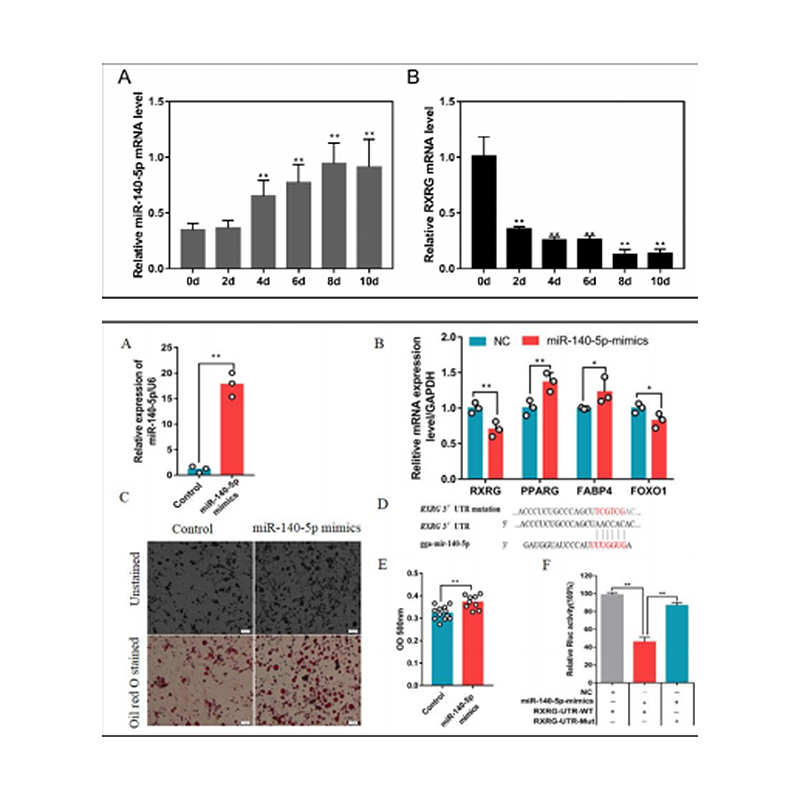
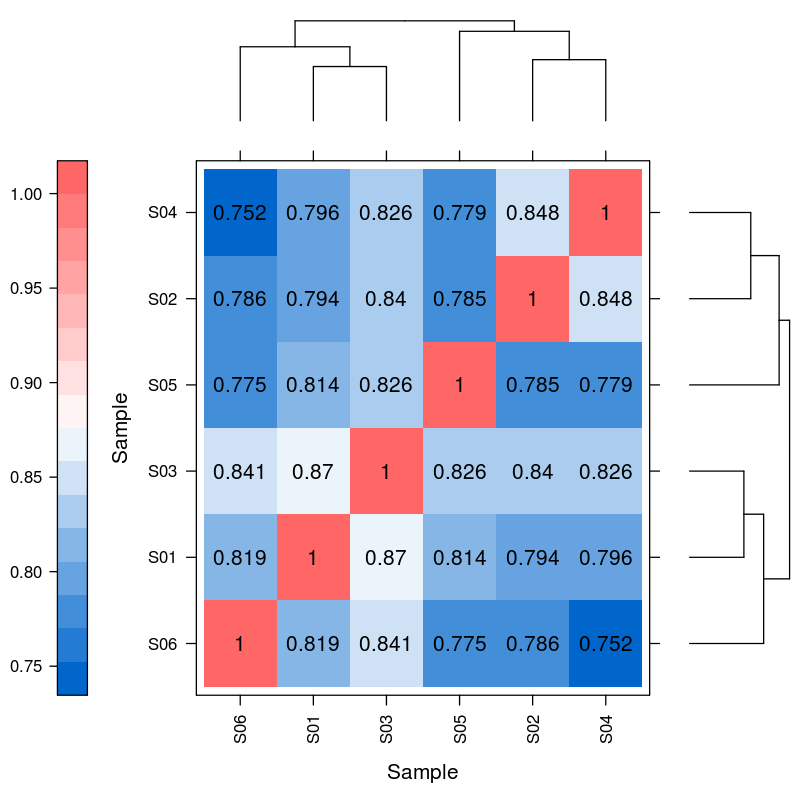
Espressione differenziale di miRNA - clustering hearchical
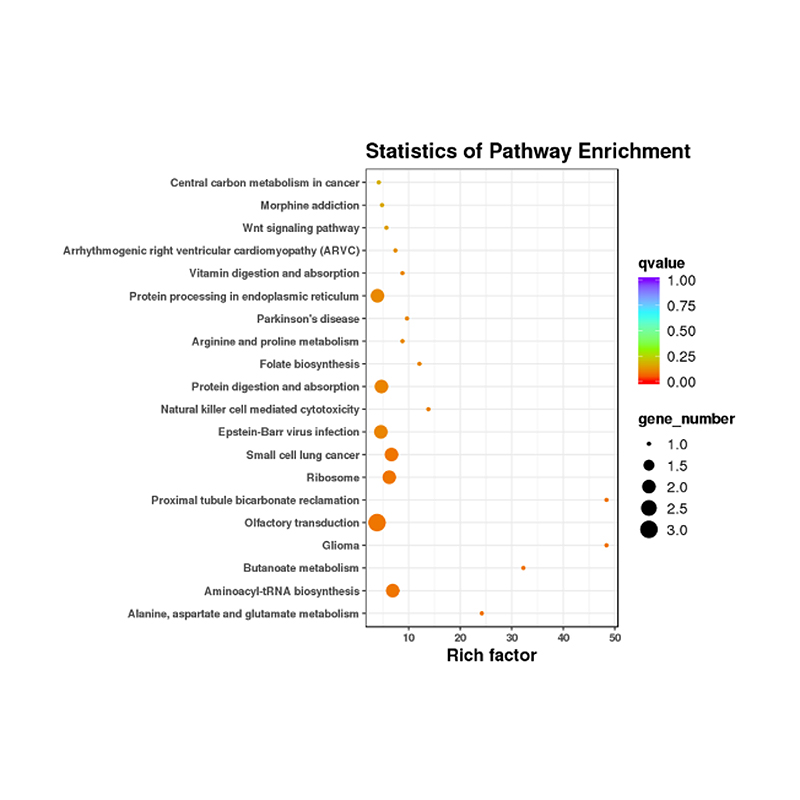
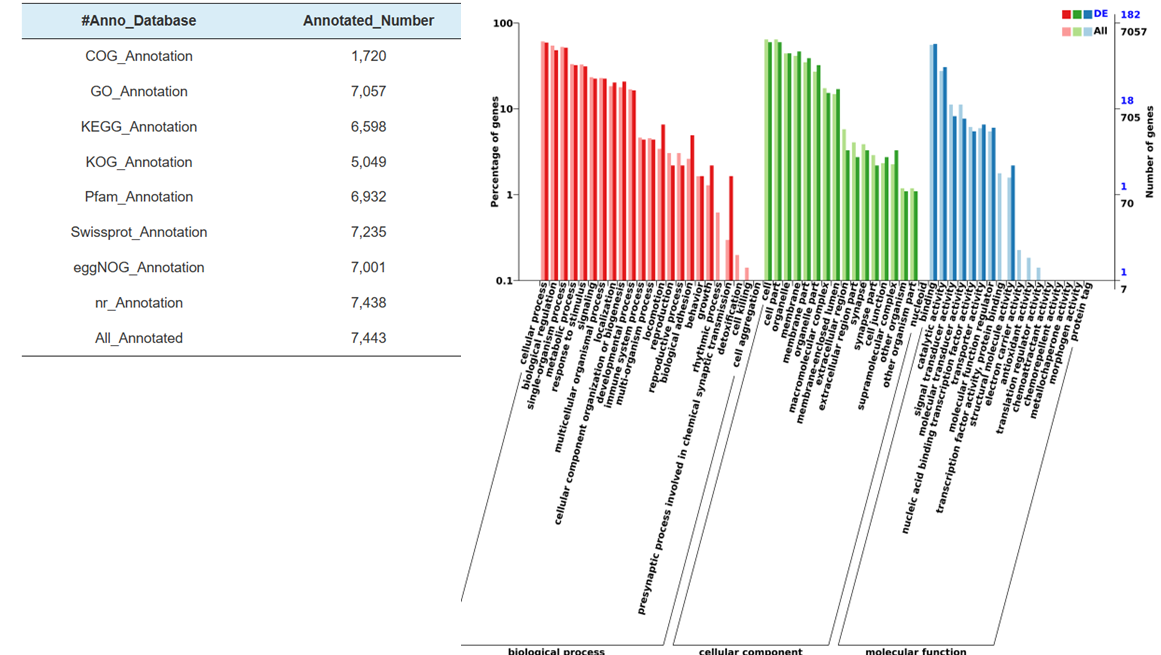
Annotazione funzionale del bersaglio di miRNA espressi in modo differenziato
Esplora i progressi della ricerca facilitati dai servizi di sequenziamento SRNA di BMKGENE attraverso una raccolta curata di pubblicazioni.
Chen, H. et al. (2023) "Le infezioni virali inibiscono la biosintesi e la fotosintesi della saponina in Panax notoginseng", Fisiologia delle piante e biochimica, 203, p. 108038. Doi: 10.1016/j.plaphy.2023.108038.
Li, H. et al. (2023) "La proteina Free1 contenente il dominio della pianta FYVE1 si associa a componenti a microprocessore per reprimere la biogenesi del miRNA", report EMBO, 24 (1). doi: 10.15252/emp.202255037/Suppl_file/EMB202255037-SUP-0004-SDATAFIG4.tif.
Yu, J. et al. (2023) 'The Microrna Ame-Bantam-3p controlla lo sviluppo della pupa larvale prendendo di mira i più domini simili a fattori di crescita epidermica 8 (MEGF8) in Honeybee, APIS Mellifera', International Journal of Molecular Sciences, 24 (6), p . 5726. Doi: 10.3390/ijms24065726/s1.
Zhang, M. et al. (2018) "L'analisi integrata di miRNA e geni associati alla qualità della carne rivela che GGA-MIR-140-5p influenza la deposizione di grasso intramuscolare nei polli", Fisiologia cellulare e Biochimica, 46 (6), pp. 2421-2433. doi: 10.1159/000489649.