-

Sequenziamento metagenomico -NGS
Un metagenoma è una raccolta del materiale genetico totale di una comunità mista di organismi, come metagenomi ambientali e umani. Contiene genomi di microrganismi coltivabili e non coltivabili. Il sequenziamento metagenomico Shotgun con NGS consente lo studio di questi intricati paesaggi genomici incorporati in campioni ambientali fornendo più di una semplice profilazione tassonomica, fornendo anche approfondimenti granulari sulla diversità delle specie, sulle dinamiche di abbondanza e sulle complesse strutture delle popolazioni. Oltre agli studi tassonomici, la metagenomica offre anche una prospettiva di genomica funzionale, consentendo l’esplorazione dei geni codificati e dei loro presunti ruoli nei processi ecologici. Infine, la creazione di reti di correlazione tra elementi genetici e fattori ambientali contribuisce a una comprensione olistica dell’intricata interazione tra le comunità microbiche e il loro background ecologico. In conclusione, il sequenziamento metagenomico rappresenta uno strumento fondamentale per svelare le complessità genomiche di diverse comunità microbiche, illuminando le molteplici relazioni tra genetica ed ecologia all’interno di questi ecosistemi complessi.
Piattaforme: Illumina NovaSeq e DNBSEQ-T7
-

Sequenziamento metagenomico-TGS
Un metagenoma è una raccolta del materiale genetico di una comunità mista di organismi, come metagenomi ambientali e umani. Contiene genomi di microrganismi coltivabili e non coltivabili. Il sequenziamento metagenomico consente lo studio di questi intricati paesaggi genomici incorporati in campioni ecologici fornendo qualcosa di più della semplice profilazione tassonomica. Offre inoltre una prospettiva di genomica funzionale esplorando i geni codificati e i loro presunti ruoli nei processi ambientali. Mentre gli approcci tradizionali con il sequenziamento Illumina sono stati ampiamente utilizzati negli studi metagenomici, l’avvento del sequenziamento a lettura lunga Nanopore e PacBio ha cambiato il campo. Le tecnologie Nanopore e PacBio migliorano le analisi bioinformatiche a valle, in particolare l'assemblaggio del metagenoma, garantendo assemblaggi più continui. I rapporti indicano che la metagenomica basata su nanopori e PacBio ha generato con successo genomi batterici completi e chiusi da microbiomi complessi (Moss, EL, et al., Nature Biotech, 2020). L'integrazione delle letture Nanopore con le letture Illumina fornisce un approccio strategico per la correzione degli errori, mitigando la bassa precisione intrinseca di Nanopore. Questa combinazione sinergica sfrutta i punti di forza di ciascuna piattaforma di sequenziamento, offrendo una soluzione solida per superare potenziali limiti e migliorare la precisione e l'affidabilità delle analisi metagenomiche.
Piattaforma: Nanopore PromethION 48, Illumia e PacBio Revio
-

Sequenziamento dell'intero genoma con bisolfito (WGBS)
Il sequenziamento del bisolfito dell'intero genoma (WGBS) rappresenta la metodologia gold-standard per l'esplorazione approfondita della metilazione del DNA, in particolare la quinta posizione nella citosina (5-mC), un regolatore fondamentale dell'espressione genica e dell'attività cellulare. Il principio alla base del WGBS prevede il trattamento con bisolfito, che induce la conversione delle citosine non metilate in uracile (da C a U), lasciando invariate le citosine metilate. Questa tecnica offre una risoluzione a base singola, consentendo ai ricercatori di indagare in modo completo sul metiloma e scoprire modelli di metilazione anomali associati a varie condizioni, in particolare al cancro. Utilizzando il WGBS, gli scienziati possono ottenere informazioni senza precedenti sui paesaggi di metilazione dell'intero genoma, fornendo una comprensione sfumata dei meccanismi epigenetici che sono alla base di diversi processi biologici e malattie.
-

Saggio per la cromatina accessibile alla trasposasi con sequenziamento ad alta produttività (ATAC-seq)
ATAC-seq è una tecnica di sequenziamento ad alto rendimento utilizzata per l'analisi dell'accessibilità della cromatina a livello dell'intero genoma. Il suo utilizzo fornisce una comprensione più profonda dei complessi meccanismi del controllo epigenetico globale sull’espressione genica. Il metodo utilizza una trasposasi Tn5 iperattiva per frammentare e contrassegnare contemporaneamente regioni di cromatina aperte inserendo adattatori di sequenziamento. La successiva amplificazione PCR porta alla creazione di una libreria di sequenziamento, che consente l'identificazione completa delle regioni di cromatina aperte in specifiche condizioni spazio-temporali. ATAC-seq fornisce una visione olistica dei paesaggi cromatinici accessibili, a differenza dei metodi che si concentrano esclusivamente sui siti di legame dei fattori di trascrizione o su specifiche regioni modificate dagli istoni. Sequenziando queste regioni di cromatina aperte, ATAC-seq rivela regioni con maggiori probabilità di sequenze regolatorie attive e potenziali siti di legame per fattori di trascrizione, offrendo preziose informazioni sulla modulazione dinamica dell'espressione genica attraverso il genoma.
-

Sequenziamento degli ampliconi 16S/18S/ITS-PacBio
I geni rRNA 16S e 18S, insieme alla regione ITS (Internal Transcript Spacer), fungono da marcatori molecolari fondamentali per l'impronta digitale grazie alla loro combinazione di regioni altamente conservate e ipervariabili, che li rendono strumenti preziosi per caratterizzare gli organismi procarioti ed eucariotici. L'amplificazione e il sequenziamento di queste regioni offrono un approccio privo di isolamento per studiare la composizione microbica e la diversità nei vari ecosistemi. Mentre il sequenziamento Illumina tipicamente prende di mira regioni ipervariabili brevi come V3-V4 di 16S e ITS1, è stato dimostrato che un'annotazione tassonomica superiore è ottenibile sequenziando l'intera lunghezza di 16S, 18S e ITS. Questo approccio globale si traduce in percentuali più elevate di sequenze accuratamente classificate, raggiungendo un livello di risoluzione che si estende all'identificazione delle specie. La piattaforma di sequenziamento Single-Molecule Real-Time (SMRT) di PacBio si distingue fornendo letture lunghe (HiFi) altamente accurate che coprono gli ampliconi a lunghezza intera, rivaleggiando con la precisione del sequenziamento Illumina. Questa capacità consente ai ricercatori di ottenere un vantaggio senza pari: una visione panoramica del panorama genetico. La copertura estesa aumenta significativamente la risoluzione nell’annotazione delle specie, in particolare all’interno delle comunità batteriche o fungine, consentendo una comprensione più profonda delle complessità delle popolazioni microbiche.
-

Sequenziamento degli ampliconi 16S/18S/ITS-NGS
Il sequenziamento degli ampliconi con la tecnologia Illumina, mirato specificamente ai marcatori genetici 16S, 18S e ITS, è un metodo potente per svelare la filogenesi, la tassonomia e l'abbondanza delle specie all'interno delle comunità microbiche. Questo approccio prevede il sequenziamento delle regioni ipervariabili dei marcatori genetici domestici. Originariamente introdotta come impronta molecolare daWoeses et alnel 1977, questa tecnica ha rivoluzionato la profilazione del microbioma consentendo analisi senza isolamento. Attraverso il sequenziamento di 16S (batteri), 18S (funghi) e Internal Transcript Spacer (ITS, funghi), i ricercatori possono identificare non solo specie abbondanti ma anche specie rare e non identificate. Ampiamente adottato come strumento fondamentale, il sequenziamento degli ampliconi è diventato determinante nel discernere le composizioni microbiche differenziali in diversi ambienti, tra cui la bocca umana, l’intestino, le feci e oltre.
-

Risequenziamento dell'intero genoma batterico e fungino
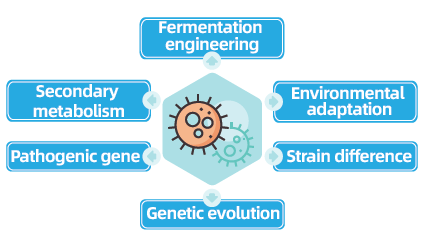
I progetti di risequenziamento dell’intero genoma di batteri e funghi sono fondamentali per il progresso della genomica microbica consentendo il completamento e il confronto dei genomi microbici. Ciò facilita l'ingegneria della fermentazione, l'ottimizzazione dei processi industriali e l'esplorazione delle vie del metabolismo secondario. Inoltre, il risequenziamento di funghi e batteri è fondamentale per comprendere l’adattamento ambientale, ottimizzare i ceppi e rivelare le dinamiche dell’evoluzione genetica, con ampie implicazioni in medicina, agricoltura e scienze ambientali.
-

Sequenziamento degli ampliconi 16S/18S/ITS a lunghezza intera PacBio
La piattaforma Amplicon (16S/18S/ITS) è sviluppata con anni di esperienza nell'analisi di progetti sulla diversità microbica, che contiene analisi di base standardizzate e analisi personalizzate: l'analisi di base copre il contenuto di analisi principale dell'attuale ricerca microbica, il contenuto dell'analisi è ricco e completo, e i risultati dell'analisi sono presentati sotto forma di relazioni di progetto; Il contenuto dell'analisi personalizzata è vario. È possibile selezionare i campioni e impostare i parametri in modo flessibile in base al rapporto di analisi di base e allo scopo della ricerca, per realizzare requisiti personalizzati. Sistema operativo Windows, semplice e veloce.
-

PacBio-Trascrittoma a lunghezza intera (non di riferimento)
Prendendo come input i dati di sequenziamento delle isoforme di Pacific Biosciences (PacBio), questa app è in grado di identificare sequenze di trascrizione a lunghezza intera (senza assemblaggio). Mappando sequenze a lunghezza intera rispetto al genoma di riferimento, le trascrizioni possono essere ottimizzate da geni, trascrizioni, regioni codificanti noti, ecc. In questo caso, è possibile ottenere un'identificazione più accurata delle strutture dell'mRNA, come lo splicing alternativo, ecc. L'analisi congiunta con i dati di sequenziamento del trascrittoma NGS consente un'annotazione più completa e una quantificazione più accurata dell'espressione a livello di trascrizione, il che avvantaggia ampiamente l'espressione differenziale a valle e l'analisi funzionale.
-

Sequenziamento del bisolfito a rappresentazione ridotta (RRBS)
Il sequenziamento del bisolfito a rappresentazione ridotta (RRBS) è emerso come un'alternativa economica ed efficiente al sequenziamento del bisolfito dell'intero genoma (WGBS) nella ricerca sulla metilazione del DNA. Sebbene il WGBS fornisca approfondimenti completi esaminando l'intero genoma con una risoluzione di base singola, il suo costo elevato può essere un fattore limitante. RRBS mitiga strategicamente questa sfida analizzando selettivamente una porzione rappresentativa del genoma. Questa metodologia si basa sull'arricchimento delle regioni ricche di isole CpG mediante scissione MspI seguita dalla selezione delle dimensioni di frammenti di 200-500/600 bps. Di conseguenza, vengono sequenziate solo le regioni prossimali alle isole CpG, mentre quelle con isole CpG distanti sono escluse dall'analisi. Questo processo, combinato con il sequenziamento con bisolfito, consente il rilevamento ad alta risoluzione della metilazione del DNA e l'approccio di sequenziamento, PE150, si concentra specificamente sulle estremità degli inserti anziché sulla parte centrale, aumentando l'efficienza del profilo di metilazione. L'RRBS è uno strumento inestimabile che consente una ricerca economicamente vantaggiosa sulla metilazione del DNA e fa avanzare la conoscenza dei meccanismi epigenetici.
-

Sequenziamento dell'RNA procariotico
Il sequenziamento dell'RNA consente la profilazione completa di tutte le trascrizioni di RNA all'interno delle cellule in condizioni specifiche. Questa tecnologia all’avanguardia funge da potente strumento, svelando intricati profili di espressione genetica, strutture genetiche e meccanismi molecolari associati a diversi processi biologici. Ampiamente adottato nella ricerca fondamentale, nella diagnostica clinica e nello sviluppo di farmaci, il sequenziamento dell'RNA offre approfondimenti sulle complessità della dinamica cellulare e della regolazione genetica. La nostra elaborazione dei campioni di RNA procariotico è personalizzata per i trascrittomi procariotici, coinvolgendo l'esaurimento dell'rRNA e la preparazione della libreria direzionale.
Piattaforma: Illumina NovaSeq
-

Sequenziamento del metatrascrittoma
Sfruttando la tecnologia di sequenziamento Illumina, il servizio di sequenziamento del metatrascrittoma di BMKGENE svela l'espressione genetica dinamica di una vasta gamma di microbi, dagli eucarioti ai procarioti e ai virus, all'interno di ambienti naturali come suolo, acqua, mare, feci e intestino. Il nostro servizio completo consente ai ricercatori di approfondire i profili completi di espressione genetica delle comunità microbiche complesse. Oltre all'analisi tassonomica, il nostro servizio di sequenziamento del metatrascrittoma facilita l'esplorazione dell'arricchimento funzionale, facendo luce sui geni espressi in modo differenziale e sui loro ruoli. Scopri una vasta gamma di approfondimenti biologici mentre esplori i complessi paesaggi dell'espressione genetica, della diversità tassonomica e delle dinamiche funzionali all'interno di queste diverse nicchie ambientali.
-

Assemblaggio del genoma fungino de novo
BMKGENE offre soluzioni versatili per i genomi fungini, soddisfacendo le diverse esigenze di ricerca e la completezza del genoma desiderata. Il solo utilizzo del sequenziamento Illumina a lettura breve consente la generazione di una bozza di genoma. Il sequenziamento a lettura breve e a lettura lunga utilizzando Nanopore o Pacbio viene combinato per ottenere un genoma fungino più raffinato con contigui più lunghi. Inoltre, l'integrazione del sequenziamento Hi-C migliora ulteriormente le capacità, consentendo il raggiungimento di un genoma completo a livello cromosomico.