Sequenziamento di mRNA-NGS non basato su riferimento
Caratteristiche
● Cattura del polimRNA prima della preparazione della libreria
● Indipendente da qualsiasi genoma di riferimento: basato sull'assemblaggio de novo delle trascrizioni, generando un elenco di unigeni annotati con più database (NR, Swiss-Prot, COG, KOG, EggNOG, Pfam, GO, KEGG)
● Analisi bioinformatica completa dell'espressione genica e della struttura della trascrizione
Vantaggi del servizio
●Ampia competenza: con un track record di elaborazione di oltre 600.000 campioni presso BMKGENE, che abbracciano diversi tipi di campioni come colture cellulari, tessuti e fluidi corporei, il nostro team apporta una vasta esperienza a ogni progetto. Abbiamo chiuso con successo oltre 100.000 progetti mRNA-Seq in vari ambiti di ricerca.
●Controllo qualità rigoroso: implementiamo punti di controllo fondamentali in tutte le fasi, dalla preparazione dei campioni e delle librerie al sequenziamento e alla bioinformatica. Questo meticoloso monitoraggio garantisce la fornitura di risultati costantemente di alta qualità.
● Annotazione completa: utilizziamo più database per annotare funzionalmente i geni differenzialmente espressi (DEG) ed eseguire la corrispondente analisi di arricchimento, fornendo approfondimenti sui processi cellulari e molecolari alla base della risposta del trascrittoma.
●Supporto post-vendita: il nostro impegno va oltre il completamento del progetto con un periodo di servizio post-vendita di 3 mesi. Durante questo periodo, offriamo follow-up del progetto, assistenza per la risoluzione dei problemi e sessioni di domande e risposte per rispondere a qualsiasi domanda relativa ai risultati.
Requisiti e consegna del campione
| Biblioteca | Strategia di sequenziamento | Dati consigliati | Controllo di qualità |
| Poli A arricchito | Illumina PE150 DNBSEQ-T7 | 6-10 GB | Q30≥85% |
Requisiti del campione:
Nucleotidi:
| Concentrazione (ng/μl) | Quantità (μg) | Purezza | Integrità |
| ≥ 10 | ≥ 0,2 | DE260/280=1,7-2,5 DE260/230=0,5-2,5 Contaminazione limitata o assente di proteine o DNA mostrata sul gel. | Per gli impianti: RIN≥4,0; Per gli animali: RIN≥4,5; 5,0≥28S/18S≥1,0; elevazione della linea di base limitata o assente |
● Piante:
Radice, stelo o petalo: 450 mg
Foglia o seme: 300 mg
Frutta: 1,2 g
● Animale:
CUORE o Intestino: 300 mg
Visceri o cervello: 240 mg
Muscolo: 450 mg
Ossa, capelli o pelle: 1 g
● Artropodi:
Insetti: 6g
Crostacei: 300 mg
● Sangue intero: 1 tubo
● Celle: 106 cellule
Consegna del campione consigliata
Contenitore: provetta da centrifuga da 2 ml (la carta stagnola non è consigliata)
Etichettatura del campione: Gruppo+replica, ad esempio A1, A2, A3; B1, B2, B3.
Spedizione:
1. Ghiaccio secco: i campioni devono essere confezionati in sacchetti e sepolti nel ghiaccio secco.
2. Provette RNAstable: i campioni di RNA possono essere essiccati in provette di stabilizzazione dell'RNA (ad esempio RNAstable®) e spediti a temperatura ambiente.
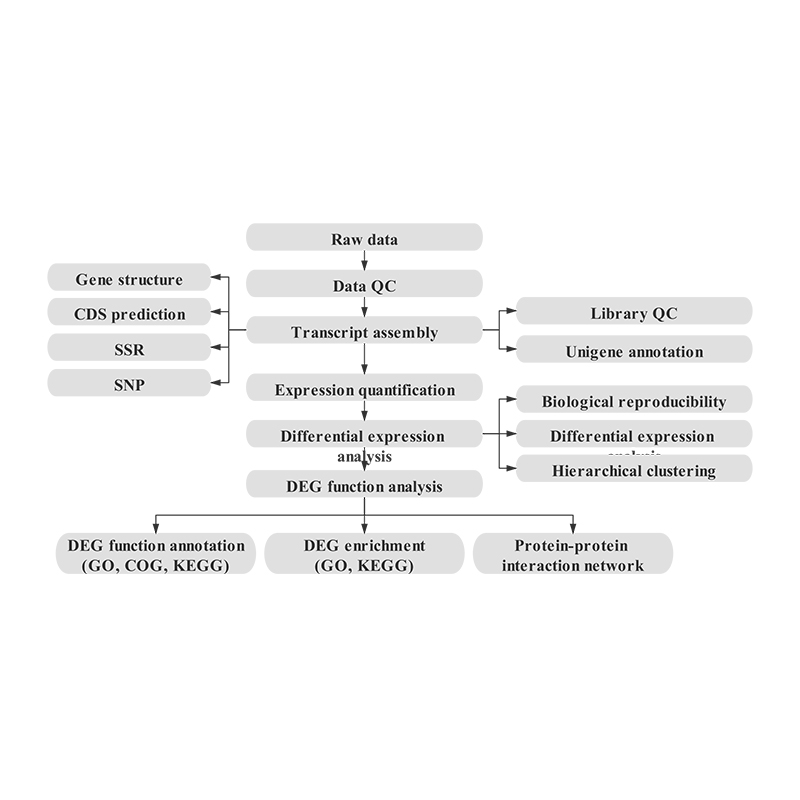
Flusso di lavoro del servizio

Progettazione dell'esperimento

Consegna del campione

Estrazione dell'RNA

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Servizi post-vendita
Bioinformatica
Assemblaggio del trascrittoma e selezione unigenica
Annotazione Unigene
Correlazione dei campioni e valutazione delle repliche biologiche
Geni differenzialmente espressi (DEG)
Annotazione funzionale dei DEG
Arricchimento funzionale dei DEG
Esplora i progressi facilitati dai servizi di sequenziamento dell'mRNA NGS eucariotico di BMKGene attraverso una raccolta curata di pubblicazioni.
Shen, F. et al. (2020) "Assemblaggio del trascrittoma de novo ed espressione genetica distorta dal sesso nelle gonadi del pesce gatto dell'Amur (Silurus asotus)", Genomics, 112(3), pp. 2603–2614. doi: 10.1016/J.YGENO.2020.01.026.
Zhang, C. et al. (2016) "Analisi del trascrittoma del metabolismo del saccarosio durante il rigonfiamento e lo sviluppo del bulbo nella cipolla (Allium cepa L.)", Frontiers in Plant Science, 7 (settembre), p. 212763.doi: 10.3389/FPLS.2016.01425/BIBTEX.
Zhu, C. et al. (2017) "Assemblaggio de novo, caratterizzazione e annotazione per il trascrittoma di Sarcocheilichthys sinensis", PLoS ONE, 12(2). doi: 10.1371/JOURNAL.PONE.0171966.
Zou, L. et al. (2021) "L'analisi del trascrittoma de novo fornisce approfondimenti sulla tolleranza al sale di Podocarpus macrophyllus sotto stress da salinità", BMC Plant Biology, 21(1), pp. 1–17. doi: 10.1186/S12870-021-03274-1/FIGURES/9.