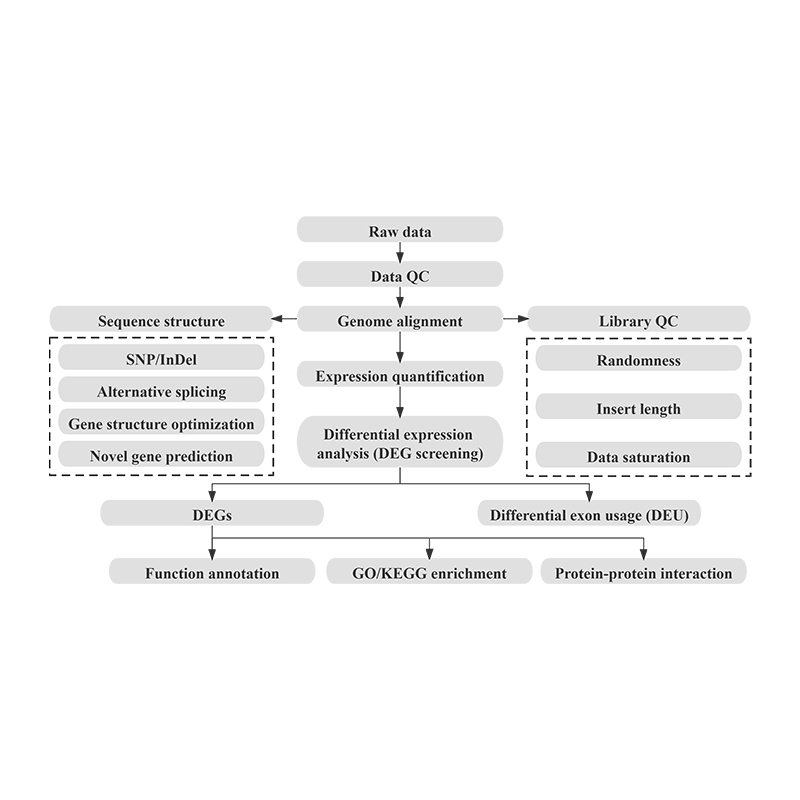
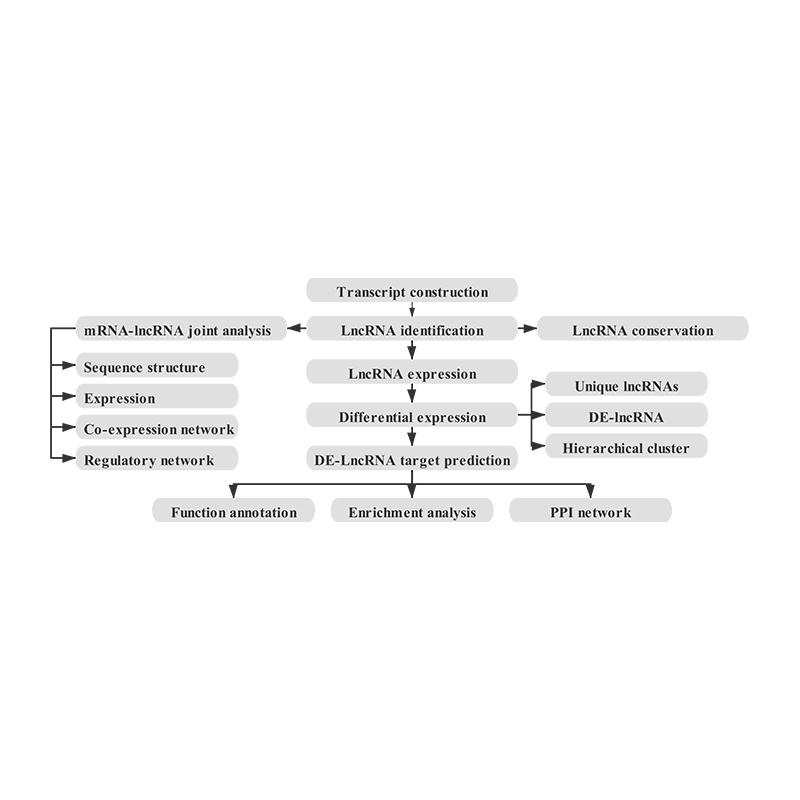
Sequenziamento lungo non codificante-Illumina
Vantaggi del servizio
●Analisi congiunta di mRNA e lncRNA: combinando la quantificazione dei trascritti di mRNA con lo studio degli lncRNA e dei loro target, è possibile ottenere una panoramica approfondita del meccanismo di regolazione alla base della risposta cellulare.
●Ampia competenza: Il nostro team apporta una vasta esperienza a ogni progetto, con un track record di elaborazione di oltre 23.000 campioni presso BMK che abbraccia diversi tipi di campioni e progetti lncRNA.
●Controllo qualità rigoroso: Implementiamo punti di controllo fondamentali in tutte le fasi, dalla preparazione dei campioni e delle librerie al sequenziamento e alla bioinformatica. Questo meticoloso monitoraggio garantisce la fornitura di risultati costantemente di alta qualità.
●Supporto post-vendita: Il nostro impegno si estende oltre il completamento del progetto con un periodo di servizio post-vendita di 3 mesi. Durante questo periodo, offriamo follow-up del progetto, assistenza per la risoluzione dei problemi e sessioni di domande e risposte per rispondere a qualsiasi domanda relativa ai risultati.
Requisiti e consegna del campione
| Biblioteca | Piattaforma | Dati consigliati | Controllo qualità dei dati |
| Libreria direzionale impoverita di rRNA | Illumina PE150 | 10-16 GB | Q30≥85% |
Nucleotidi:
| Concentrazione (ng/μl) | Quantità (μg) | Purezza | Integrità |
| ≥ 80 | ≥ 0,8 | DE260/280=1,7-2,5 DE260/230=0,5-2,5 Contaminazione limitata o assente di proteine o DNA mostrata sul gel. | RIN≥6.0; 5,0≥28S/18S≥1,0; elevazione della linea di base limitata o assente |
● Piante:
Radice, stelo o petalo: 450 mg
Foglia o seme: 300 mg
Frutta: 1,2 g
● Animale:
Cuore o Intestino: 450 mg
Visceri o cervello: 240 mg
Muscolo: 600 mg
Ossa, capelli o pelle: 1,5 g
● Artropodi:
Insetti: 9 g
Crostacei: 450 mg
● Sangue intero:2 tubi
● Celle: 106 cellule
● Siero e plasma: 6 ml
Consegna del campione consigliata
Contenitore: provetta da centrifuga da 2 ml (la carta stagnola non è consigliata)
Etichettatura del campione: Gruppo+replica, ad esempio A1, A2, A3; B1, B2, B3.
Spedizione:
1. Ghiaccio secco: i campioni devono essere confezionati in sacchetti e sepolti nel ghiaccio secco.
2. Provette RNAstable: i campioni di RNA possono essere essiccati in provette di stabilizzazione dell'RNA (ad esempio RNAstable®) e spediti a temperatura ambiente.
Flusso di lavoro del servizio

Progettazione dell'esperimento

Consegna del campione

Estrazione dell'RNA

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Servizi post-vendita
Bioinformatica
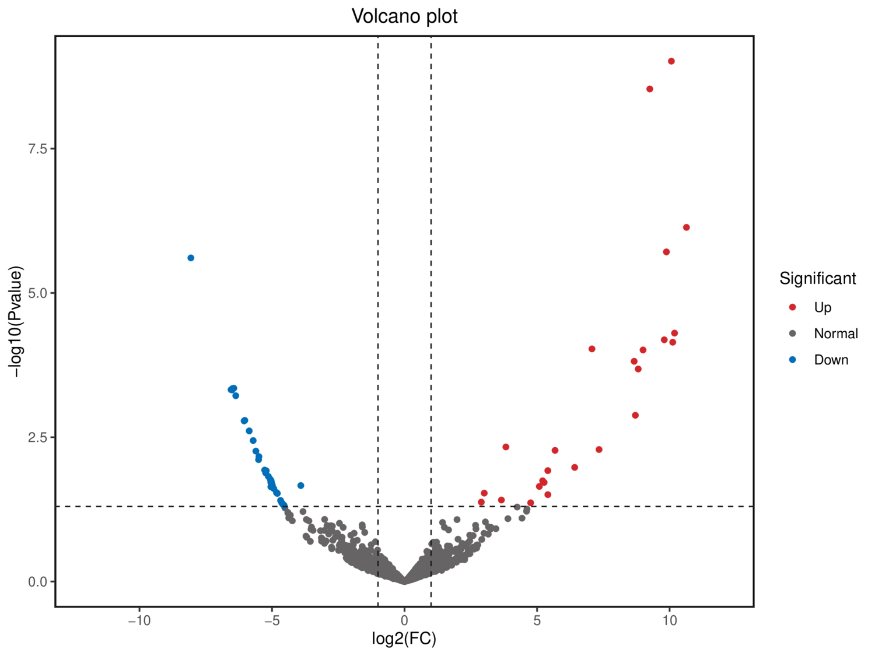
Analisi dell'espressione genica differenziale (DEG).
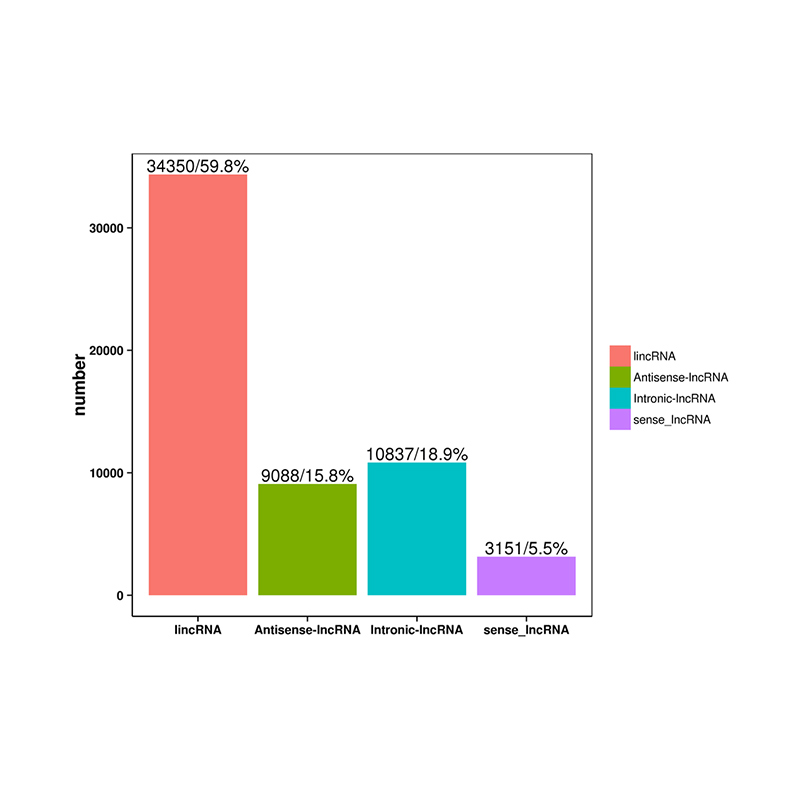
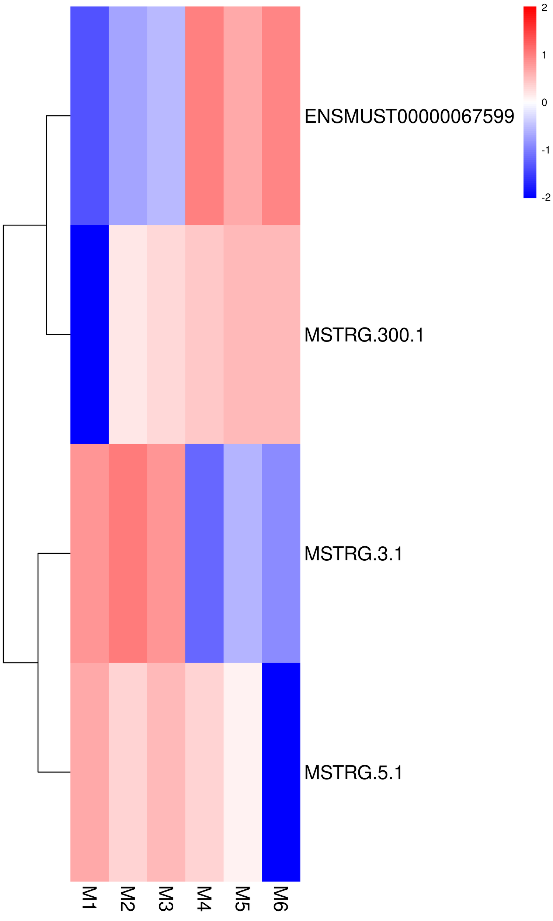
Quantificazione dell'espressione di lncRNA – clustering
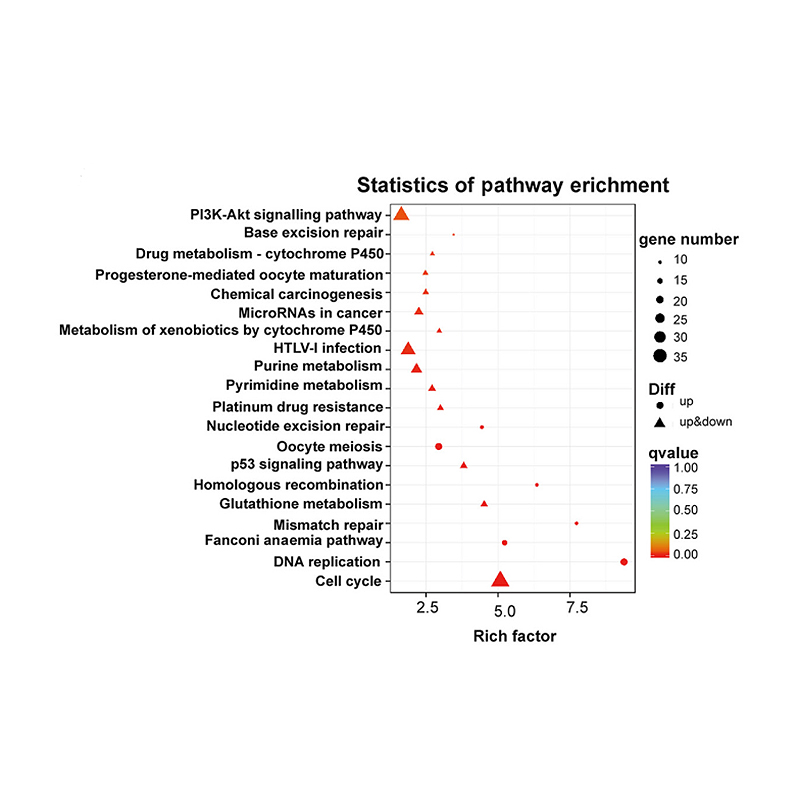
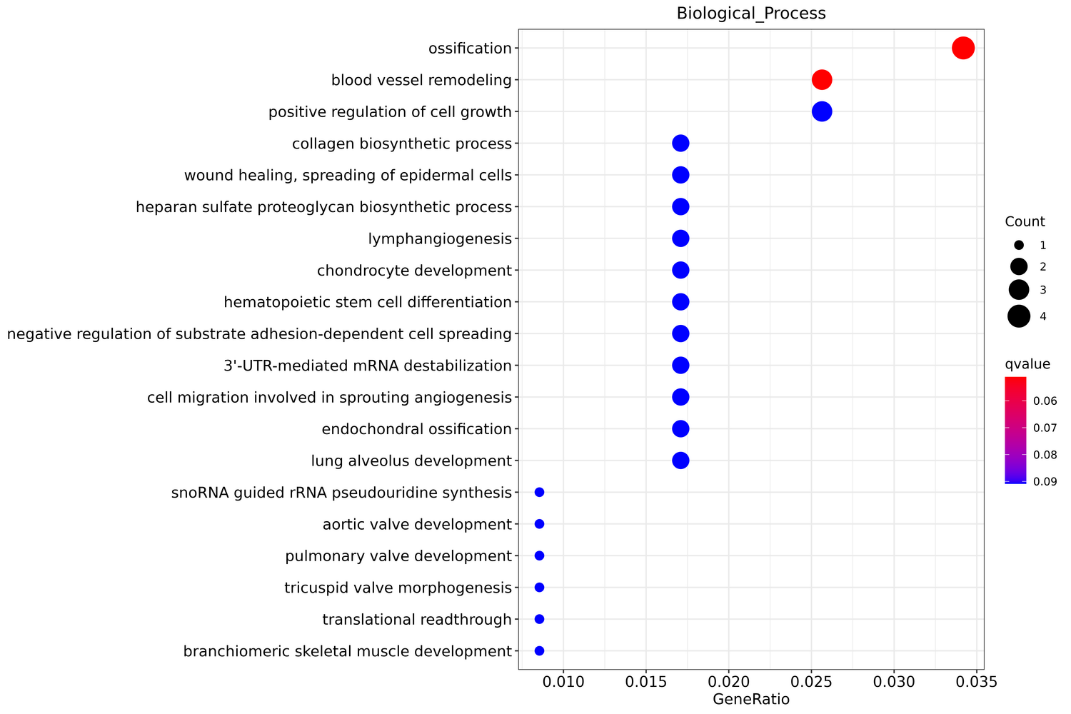
Arricchimento dei geni bersaglio dell'lncRNA
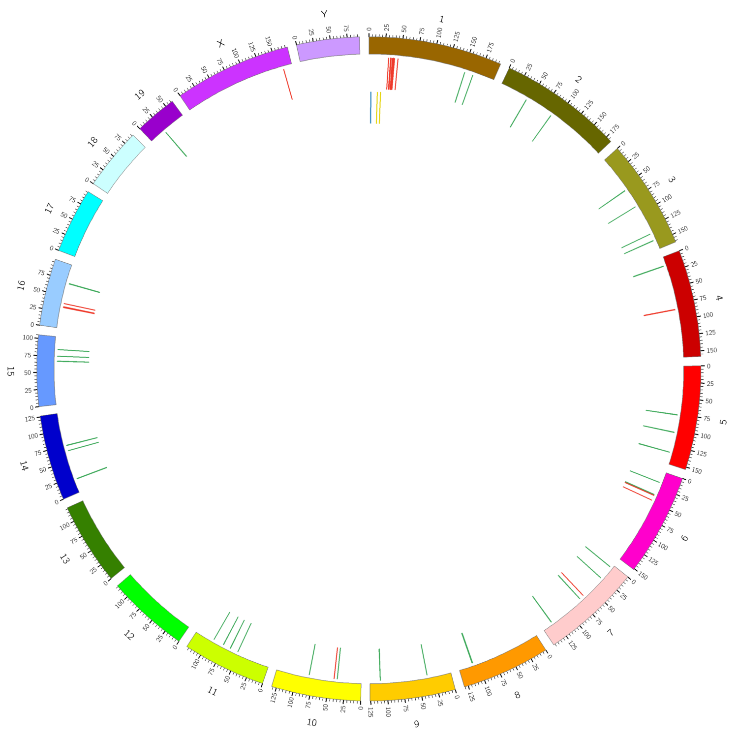
Analisi della posizione congiunta di mRNA e lncRNA – Grafico Circos (il cerchio centrale è l'mRNA e il cerchio interno è lncRNA)
Esplora i progressi facilitati dai servizi di sequenziamento lncRNA di BMKGene attraverso una raccolta curata di pubblicazioni.
Ji, H. et al. (2020) "Identificazione, previsione funzionale e verifica chiave dell'lncRNA degli lncRNA legati allo stress da freddo nel fegato dei ratti", Scientific Reports 2020 10:1, 10(1), pp. 1–14. doi: 10.1038/s41598-020-57451-7.
Jia, Z. et al. (2021) "L'analisi trascrittomica integrativa rivela il meccanismo immunitario per un ceppo di carpa comune resistente al CyHV-3", Frontiers in Immunology, 12, p. 687151.doi: 10.3389/FIMMU.2021.687151/BIBTEX.
Wang, XJ et al. (2022) "Priorità basata sull'integrazione multi-omica delle reti concorrenti di regolazione dell'RNA endogeno nel cancro del polmone a piccole cellule: caratteristiche molecolari e candidati farmaceutici", Frontiers in Oncology, 12, p. 904865. doi: 10.3389/FONC.2022.904865/BIBTEX.
Xiao, L. et al. (2020) "Dissezione genetica della rete di coespressione genetica alla base della fotosintesi in Populus", Plant Biotechnology Journal, 18(4), pp. 1015–1026. doi: 10.1111/PBI.13270.
Zheng, H. et al. (2022) "Una rete di regolamentazione globale per l'espressione genica disregolata e la segnalazione metabolica anomala nelle cellule immunitarie nel microambiente della malattia di Graves e della tiroidite di Hashimoto", Frontiers in Immunology, 13, p. 879824.doi: 10.3389/FIMMU.2022.879824/BIBTEX.