Assemblaggio del genoma a base di hi-C
Caratteristiche del servizio
● Sequenziamento su Illumina Novaseq con PE150.
● Il servizio richiede campioni di tessuto, anziché acidi nucleici estratti, per collegare con la formaldeide e conservare le interazioni del DNA-proteina.
● L'esperimento HI-C prevede la restrizione e la riparazione delle estremità delle estremità appiccicose con biotina, seguita dalla circolarizzazione delle estremità contundenti risultanti mentre si preserva le interazioni. Il DNA viene quindi tirato giù con perle di streptavidina e purificato per la successiva preparazione della biblioteca.
Vantaggi del servizio

Panoramica di Hi-C
(Lieberman-Aiden E et al.,Scienza, 2009)
●Eliminare la necessità di dati genetici della popolazione:HI-C sostituisce le informazioni essenziali richieste per l'ancoraggio di contig.
●Alta densità del marcatore:portando a un rapporto di ancoraggio ad alto contenuto di contig sopra il 90%.
●Registri di competenze e pubblicazioni estese:BMKGENE ha una vasta esperienza con oltre 2000 casi di assemblaggio del genoma hi-C da 1000 specie diverse e vari brevetti. Oltre 200 casi pubblicati hanno un fattore di impatto accumulativo di oltre 2000.
●Team di bioinformatica altamente qualificata:Con brevetti interni e diritti d'autore del software per esperimenti hi-C e analisi dei dati, il software di dati di visualizzazione auto-sviluppato consente di spostare, invertire, revocare e rifare a blocchi manuali.
●Supporto post-vendita:Il nostro impegno si estende oltre il completamento del progetto con un periodo di servizio post-vendita di 3 mesi. Durante questo periodo, offriamo un follow-up del progetto, assistenza per la risoluzione dei problemi e sessioni di domande e risposte per affrontare eventuali domande relative ai risultati.
●Annotazione completa: Utilizziamo più database per annotare funzionalmente i geni con variazioni identificate ed eseguire l'analisi di arricchimento corrispondente, fornendo approfondimenti su più progetti di ricerca.
Specifiche del servizio
| Preparazione della biblioteca | Strategia di sequenziamento | Output dei dati consigliato | Controllo di qualità |
| Biblioteca Hi-C. | Illumina Novaseq PE150 | 100x | Q30 ≥ 85% |
Requisiti del campione
| Tessuto | Importo richiesto |
| Animale viscer | ≥ 2 g |
| Muscolo animale | |
| Sangue di mammifero | ≥ 2 ml |
| Pollame/sangue di pesce | |
| Foglia fresca di pianta | ≥ 3 g |
| Cellule coltivate | ≥ 1x107 |
| Insetto | ≥ 2 g |
Flusso di lavoro di servizio

Design dell'esperimento

Consegna del campione

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Servizi post-vendita
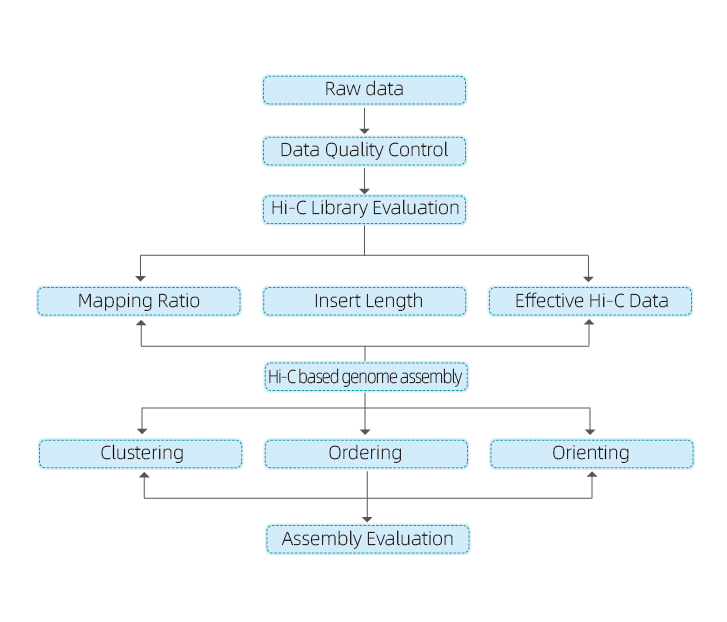
1) Dati grezzi QC
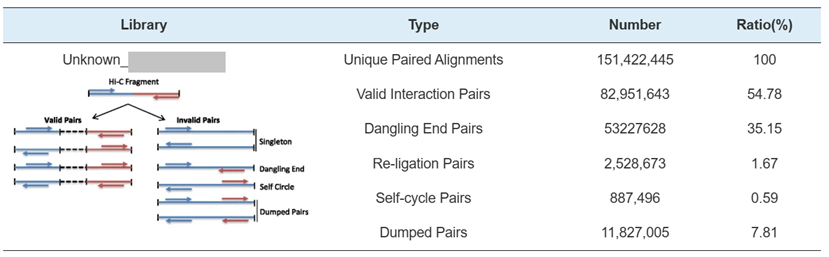
2) Biblioteca Hi-C QC: stima di interazioni Hi-C valide
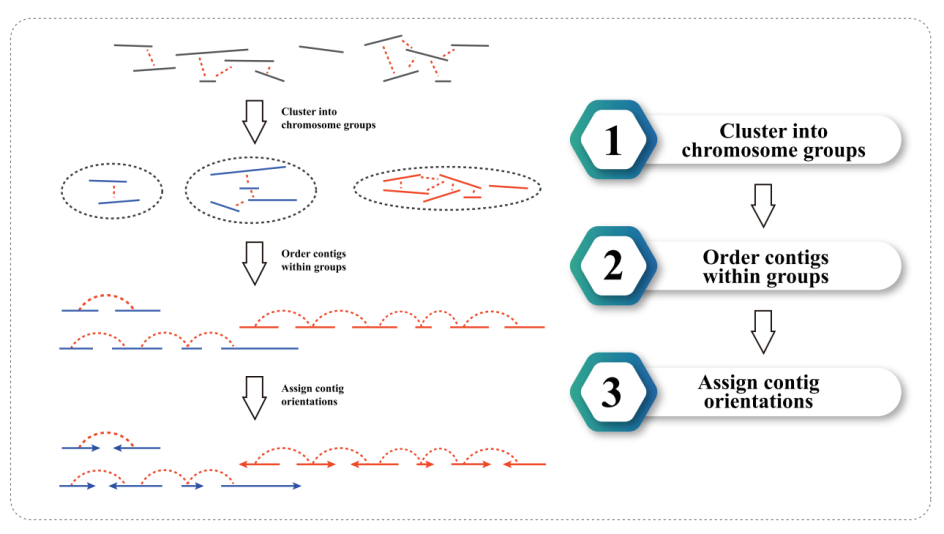
3) Assemblaggio hi-C: clustering di contigs in gruppi, seguito dall'ordinamento contig all'interno di ciascun gruppo e assegnazione dell'orientamento contig
4) Valutazione HI-C
Biblioteca Hi-C QC-stima di coppie di interazioni valide Hi-C
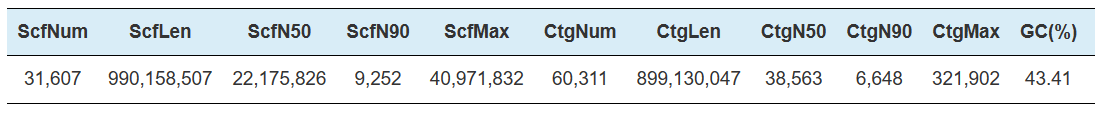
Assemblaggio Hi-C-Statistiche
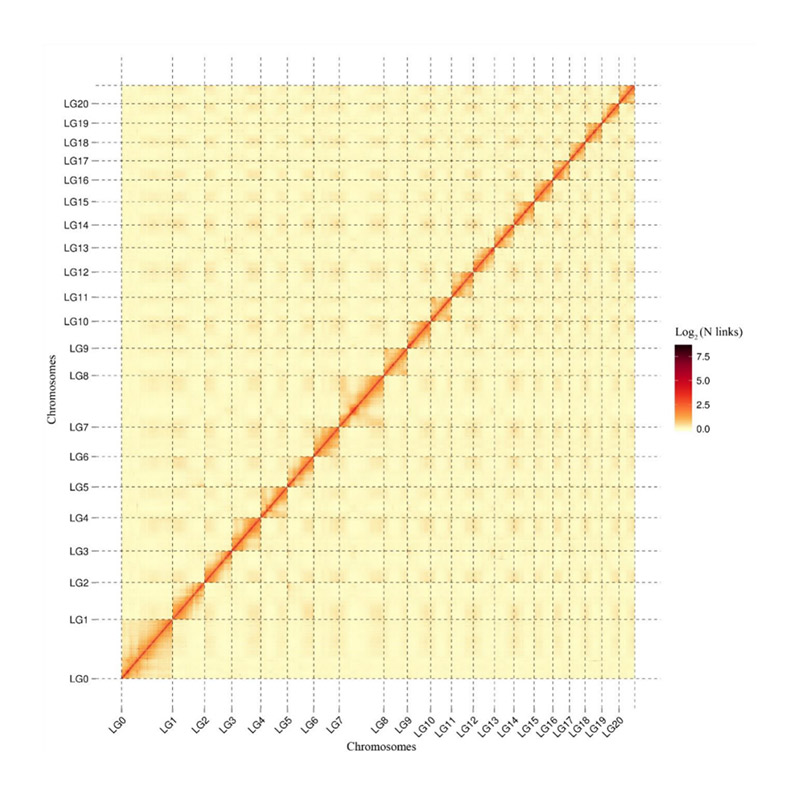
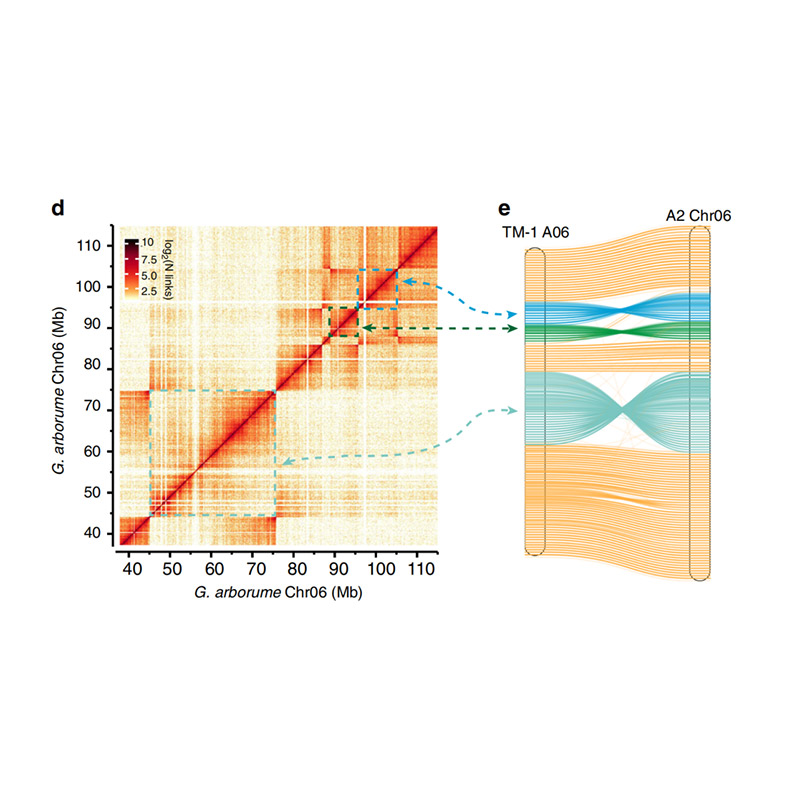
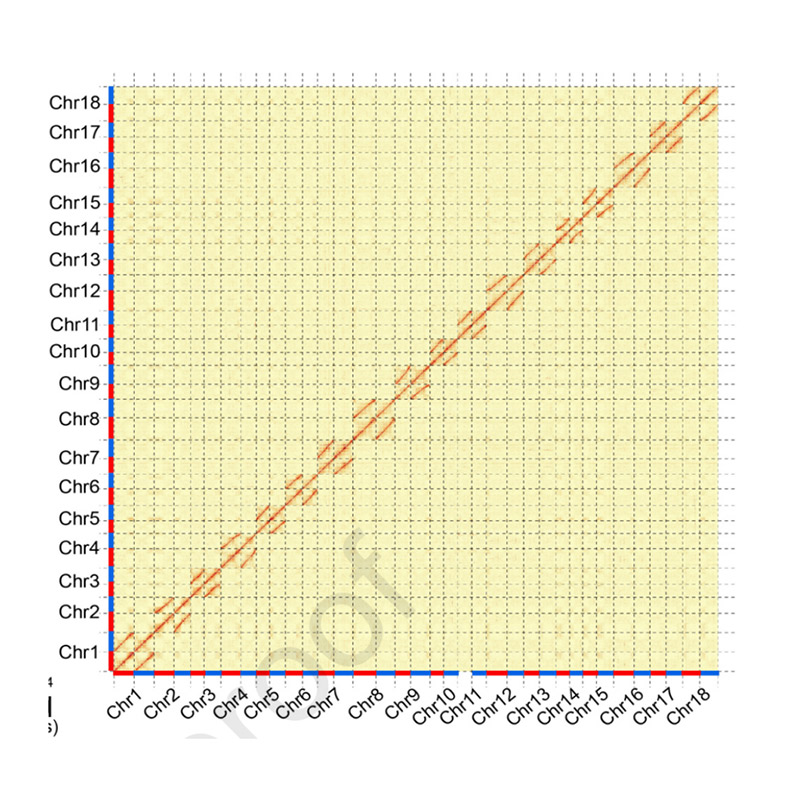
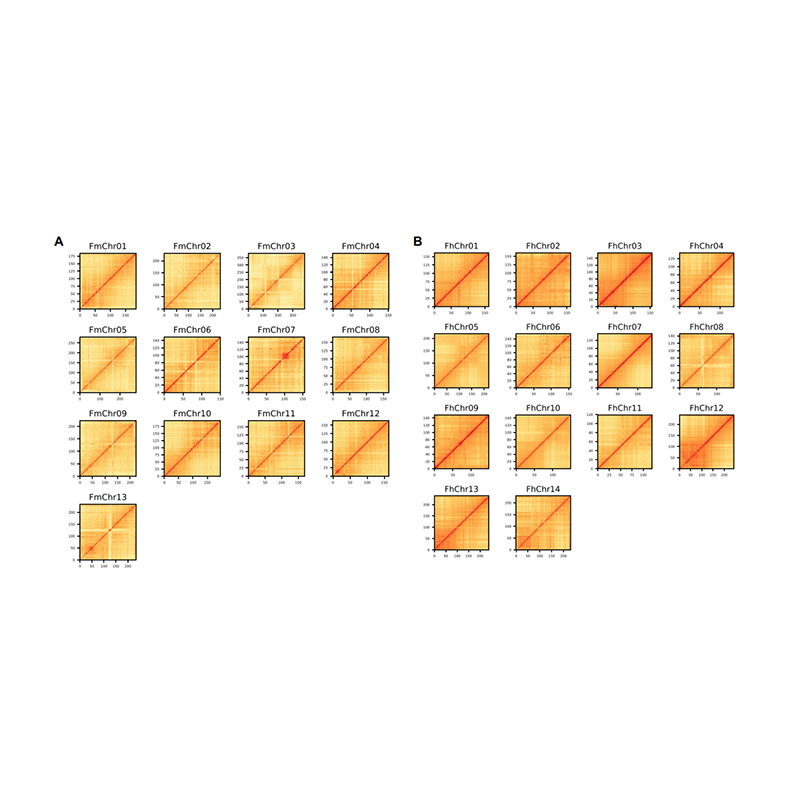
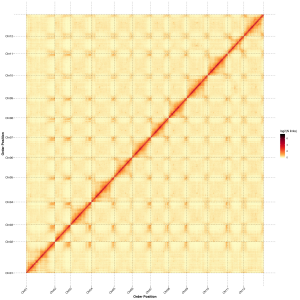
Valutazione post-assemblaggio-Mappa di calore dell'intensità del segnale tra i contenitori
Esplora i progressi facilitati dai servizi di assemblaggio hi-C di BMKGENE attraverso una raccolta curata di pubblicazioni.
Tian, T. et al. (2023) "Assemblaggio del genoma e dissezione genetica di un prominente germoplasma di mais resistente alla siccità", Nature Genetics 2023 55: 3, 55 (3), pp. 496–506. doi: 10.1038/s41588-023-01297-y.
Wang, Zl et al. (2020) "Un'assemblea su scala cromosomica del genoma Apis Cerana asiatico di abee Cerana", Frontiers in Genetics, 11, p. 524140. Doi: 10.3389/fGene.2020.00279/Bibtex.
Zhang, F. et al. (2023) "Rivelando l'evoluzione della biosintesi alcaloide tropana analizzando due genomi nella famiglia Solanaceae", Nature Communications 2023 14: 1, 14 (1), pagg. 1–18. doi: 10.1038/s41467-023-37133-4.
Zhang, X. et al. (2020) "I genomi dell'albero di Banyan e della vespa impollinatore forniscono approfondimenti sulla coevoluzione di FIG-WASP", Cell, 183 (4), pagg. 875-889.E17. doi: 10.1016/j.cell.2020.09.043