Interazione con cromatina basata su hi-C
Caratteristiche del servizio
● Sequenziamento su Illumina Novaseq con PE150.
● Il servizio richiede campioni di tessuto, anziché acidi nucleici estratti, per collegare con la formaldeide e conservare le interazioni del DNA-proteina.
● L'esperimento HI-C prevede la restrizione e la riparazione delle estremità delle estremità appiccicose con biotina, seguita dalla circolarizzazione delle estremità contundenti risultanti mentre si preserva le interazioni. Il DNA viene quindi tirato giù con perle di streptavidina e purificato per la successiva preparazione della biblioteca.
Vantaggi del servizio
●Design di enzimi di restrizione ottimale: per garantire un'elevata efficienza HI-C su specie diverse con coppie di interazioni valide fino al 93%.
●Registri di competenze e pubblicazioni estese:BMKGENE ha una vasta esperienza con> 2000 progetti di sequenziamento Hi-C di 800 specie diverse e vari brevetti. Oltre 100 casi pubblicati con un fattore di impatto accumulativo di oltre 900.
●Team di bioinformatica altamente qualificata:Con brevetti interni e diritti d'autore del software per esperimenti HI-C e analisi dei dati e un software di dati di visualizzazione auto-sviluppato.
●Supporto post-vendita:Il nostro impegno si estende oltre il completamento del progetto con un periodo di servizio post-vendita di 3 mesi. Durante questo periodo, offriamo un follow-up del progetto, assistenza per la risoluzione dei problemi e sessioni di domande e risposte per affrontare eventuali domande relative ai risultati.
●Annotazione completa: Utilizziamo più database per annotare funzionalmente i geni con variazioni identificate ed eseguire l'analisi di arricchimento corrispondente, fornendo approfondimenti su più progetti di ricerca.
Specifiche del servizio
| Biblioteca | Strategia di sequenziamento | Output dei dati consigliato | Risoluzione del segnale HI-C |
| Biblioteca Hi-C. | Illumina PE150 | Cromatina Loop: 150x TAD: 50x | Cromatina Loop: 10kb TAD: 40kb |
Requisiti di servizio
| Tipo di campionamento | Importo richiesto |
| Tessuto animale | ≥2g |
| Sangue intero | ≥2 ml |
| Funghi | ≥1G |
| Tissutale per piante | 1g/aliquota, 2-4 Aliquote consigliate |
| Cellule coltivate | ≥1x107 |
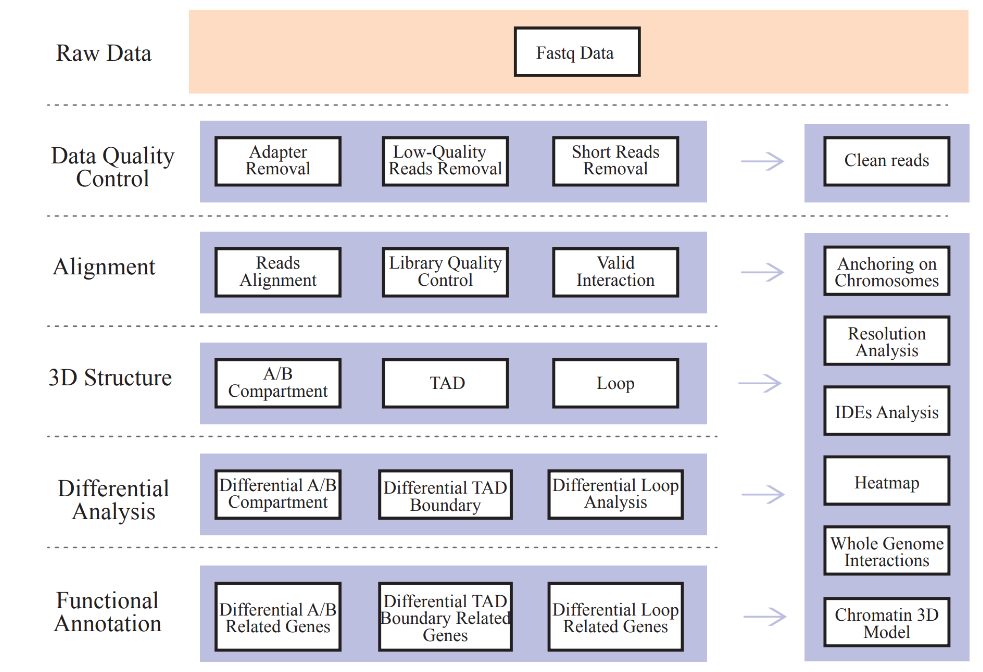
Include la seguente analisi:
● QC di dati grezzi;
● Mappatura e libreria HI-C QC: coppie di interazioni valide e esponenti di decadimento dell'interazione (IDES);
● Profilazione dell'interazione a livello del genoma: analisi CIS/trans e mappa di interazione HI-C;
● Analisi della distribuzione del compartimento A/B;
● Identificazione di TADS e anelli di cromatina;
● Analisi differenziale sugli elementi della struttura della cromatina 3D tra campioni e corrispondente annotazione funzionale dei geni associati.
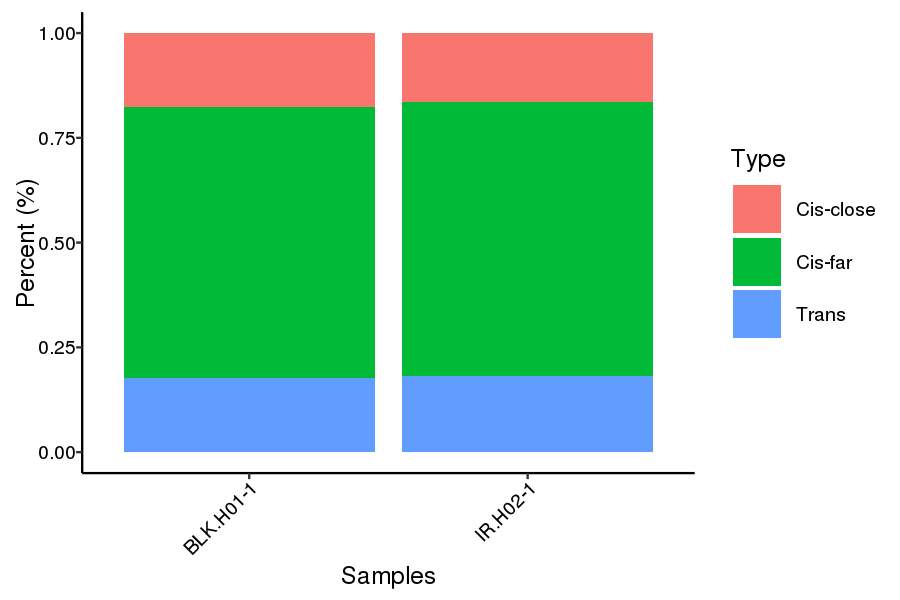
Distribuzione della proporzione cis e trans
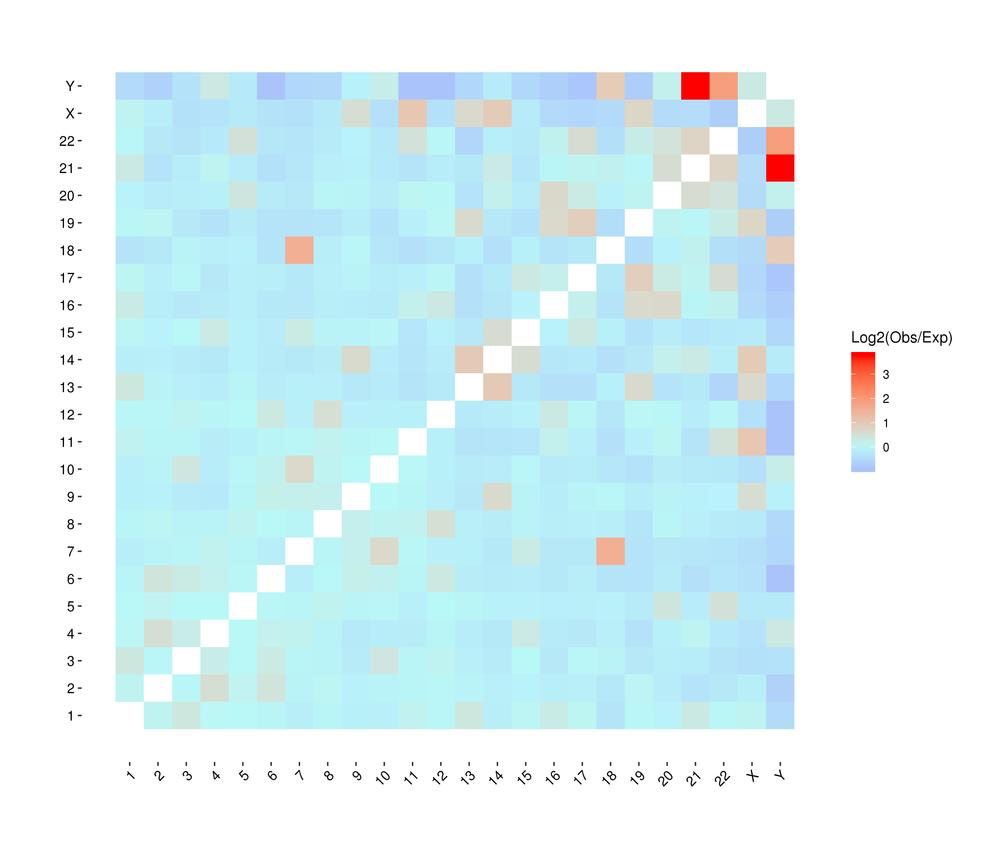
Mamap di calore delle interazioni cromosomiche tra i campioni
Distribuzione a livello del genoma di compartimenti A/B
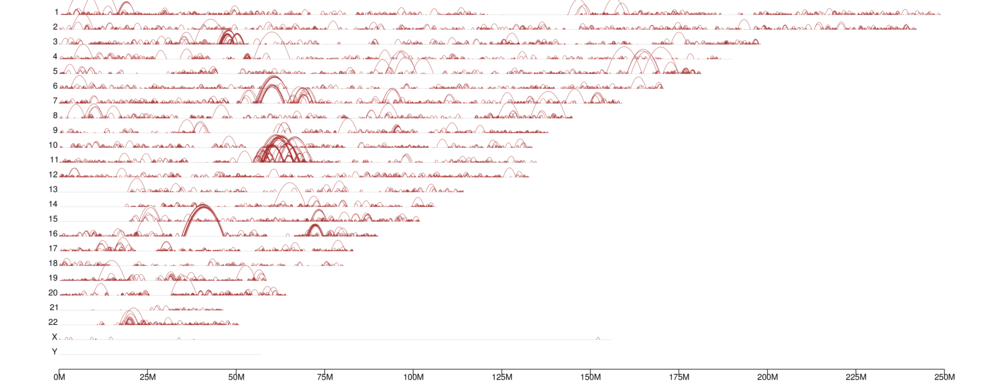
Distribuzione a livello del genoma dei loop di cromatina
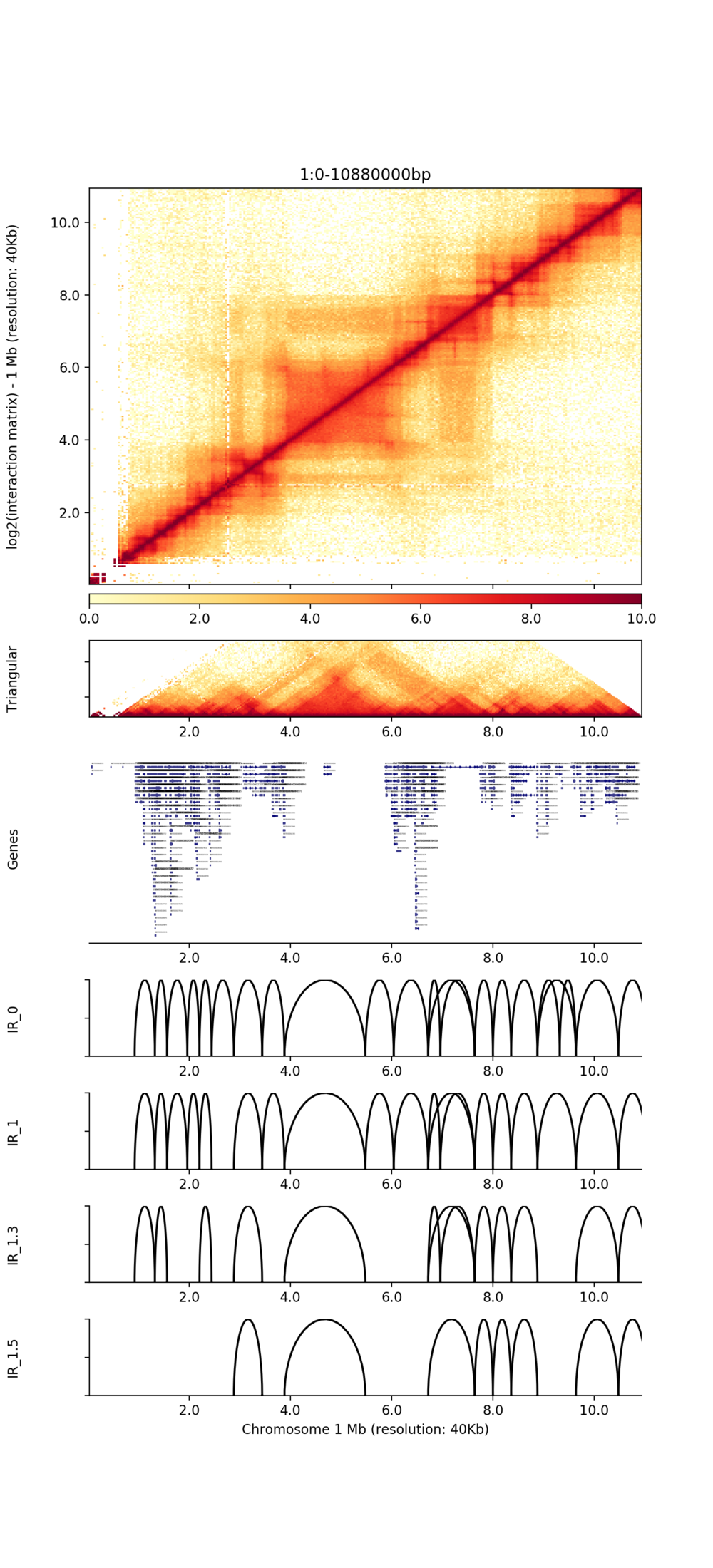
Visualizzazione di TADS
Esplora i progressi della ricerca facilitati dai servizi di sequenziamento Hi-C di BMKGENE attraverso una raccolta curata di pubblicazioni.
Meng, T. et al. (2021) "Un'analisi multi-omica integrata comparativa identifica CA2 come un nuovo bersaglio per il coordoma",Neuro-oncologia, 23 (10), pagg. 1709–1722. doi: 10.1093/neuonc/noab156.
Xu, L. et al. (2021) "La disorganizzazione 3D e il riarrangiamento del genoma forniscono approfondimenti sulla patogenesi di NAFLD mediante sequenziamento hi-C, nanoporo e RNA integrato",Acta Pharmaceutica Sinica B, 11 (10), pagg. 3150–3164. doi: 10.1016/j.apsb.2021.03.022.