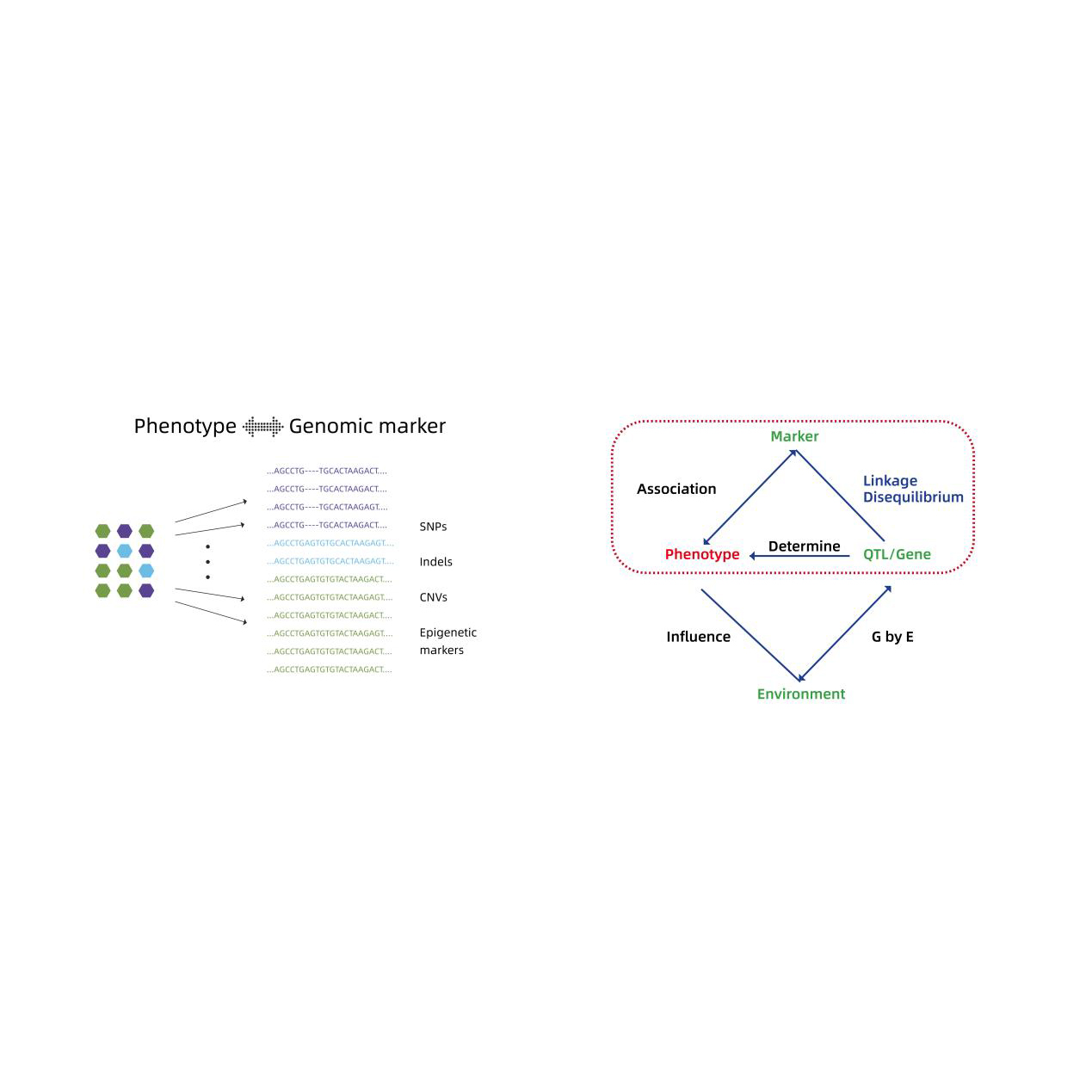
Analisi di associazione su tutto il genoma
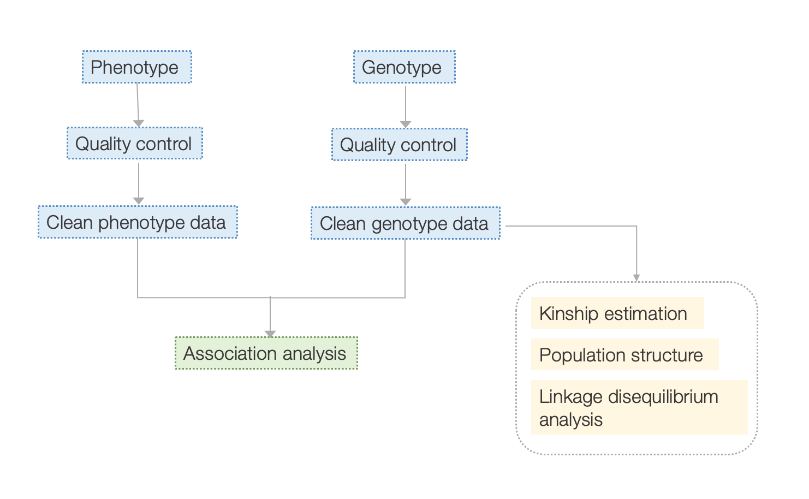
Flusso di lavoro

Vantaggi del servizio
●Vasta esperienza e record di pubblicazioni: con l'esperienza accumulata nel GWAS, BMKGene ha completato centinaia di progetti sulle specie nella ricerca GWAS sulla popolazione, ha assistito i ricercatori nella pubblicazione di più di 100 articoli e il fattore di impatto cumulativo ha raggiunto 500.
● Analisi bioinformatica completa: il flusso di lavoro include l'analisi dell'associazione dei tratti SNP, fornendo una serie di geni candidati e la loro corrispondente annotazione funzionale.
●Team bioinformatico altamente qualificato e ciclo di analisi breve: con una grande esperienza nell'analisi genomica avanzata, il team di BMKGene fornisce analisi complete con tempi di consegna rapidi.
●Supporto post-vendita:Il nostro impegno si estende oltre il completamento del progetto con un periodo di servizio post-vendita di 3 mesi. Durante questo periodo, offriamo follow-up del progetto, assistenza per la risoluzione dei problemi e sessioni di domande e risposte per rispondere a qualsiasi domanda relativa ai risultati.
Specifiche e requisiti del servizio
| Tipo di sequenziamento | Scala di popolazione consigliata | Strategia di sequenziamento | Requisiti nucleotidici |
| Sequenziamento dell'intero genoma | 200 campioni | 10x | Concentrazione: ≥ 1 ng/μL Importo totale ≥ 30 ng Degrado o contaminazione limitati o assenti |
| Frammento amplificato con locus specifico (SLAF) | Profondità etichetta: 10x Numero di tag: < 400 Mb: si consiglia WGS < 1 Gb: 100.000 tag 1 GB > 2Gb: 300.000 tag Massimo 500.000 tag | Concentrazione ≥ 5 ng/μL Quantità totale ≥ 80 ng Nanogoccia OD260/280=1,6-2,5 Gel di agarosio: degradazione o contaminazione assente o limitata
|
Selezione dei materiali



Diverse varietà, sottospecie, varietà autoctone/banche genetiche/famiglie miste/risorse selvatiche
Diverse varietà, sottospecie, varietà autoctone
Famiglia di fratellastri/famiglia di fratelli pieni/risorse selvagge
Flusso di lavoro del servizio

Progettazione dell'esperimento

Consegna del campione

Estrazione dell'RNA

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Servizi post-vendita
Include la seguente analisi:
- Analisi di associazione sull'intero genoma: modello LM, LMM, EMMAX, FASTLMM
- Annotazione funzionale dei geni candidati
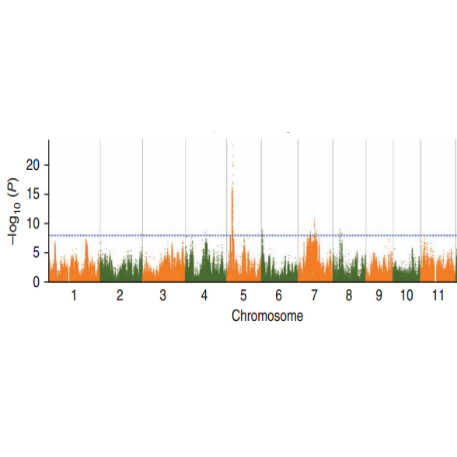
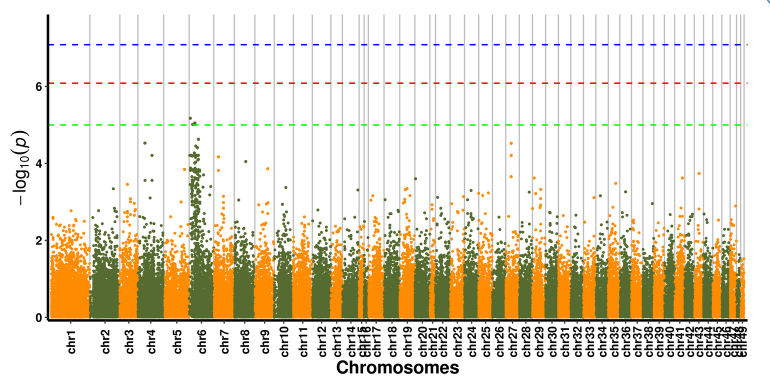
Analisi dell'associazione dei tratti SNP – diagramma di Manhattan
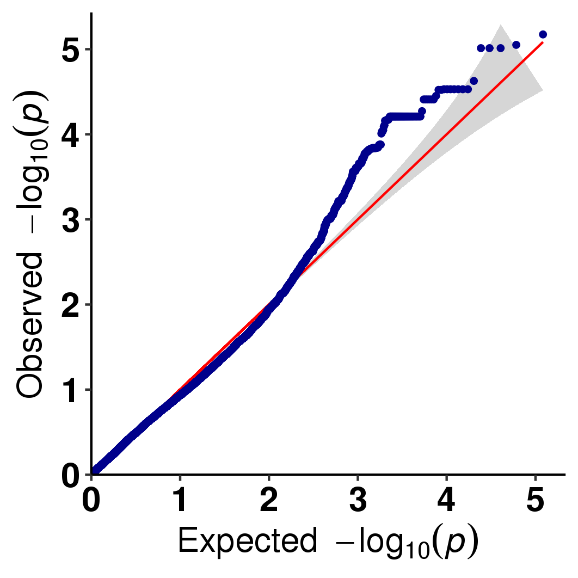
Analisi dell'associazione dei tratti SNP - grafico QQ
Esplora i progressi facilitati dai servizi de GWAS di BMKGene attraverso una raccolta curata di pubblicazioni:
Lv, L. et al. (2023) "Approfondimento sulle basi genetiche della tolleranza all'ammoniaca nei cannolicchi Sinonovacula constricta mediante uno studio di associazione sull'intero genoma",Acquacoltura, 569, pag. 739351. doi: 10.1016/J.AQUACULTURE.2023.739351.
Li, X. et al. (2022) "Le analisi multi-omiche di 398 accessioni di miglio di coda di volpe rivelano regioni genomiche associate alla domesticazione, tratti metabolitici ed effetti antinfiammatori",Pianta molecolare, 15(8), pp. 1367–1383. doi: 10.1016/j.molp.2022.07.003.
Li, J. et al. (2022) "Mappatura dell'associazione a livello genomico di fenotipi appena senza scafo in ambienti siccitosi",Frontiere nella scienza delle piante, 13, pag. 924892.doi: 10.3389/FPLS.2022.924892/BIBTEX.
Zhao, X. et al. (2021) "GmST1, che codifica per una sulfotransferasi, conferisce resistenza ai ceppi G2 e G3 del virus del mosaico della soia",Pianta, cellula e ambiente, 44(8), pp. 2777–2792. doi: 10.1111/PCE.14066.