Assemblea del genoma fungino de novo
Caratteristiche del servizio
Con tre possibili opzioni tra cui scegliere a seconda del grado desiderato di completezza del genoma:
● Draft Genome Option: sequenziamento a lettura corta con Illumina Novaseq PE150.
● Opzione del genoma sottile fungino:
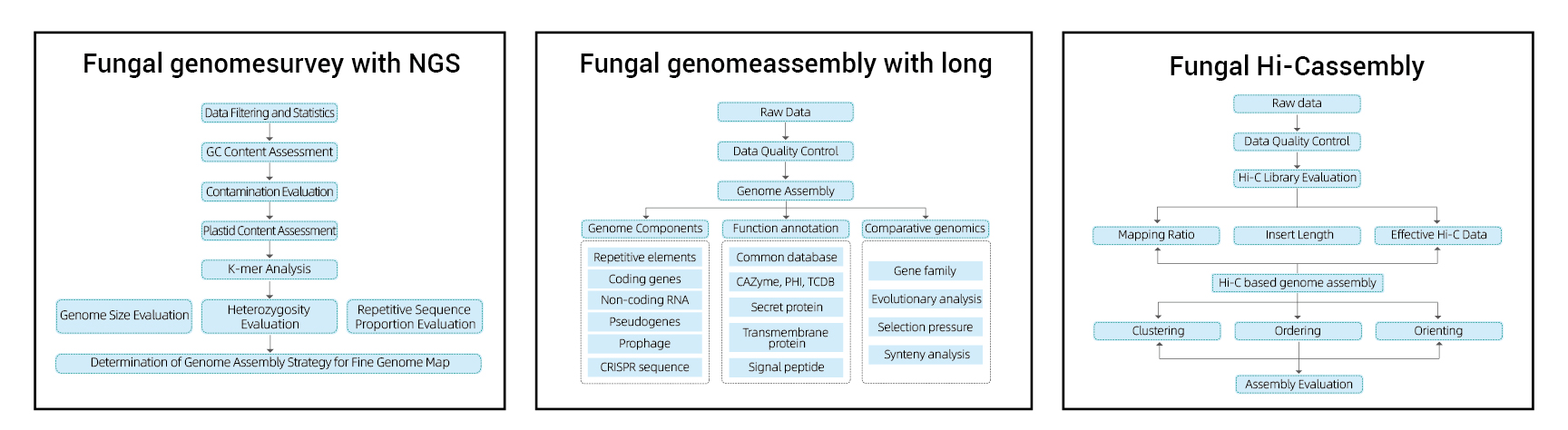
Genome Survey: Illumina Novaseq PE150.
Assemblea del genoma: Pacbio Revio (HIFI legge) o Nanopore Prometion 48.
● Genoma fungino a livello di cromosoma:
Genome Survey: Illumina Novaseq PE150.
Assemblea del genoma: Pacbio Revio (HIFI legge) o Nanopore Prometion 48.
Contigare l'ancoraggio con l'assemblaggio hi-C.
Vantaggi del servizio
●Strategie di sequenziamento multipli disponibili: Per diversi obiettivi di ricerca e requisiti di completezza del genoma
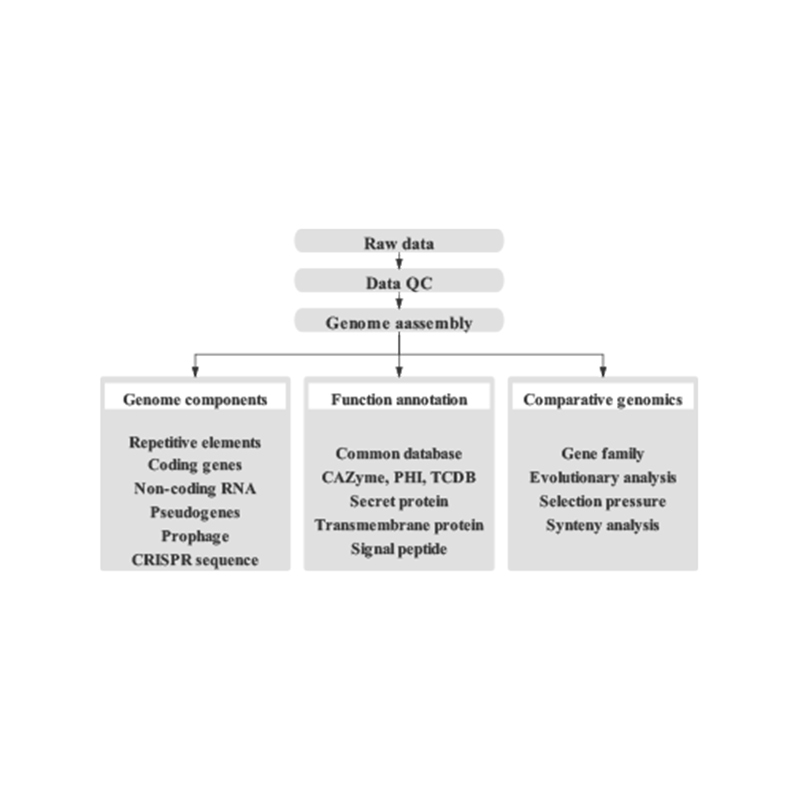
●Flusso di lavoro bioinformatico completo:Ciò include l'assemblaggio del genoma e la previsione di più elementi genomici, annotazione del gene funzionale e ancoraggio di contig.
●Vasta competenza: Con oltre 12.000 genomi microbici assemblati, portiamo oltre un decennio di esperienza, un team di analisi altamente qualificato, contenuti completi e un eccellente supporto post-vendita.
●Supporto post-vendita:Il nostro impegno si estende oltre il completamento del progetto con un periodo di servizio post-vendita di 3 mesi. Durante questo periodo, offriamo un follow-up del progetto, assistenza per la risoluzione dei problemi e sessioni di domande e risposte per affrontare eventuali domande relative ai risultati.
Specifiche del servizio
| Servizio | Strategia di sequenziamento | Controllo di qualità |
| Progetto del genoma | Illumina PE150 100x | Q30≥85% |
| Genoma fine | Genome Survey: Illumina PE150 50 x Assemblaggio: Pacbio Hifi 30x o Nanopore 100x | contig N50 ≥1 MB (PacBio Unicellulare) contig N50 ≥2 MB (ONT Unicellulare) Contig N50 ≥500kb (Altri) |
| Genoma a livello di cromosoma | Genome Survey: Illumina PE150 50 x Assemblaggio: Pacbio Hifi 30x o Nanopore 100x Assemblaggio hi-C 100x | CONTIG CORSAZIONE PORO> 90%
|
Requisiti di servizio
| Concentrazione (ng/µl) | Importo totale (µg) | Volume (µL) | OD260/280 | OD260/230 | |
| Pacbio | ≥20 | ≥2 | ≥20 | 1.7-2.2 | ≥1.6 |
| Nanopore | ≥40 | ≥2 | ≥20 | 1.7-2.2 | 1.0-3.0 |
| Illumina | ≥1 | ≥0,06 | ≥20 | - | - |
Fungo unicellulare: ≥3,5x1010 cellule
MACRO FUNGUS: ≥10 g
Flusso di lavoro di servizio

Consegna del campione

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Servizi post-vendita
Include la seguente analisi:
Genome Survey:
- Controllo della qualità dei dati di sequenziamento
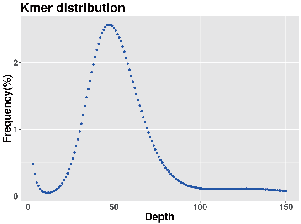
- Stima del genoma: dimensioni, eterozigosi, elementi ripetitivi
Assemblea del genoma fine:
- Controllo della qualità dei dati di sequenziamento
- De novoAssemblaggio
- Analisi dei componenti del genoma: previsione di CD e elementi genomici multipli
- Annotazione funzionale con database più generali (GO, KEGG, ecc.) E database avanzati (scheda, VFDB, ecc.)
Assemblaggio Hi-C:
- Valutazione della biblioteca Hi-C.
- Contigs Anchoring Clustering, Ordering and Oriening
- Valutazione dell'assemblaggio hi-C: in base al genoma di riferimento e alla mappa di calore
Genome Survey: K-Mer Distribution
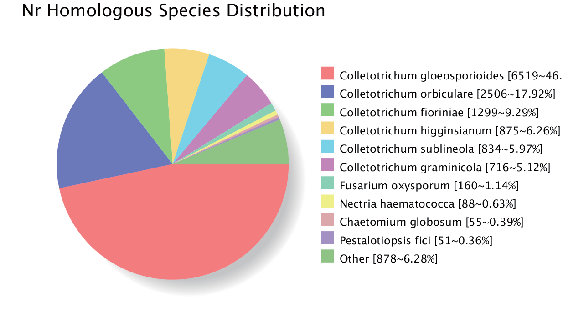
Assemblea del genoma: annotazione omologa genica (database NR)
Assemblaggio del genoma: annotazione del gene funzionale (GO)
Esplora i progressi facilitati dai servizi di assemblaggio del genoma fungino di BMKGENE attraverso una raccolta curata di pubblicazioni.
Hao, J. et al. (2023) "profilazione omic integrata del fungo medicinale inonotus obliquus in condizioni sommerse",Genomica BMC, 24 (1), pagg. 1–12. doi: 10.1186/s12864-023-09656-z/figure/3.
Lu, L. et al. (2023) "Il sequenziamento del genoma rivela l'evoluzione e i meccanismi patogeni del patogeno patogeno di grano del grano Rhizoctonia cerealis",Il diario del raccolto, 11 (2), pagg. 405–416. doi: 10.1016/j.cj.2022.07.024.
Zhang, H. et al. (2023) "Risorse del genoma per quattro specie di Clariredia che causano un punto in dollari su diversi errori",Malattia vegetale, 107 (3), pagg. 929-934. doi: 10.1094/pdis-08-22-1921-a
Zhang, SS et al. (2023) "evidenza genetica e molecolare di un sistema di accoppiamento tetrapolare nella grifola frondosa di funghi commestibili",Journal of Fungi, 9 (10), p. 959. Doi: 10.3390/jof9100959/s1.