Nanoporo per sequenziamento dell'mRNA a lunghezza intera
Caratteristiche
● Cattura dell'mRNA poli-A seguita dalla sintesi del cDNA e dalla preparazione della libreria
● Sequenziamento delle trascrizioni integrali
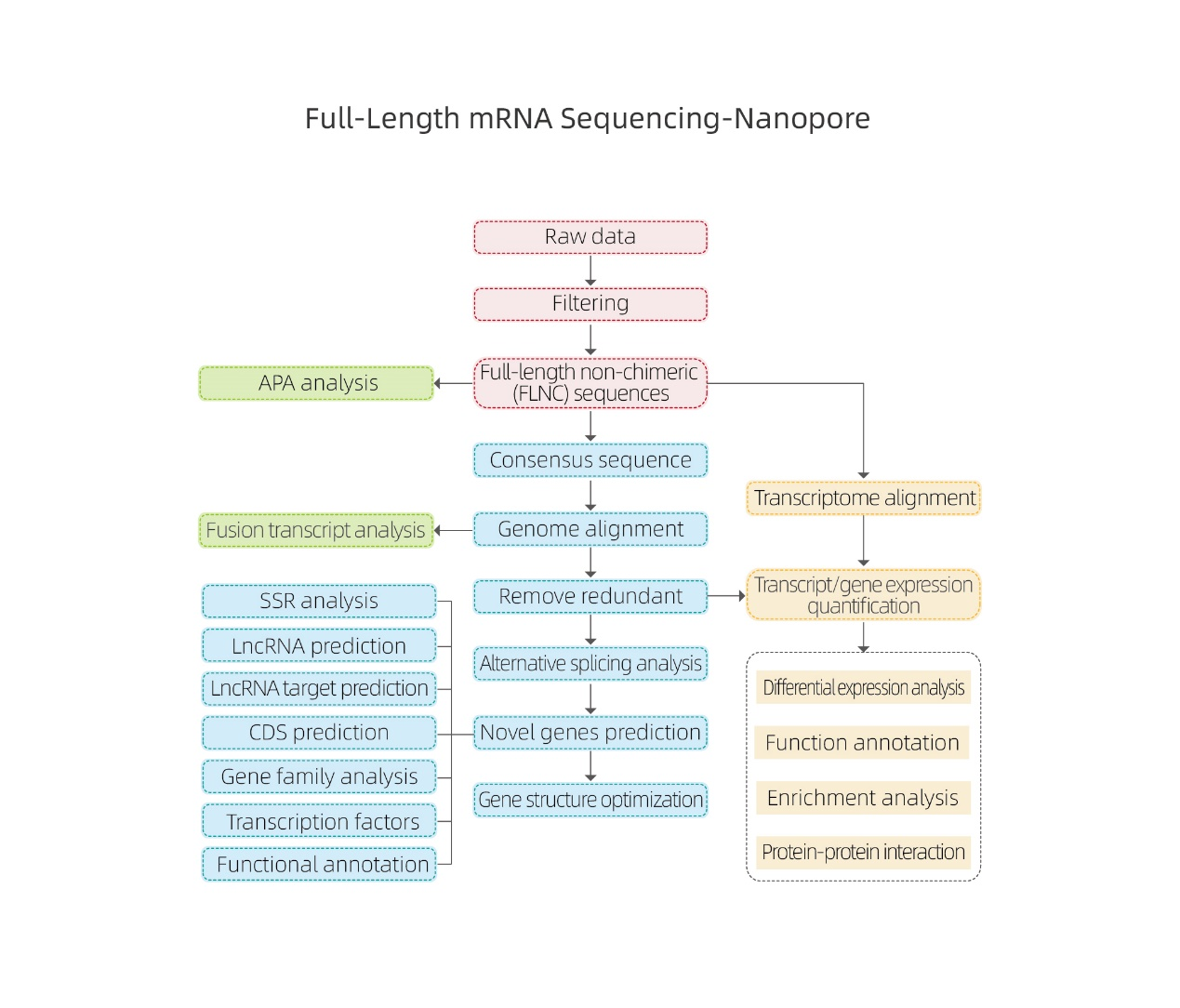
● Analisi bioinformatica basata sull'allineamento ad un genoma di riferimento
● L'analisi bioinformatica comprende non solo l'espressione a livello di geni e isoforme, ma anche l'analisi di lncRNA, fusioni geniche, poliadenilazione e struttura genica
Vantaggi del servizio
●Quantificazione dell'espressione a livello di isoforme: consentendo un'analisi dell'espressione dettagliata e accurata, svelando il cambiamento che potrebbe essere mascherato durante l'analisi dell'intera espressione genetica
●Richieste di dati ridotte:Rispetto al sequenziamento di nuova generazione (NGS), il sequenziamento dei nanopori presenta requisiti di dati inferiori, consentendo livelli equivalenti di saturazione della quantificazione dell'espressione genica con dati più piccoli.
●Maggiore accuratezza della quantificazione dell'espressione: sia a livello di geni che di isoforme
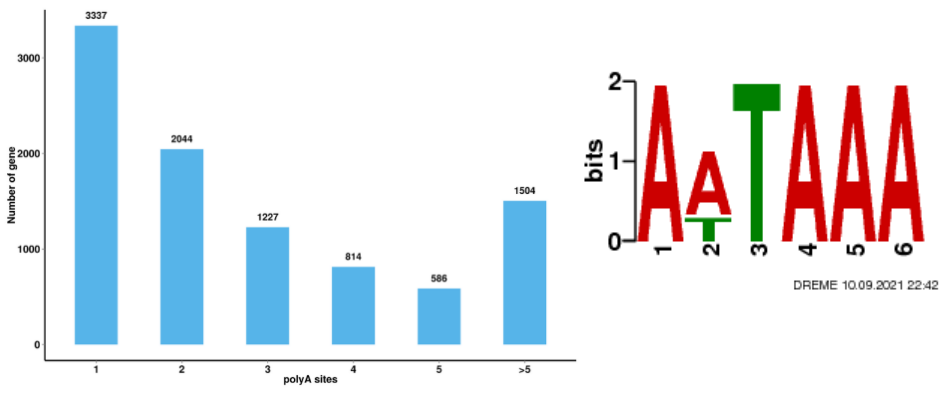
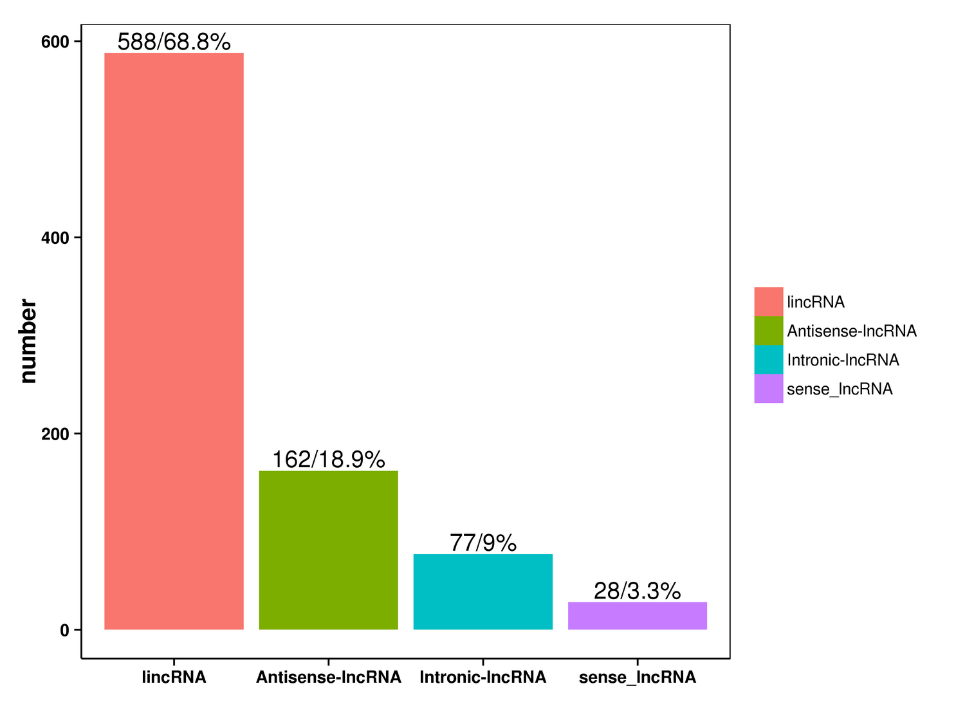
●Identificazione di informazioni trascrittomiche aggiuntive: poliadenilazione alternativa, geni di fusione e lcnRNA e loro geni bersaglio
●Ampia competenza: Il nostro team apporta una vasta esperienza a ogni progetto, avendo completato oltre 850 progetti di trascrittoma integrale Nanopore ed elaborato oltre 8.000 campioni.
●Supporto post-vendita: il nostro impegno va oltre il completamento del progetto con un periodo di servizio post-vendita di 3 mesi. Durante questo periodo, offriamo follow-up del progetto, assistenza per la risoluzione dei problemi e sessioni di domande e risposte per rispondere a qualsiasi domanda relativa ai risultati.
Requisiti e consegna del campione
| Biblioteca | Strategia di sequenziamento | Dati consigliati | Controllo di qualità |
| Poli A arricchito | Illumina PE150 | 6/12 GB | Punteggio di qualità medio: Q10 |
Requisiti del campione:
Nucleotidi:
| Concentrazione (ng/μl) | Quantità (μg) | Purezza | Integrità |
| ≥ 100 | ≥ 1,0 | DE260/280=1,7-2,5 DE260/230=0,5-2,5 Contaminazione limitata o assente di proteine o DNA mostrata sul gel. | Per gli impianti: RIN≥7,0; Per gli animali: RIN≥7,5; 5,0≥28S/18S≥1,0; elevazione della linea di base limitata o assente |
● Piante:
Radice, stelo o petalo: 450 mg
Foglia o seme: 300 mg
Frutta: 1,2 g
● Animale:
CUORE o Intestino: 300 mg
Visceri o cervello: 240 mg
Muscolo: 450 mg
Ossa, capelli o pelle: 1 g
● Artropodi:
Insetti: 6g
Crostacei: 300 mg
● Sangue intero: 1 tubo
● Celle: 106 cellule
Consegna del campione consigliata
Contenitore: provetta da centrifuga da 2 ml (la carta stagnola non è consigliata)
Etichettatura del campione: Gruppo+replica, ad esempio A1, A2, A3; B1, B2, B3.
Spedizione:
1. Ghiaccio secco: i campioni devono essere confezionati in sacchetti e sepolti nel ghiaccio secco.
2. Provette RNAstable: i campioni di RNA possono essere essiccati in provette di stabilizzazione dell'RNA (ad esempio RNAstable®) e spediti a temperatura ambiente.
Flusso di lavoro del servizio
Nucleotidi:

Consegna del campione

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Servizi post-vendita
Flusso di lavoro del servizio
Tessuto:

Progettazione dell'esperimento

Consegna del campione

Estrazione dell'RNA

Costruzione della biblioteca

Sequenziamento

Analisi dei dati

Servizi post-vendita
● Elaborazione dei dati grezzi
● Identificazione della trascrizione
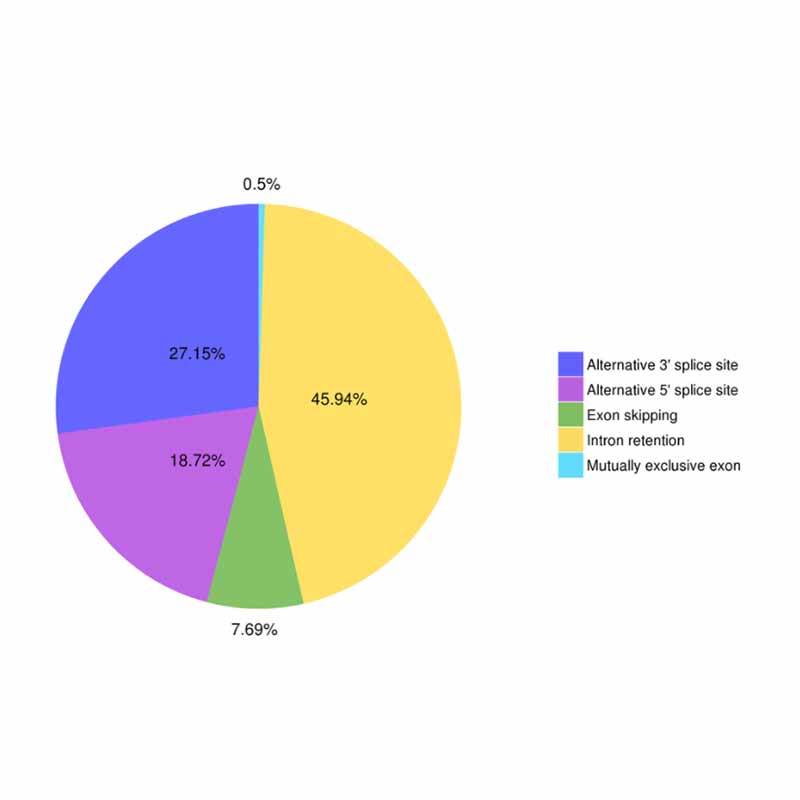
● Giunzioni alternative
● Quantificazione dell'espressione a livello genico e isoforme
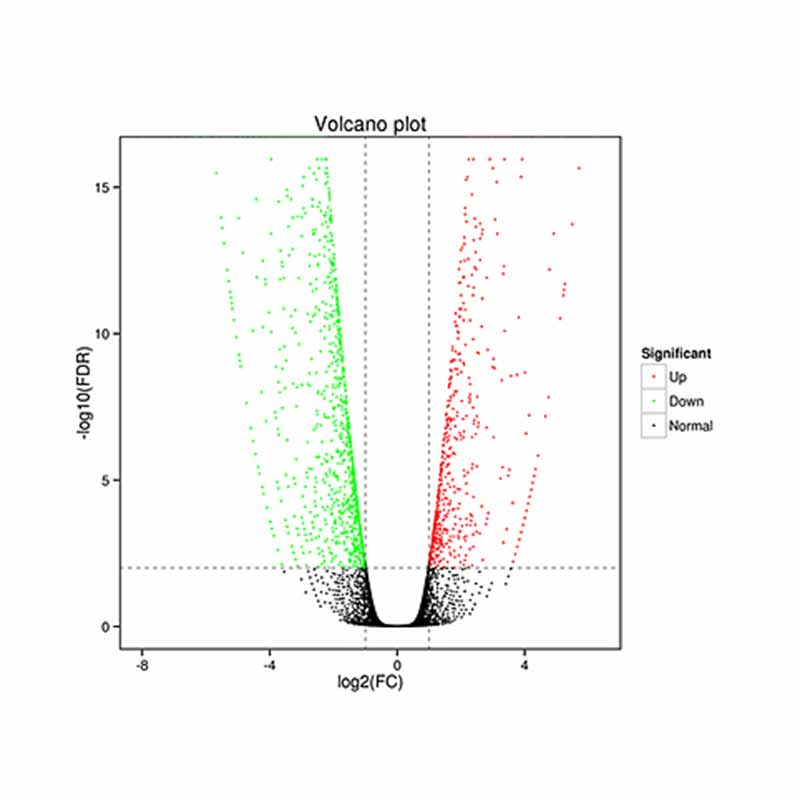
● Analisi dell'espressione differenziale
● Annotazione e arricchimento di funzioni (DEG e DET)
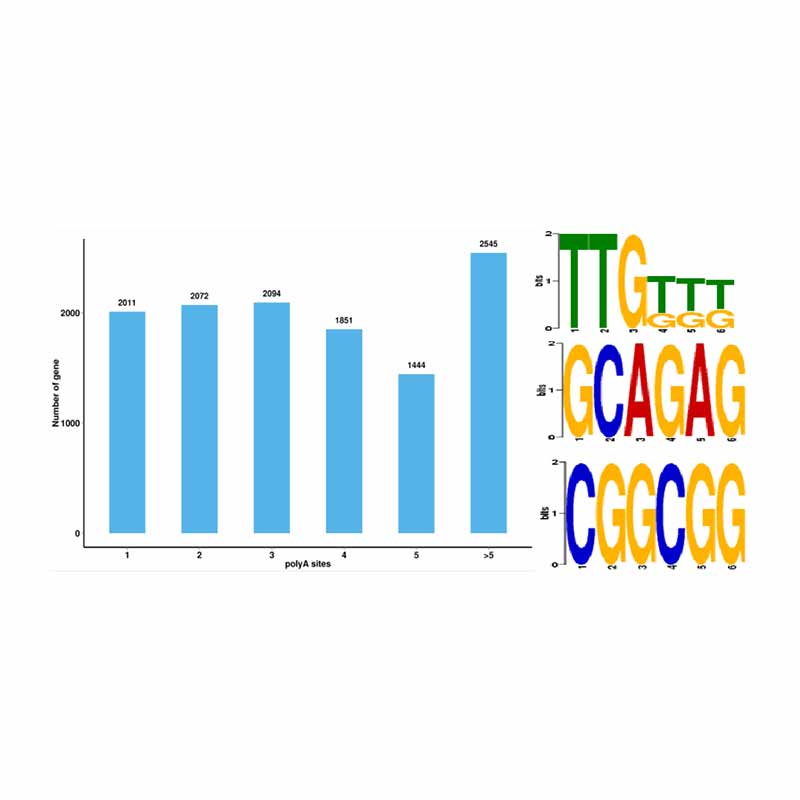
Analisi di splicing alternativo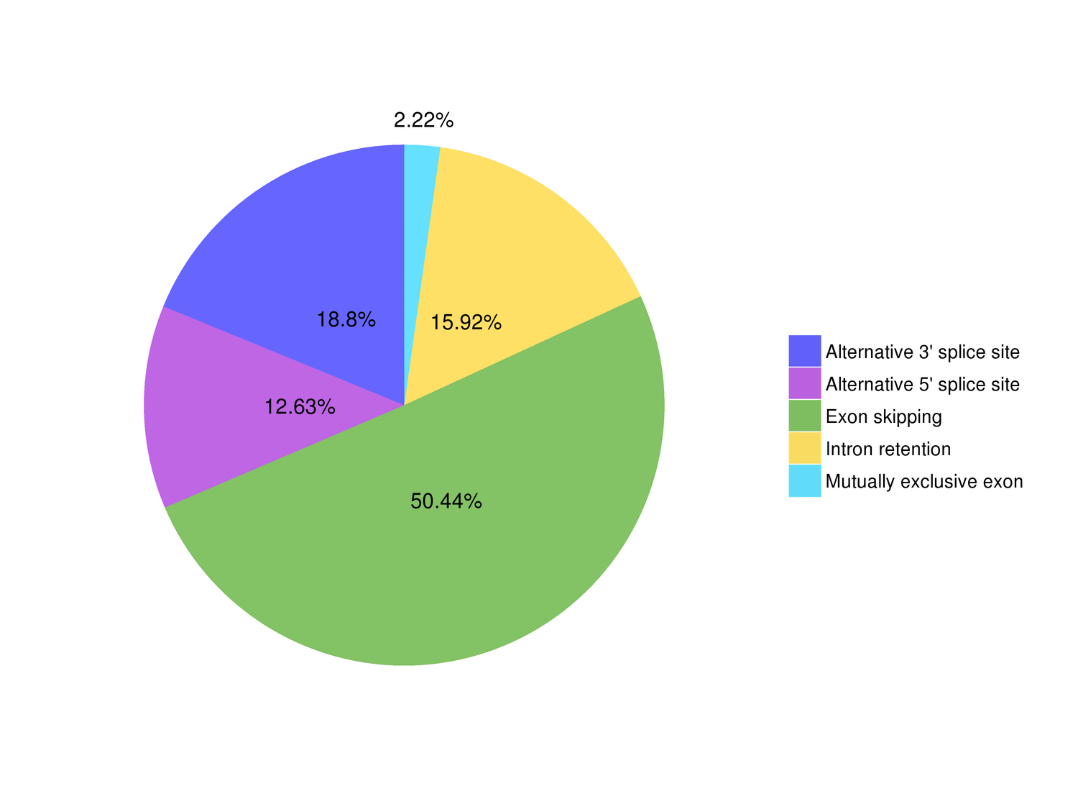 Analisi alternativa della poliadenilazione (APA)
Analisi alternativa della poliadenilazione (APA)
Previsione dell'lncRNA
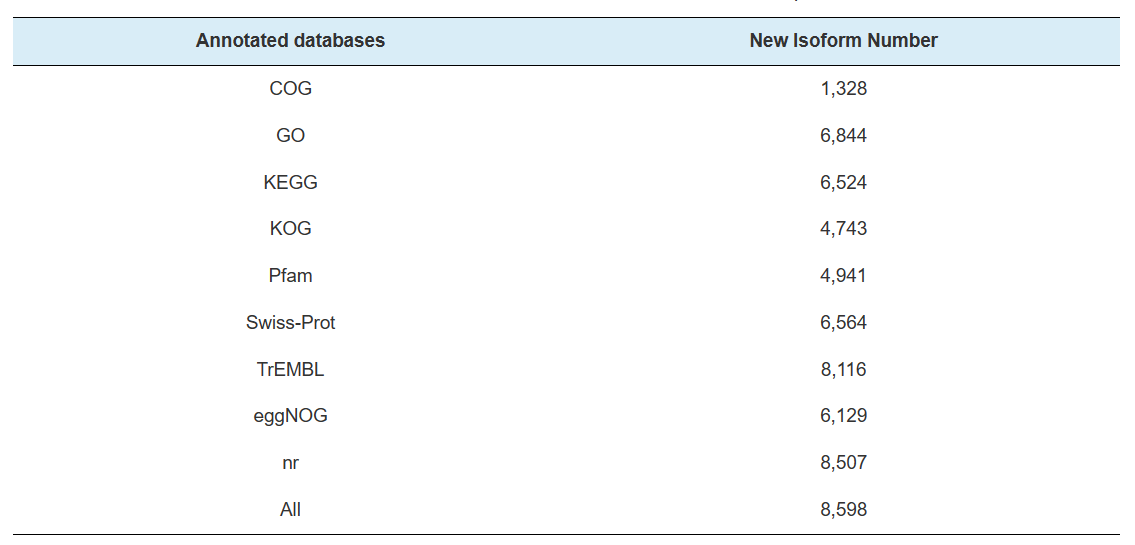
Annotazione di nuovi geni
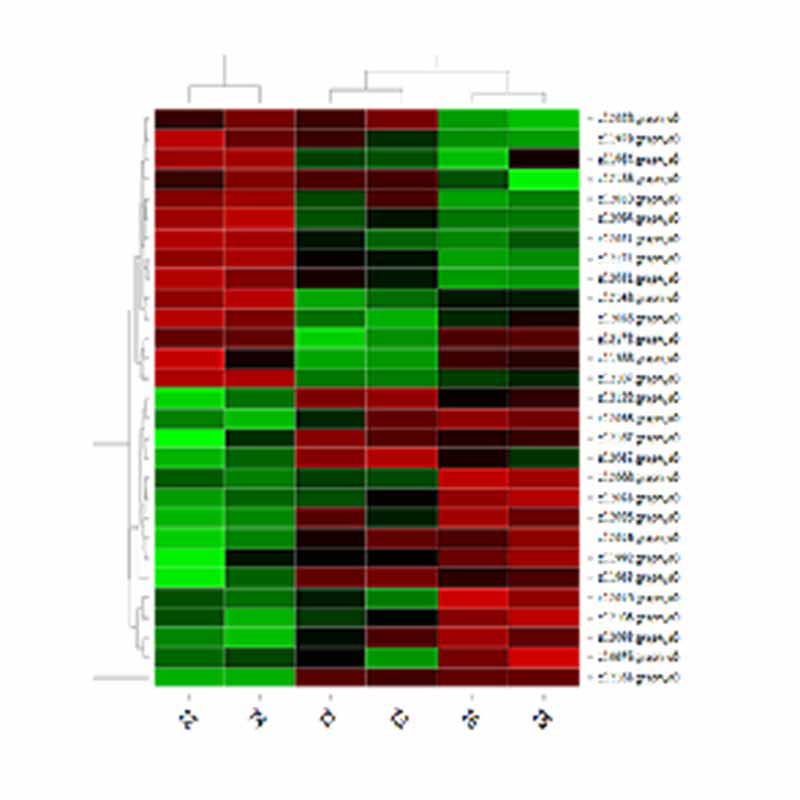
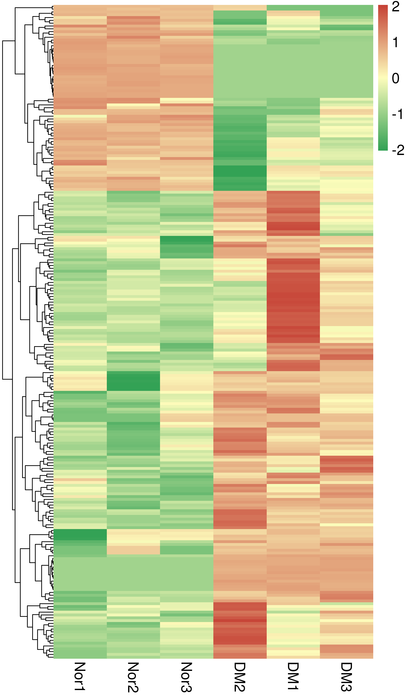
Clustering di DET
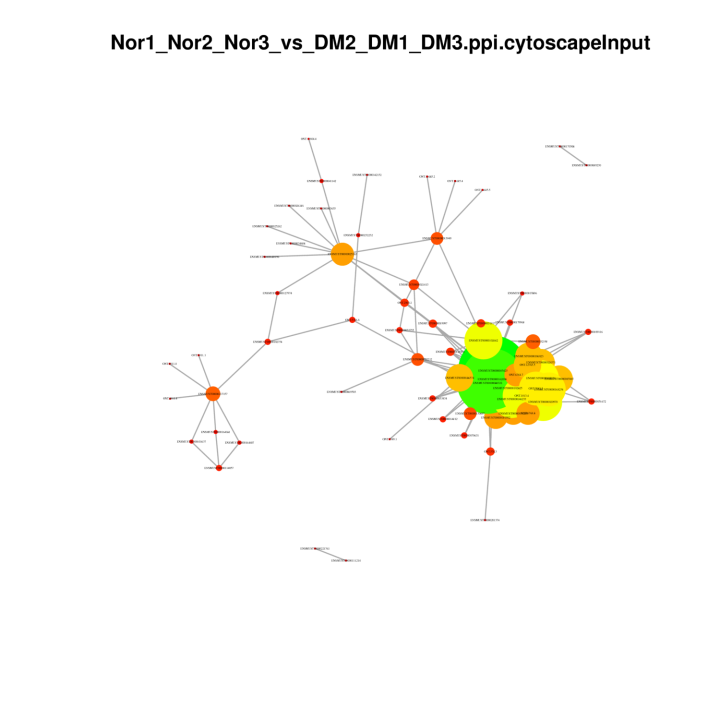
Reti proteina-proteina nei DEG
Esplora i progressi facilitati dai servizi di sequenziamento completo di mRNA Nanopore di BMKGene attraverso una raccolta curata di pubblicazioni.
Gong, B. et al. (2023) "Attivazione epigenetica e trascrizionale della chinasi secretoria FAM20C come oncogene nel glioma", Journal of Genetics and Genomics, 50(6), pp. 422–433. doi: 10.1016/J.JGG.2023.01.008.
Lui, Z. et al. (2023) "Il sequenziamento del trascrittoma a lunghezza intera dei linfociti rispondono all'IFN-γ rivela una risposta immunitaria distorta da Th1 nella passera di mare (Paralichthys olivaceus)", Fish & Shellfish Immunology, 134, p. 108636.doi: 10.1016/J.FSI.2023.108636.
Ma, Y. et al. (2023) "Analisi comparativa dei metodi di sequenziamento dell'RNA PacBio e ONT per l'identificazione del veleno di Nemopilema Nomurai", Genomics, 115(6), p. 110709.doi: 10.1016/J.YGENO.2023.110709.
Yu, D. et al. (2023) "L'analisi nano-seq rivela una diversa tendenza funzionale tra esosomi e microvescicole derivate da hUMSC", Stem Cell Research and Therapy, 14(1), pp. 1–13. doi: 10.1186/S13287-023-03491-5/TAVOLE/6.