Trascrittoma spaziale BMKMANU S1000
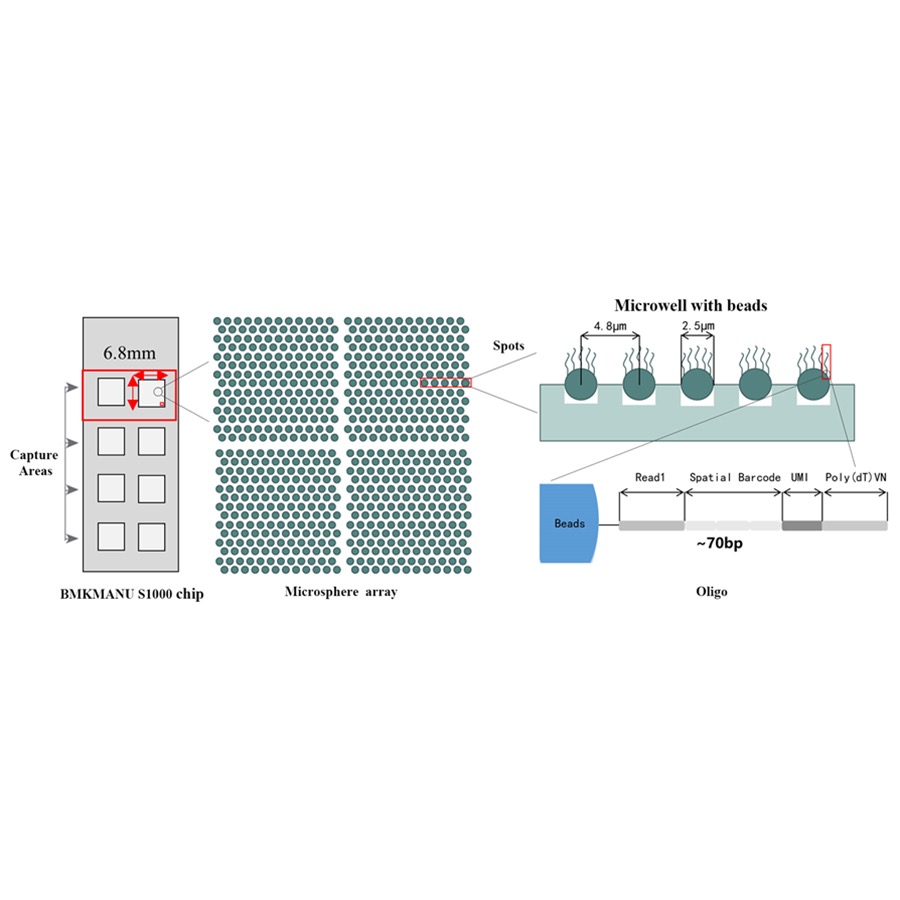
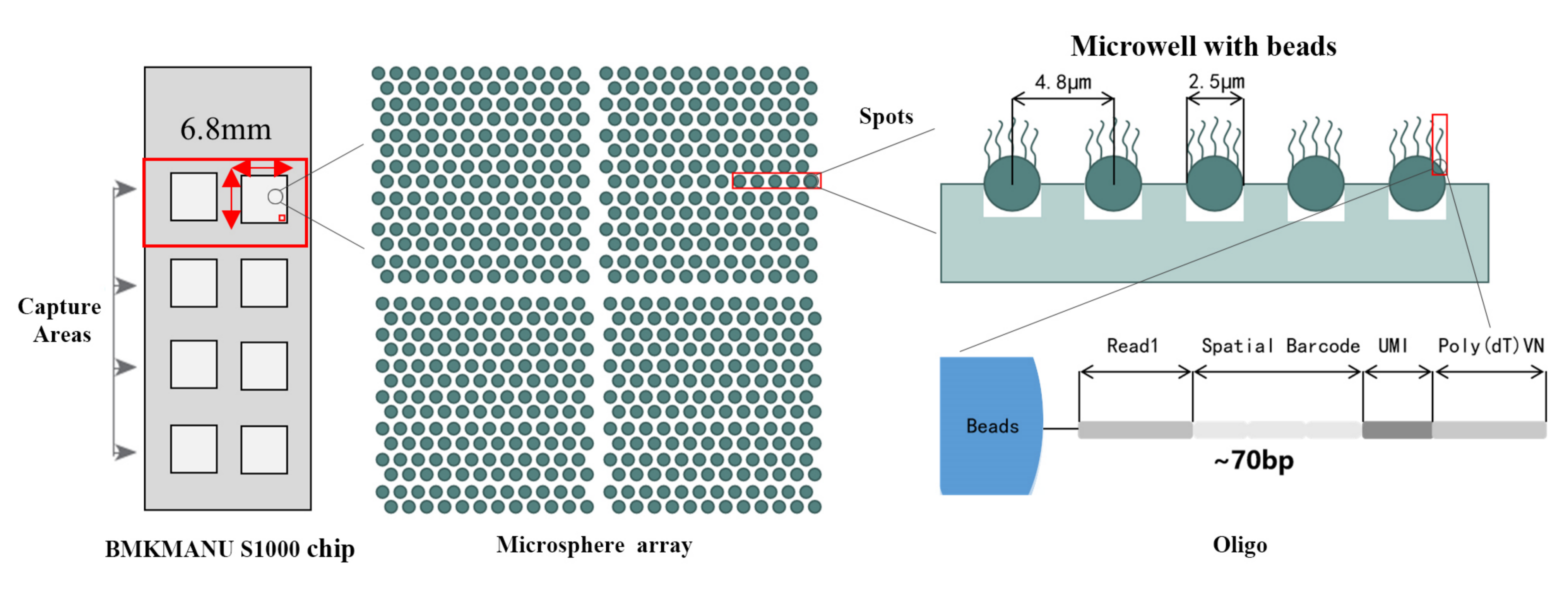
Schema tecnico del trascrittoma spaziale BMKMANU S1000
Caratteristiche
● Risoluzione: 5 µM
● Diametro dello spot: 2,5 µM
● Numero di spot: circa 2 milioni
● 3 possibili formati dell'area di acquisizione: 6,8 mm * 6,8 mm, 11 mm * 11 mm o 15 mm * 20 mm
● Ciascuna microsfera con codice a barre è caricata con primer composti da 4 sezioni:
coda poli(dT) per il priming dell'mRNA e la sintesi del cDNA
Identificatore molecolare univoco (UMI) per correggere il bias di amplificazione
Codice a barre spaziale
Sequenza legante del primer di sequenziamento di lettura parziale 1
● Colorazione H&E e fluorescente delle sezioni
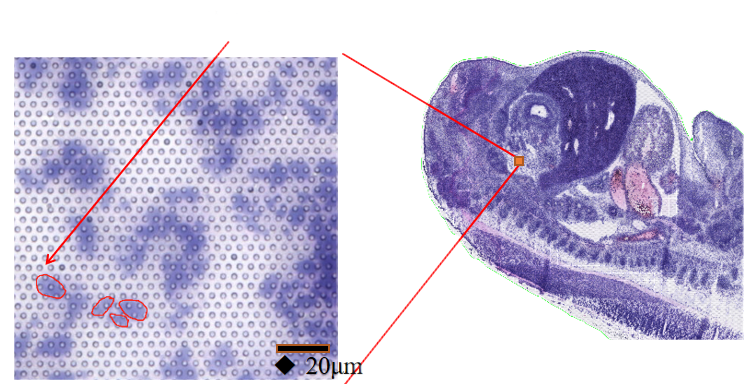
● Possibilità di utilizzotecnologia di segmentazione cellulare: integrazione della colorazione H&E, colorazione fluorescente e sequenziamento dell'RNA per determinare i confini di ciascuna cellula e assegnare correttamente l'espressione genica a ciascuna cellula.
Vantaggi di BMKMANU S1000
●Risoluzione subcellulare: Ciascuna area di cattura conteneva >2 milioni di spot spaziali con codice a barre con un diametro di 2,5 µm e una spaziatura di 5 µm tra i centri degli spot, consentendo l'analisi del trascrittoma spaziale con risoluzione subcellulare (5 µm).
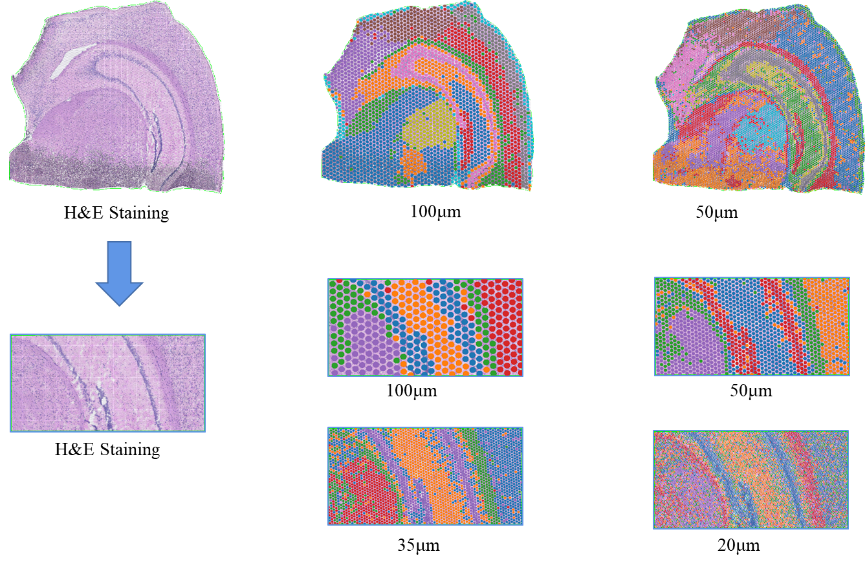
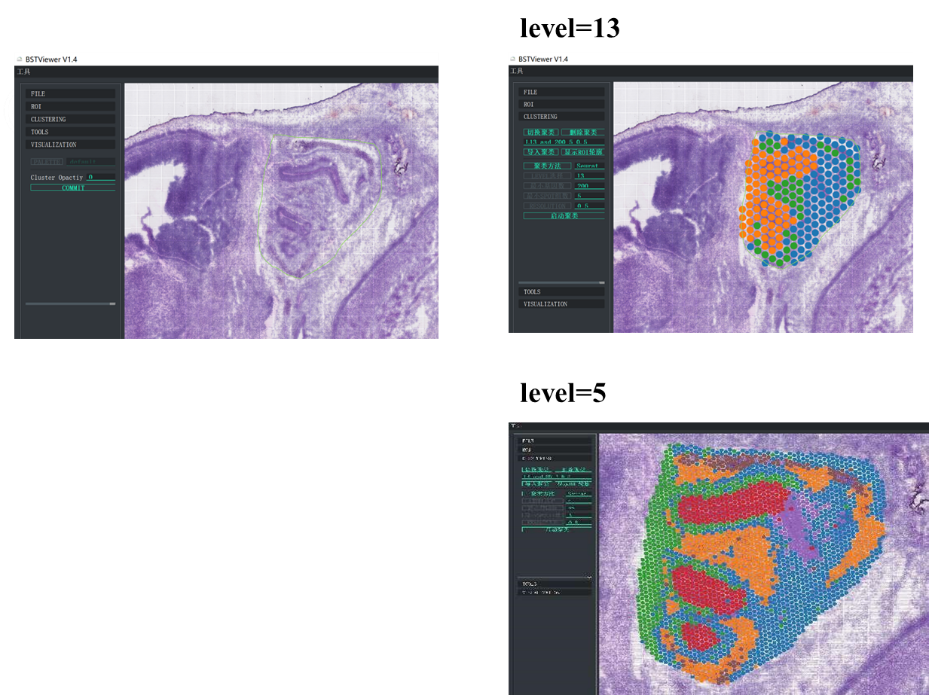
●Analisi della risoluzione multilivello:Analisi multilivello flessibile che va da 100 μm a 5 μm per risolvere diverse caratteristiche dei tessuti con una risoluzione ottimale.
●Possibilità di utilizzare la tecnologia di segmentazione cellulare “Tre in una diapositiva”:Combinando la colorazione a fluorescenza, la colorazione H&E e il sequenziamento dell'RNA su un singolo vetrino, il nostro algoritmo di analisi "tre in uno" consente l'identificazione dei confini cellulari per la successiva trascrittomica basata sulle cellule.
●Compatibile con più piattaforme di sequenziamento: Disponibili sia il sequenziamento NGS che quello a lettura lunga.
●Design flessibile di 1-8 aree di acquisizione attive: La dimensione dell'area di cattura è flessibile, essendo possibile utilizzare 3 formati (6,8 mm * 6,8 mm., 11 mm * 11 mm e 15 mm * 20 mm)
●Servizio unico: Integra tutte le fasi basate sull'esperienza e sulle competenze, tra cui criosezionamento, colorazione, ottimizzazione dei tessuti, codifica a barre spaziale, preparazione delle librerie, sequenziamento e bioinformatica.
●Bioinformatica completa e visualizzazione intuitiva dei risultati:il pacchetto include 29 analisi e oltre 100 dati di alta qualità, combinati con l'uso di software sviluppato internamente per visualizzare e personalizzare la suddivisione cellulare e il clustering spot.
●Analisi e visualizzazione personalizzata dei dati: disponibile per diverse richieste di ricerca
●Team tecnico altamente qualificato: con esperienza in oltre 250 tipi di tessuti e oltre 100 specie tra cui esseri umani, topi, mammiferi, pesci e piante.
●Aggiornamenti in tempo reale sull'intero progetto: con il pieno controllo del progresso sperimentale.
●Analisi congiunta opzionale con sequenziamento dell'mRNA di una singola cellula
Specifiche del servizio
|
Campione Requisiti
| Biblioteca |
Strategia di sequenziamento
| Dati consigliati | Controllo di qualità |
| Campioni criogenici incorporati nell'OCT, 3 blocchi per campione | Libreria di cDNA S1000 | Illumina PE150 (altre piattaforme disponibili) | 100.000 letture PE per 100 uM (60-150 GB) | RIN>7 |
Per ulteriori dettagli sulla guida alla preparazione dei campioni e sul flusso di lavoro del servizio, non esitate a parlare con aEsperto di BMKGENE
Flusso di lavoro del servizio
Nella fase di preparazione del campione, viene eseguita una prova iniziale di estrazione dell'RNA in massa per garantire che sia possibile ottenere un RNA di alta qualità. Nella fase di ottimizzazione del tessuto le sezioni vengono colorate e visualizzate e le condizioni di permeabilizzazione per il rilascio di mRNA dal tessuto vengono ottimizzate. Il protocollo ottimizzato viene quindi applicato durante la costruzione della libreria, seguito dal sequenziamento e dall'analisi dei dati.
Il flusso di lavoro completo del servizio prevede aggiornamenti in tempo reale e conferme del cliente per mantenere un ciclo di feedback reattivo, garantendo un'esecuzione regolare del progetto.
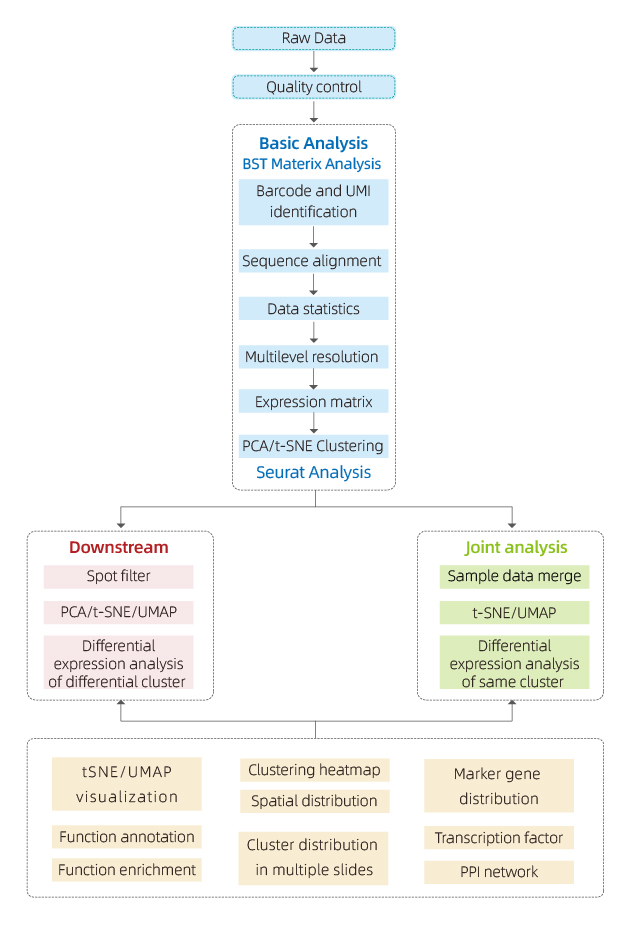
I dati generati da BMKMANU S1000 vengono analizzati utilizzando il software “BSTMatrix”, progettato in modo indipendente da BMKGENE, generando una matrice di espressione genica. Da lì, viene generato un report standard che include il controllo della qualità dei dati, l'analisi del campione interno e l'analisi intergruppo.
● Controllo della qualità dei dati:
Output dei dati e distribuzione del punteggio di qualità
Rilevazione genica per spot
Copertura dei tessuti
● Analisi del campione interno:
Ricchezza genetica
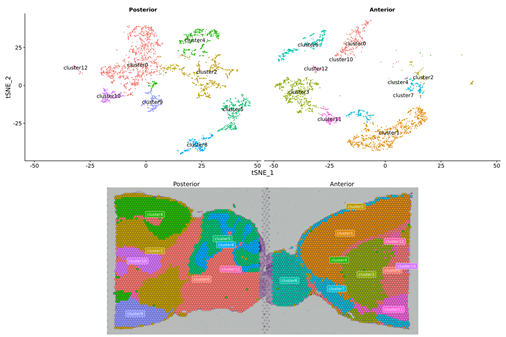
Clustering spot, inclusa l'analisi delle dimensioni ridotte
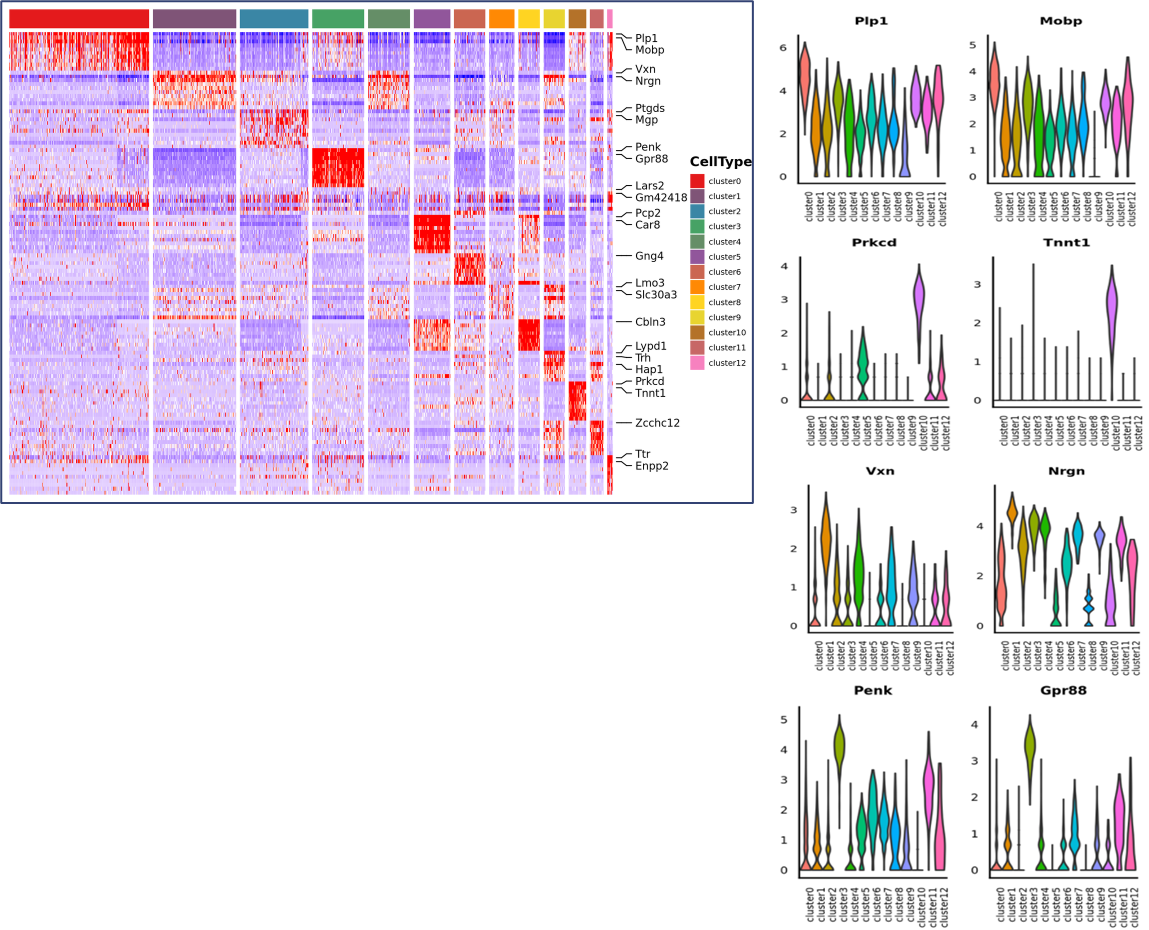
Analisi dell'espressione differenziale tra cluster: identificazione di geni marcatori
Annotazione funzionale e arricchimento di geni marcatori
● Analisi intergruppo:
Ricombinazione degli spot di entrambi i campioni (ad es. malato e controllo) e ri-cluster
Identificazione dei geni marcatori per ciascun cluster
Annotazione funzionale e arricchimento di geni marcatori
Espressione differenziale dello stesso cluster tra gruppi
Inoltre, “BSTViewer” sviluppato da BMKGENE è uno strumento intuitivo che consente all'utente di visualizzare l'espressione genica e individuare il clustering a diverse risoluzioni.
BMKGene ha sviluppato un software per una visualizzazione intuitiva
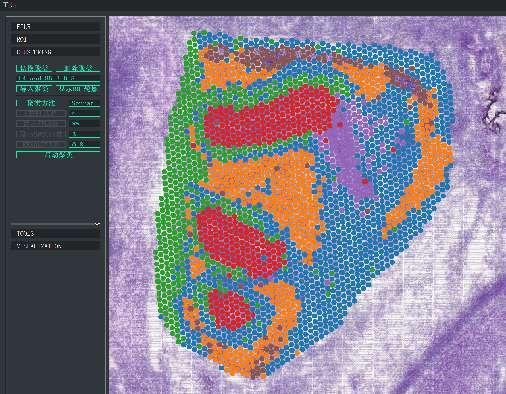
Clustering spot BSTViewer con risoluzione multilivello
BSTCellViewer: suddivisione cellulare automatica e manuale
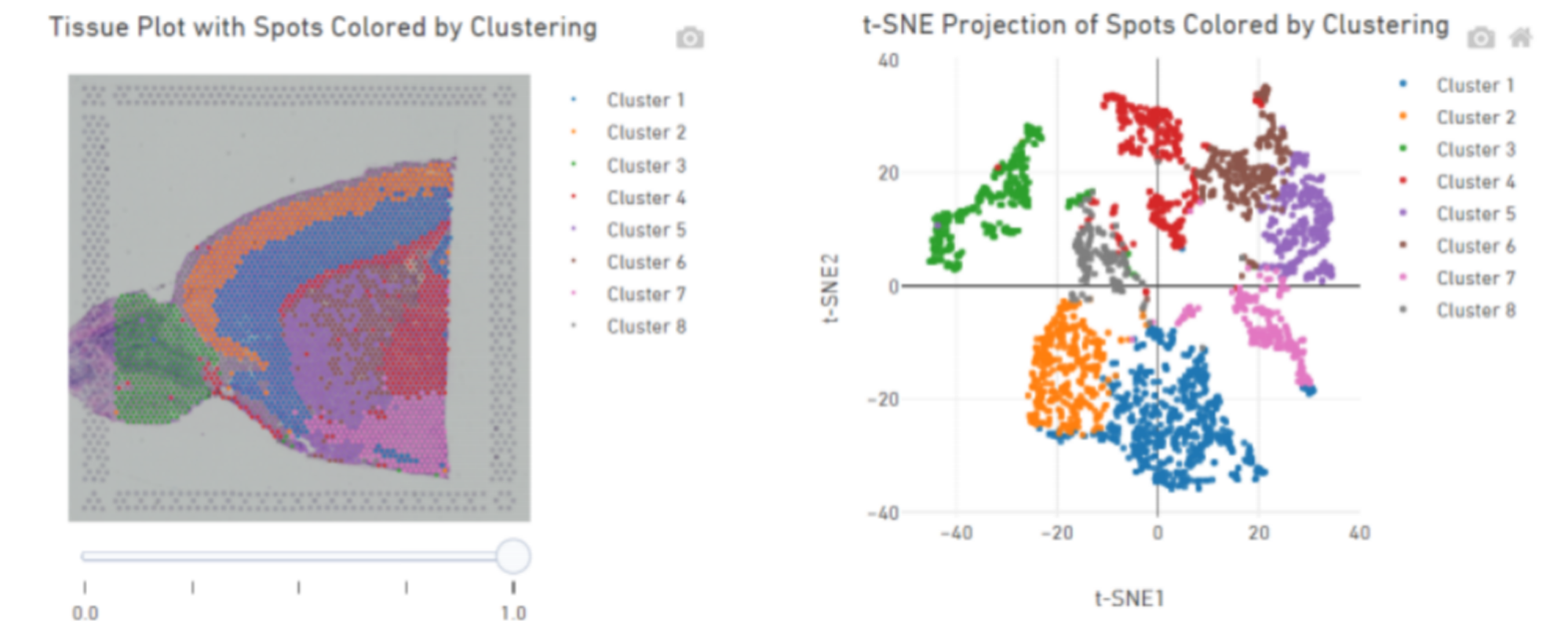
Analisi del campione interno
Raggruppamento di punti:
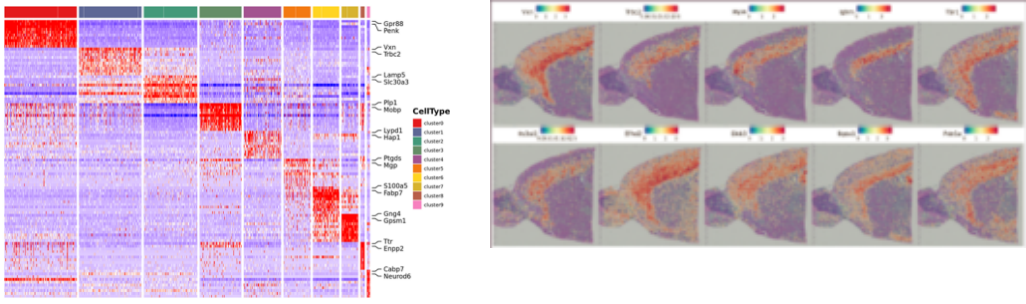
Identificazione dei geni marcatori e distribuzione spaziale:
Analisi intergruppo
Combinazione di dati da entrambi i gruppi e ri-cluster:
Geni marcatori di nuovi cluster:
Esplora i progressi facilitati dai servizi di trascrittomica spaziale di BMKGene con la tecnologia BMKManu S1000 in questa pubblicazione in primo piano:
Canzone, X. et al. (2023) "La trascrittomica spaziale rivela cellule di clorenchima indotte dalla luce coinvolte nella promozione della rigenerazione dei germogli nel callo del pomodoro",Atti dell'Accademia Nazionale delle Scienze degli Stati Uniti d'America, 120(38), pag. e2310163120. doi: 10.1073/pnas.2310163120
Tu, Y. et al. (2023) "Confronto sistematico di metodi di trascrittomica spaziale basati sul sequenziamento",bioRxiv, P. 2023.12.03.569744. doi: 10.1101/2023.12.03.569744.